Similar presentations:
Генетическая инженерия
1. Генетическая инженерия
2. АЛГОРИТМ РАБОТЫ (анализ последовательности)
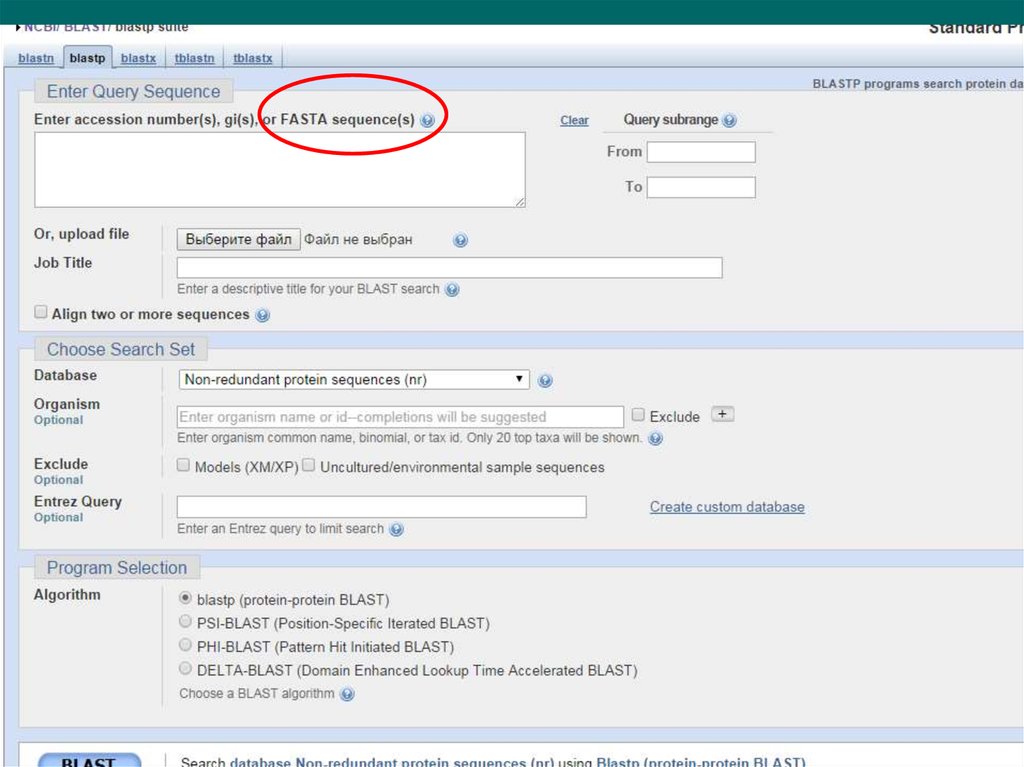
• Провести BLAST целевой последовательности• Определить гомологов
• Определить потенциальную функцию белка
• Дать статистическую оценку найденным гомологам
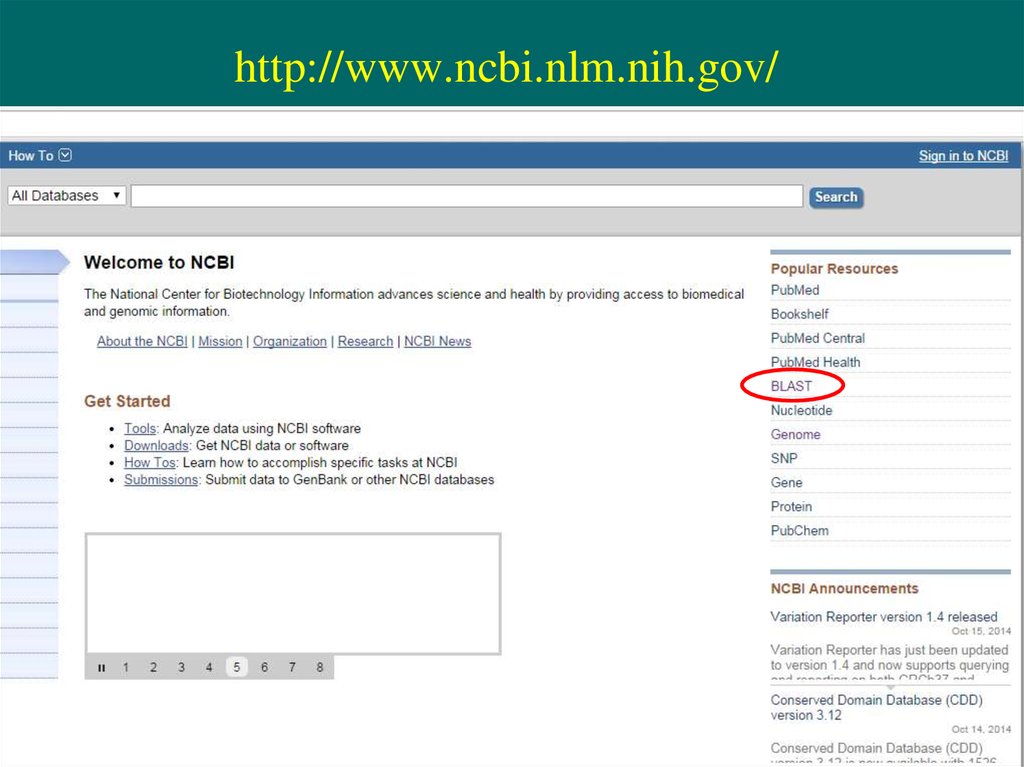
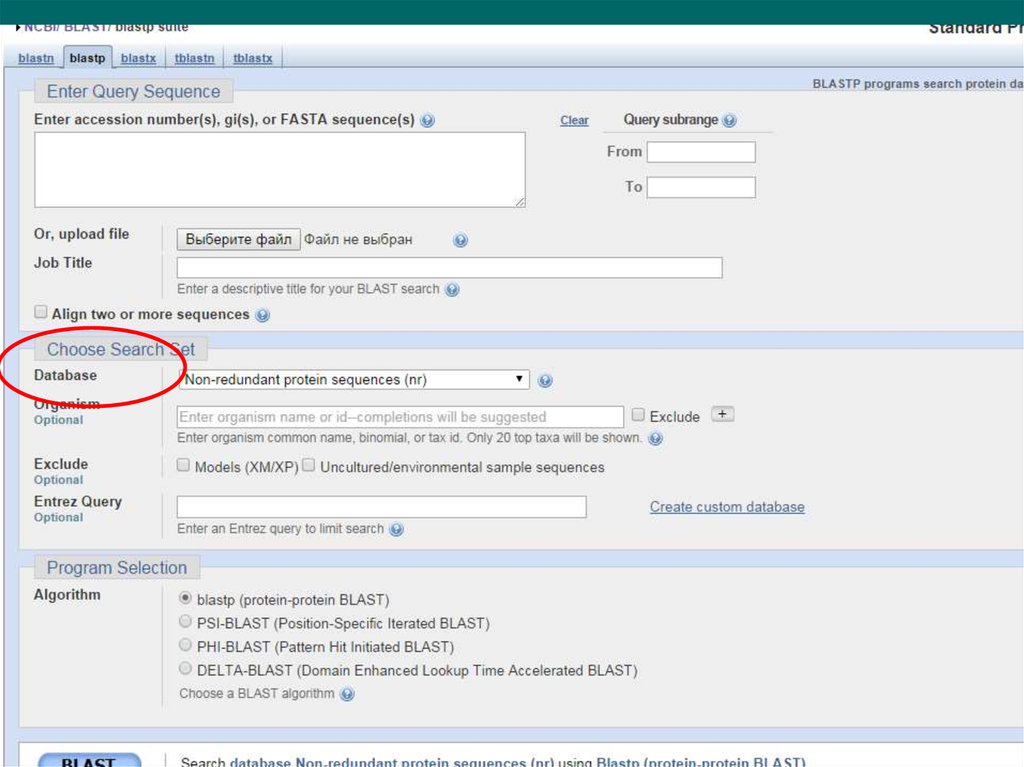
3. http://www.ncbi.nlm.nih.gov/
4.
5.
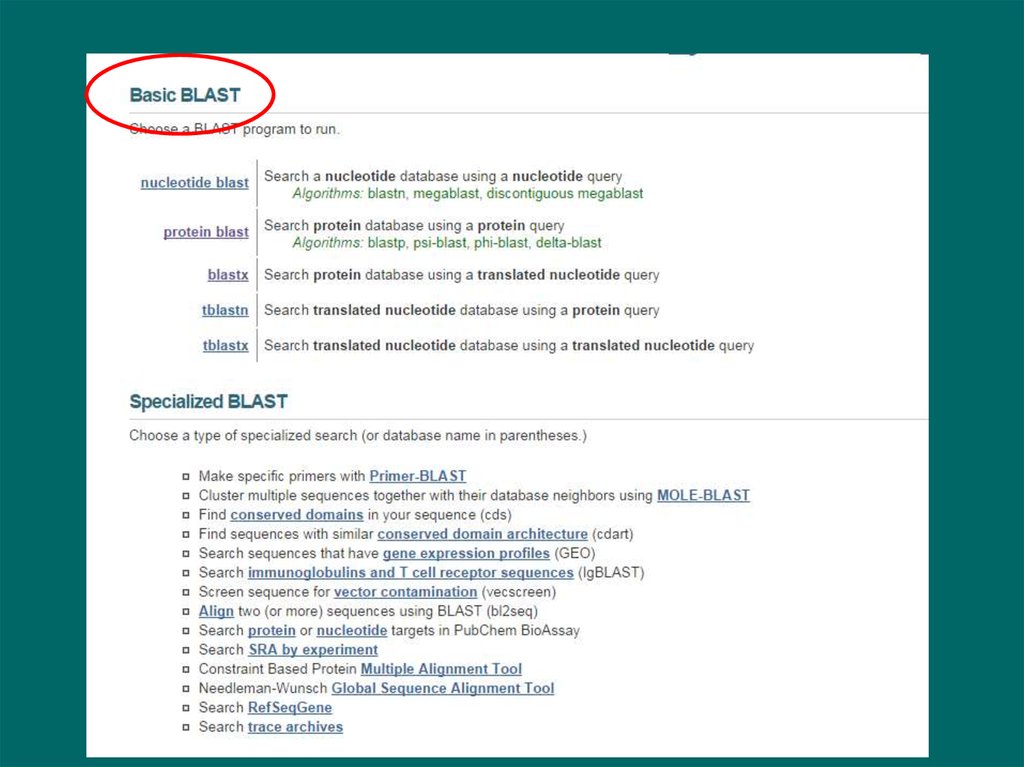
Step 2: Choose the BLAST program6.
7. Форматы файлов, используемых в биоинформатике
FASTA>roa1_drome Rea guano receptor type III >> 0.1
MVNSNQNQNGNSNGHDDDFPQDSITEPEHMRKLFIGGLDYRTTDENLKAHEKWGNIVDV
VVMKDPRTKRSRGFGFITYSHSSMIDEAQKSRPHKIDGRVEPKRAVPRQDIDSPNAGATVK
KLFVGALKDDHDEQSIRDYFQHFGNIVDNIVIDKETGKKRGFAFVEFDDYDPVDKVVLQK
QHQLNGKMVDVKKALPKNDQQGGGGGRGGPGGRAGGNRGNMGGGNYGNQNGGGNW
NNGGNNWGNNRGNDNWGNNSFGGGGGGGGGYGGGNNSWGNNNPWDNGNGGGNFGG
GGNNWNGGNDFGGYQQNYGGGPQRGGGNFNNNRMQPYQGGGGFKAGGGNQGNYGN
NQGFNNGGNNRRY
>roa2_drome Rea guano ligand
MVNSNQNQNGNSNGHDDDFPQDSITEPEHMRKLFIGGLDYRTTDENLKAHEKWGNIVDV
VVMKDPTSTSTSTSTSTSTSTSTMIDEAQKSRPHKIDGRVEPKRAVPRQDIDSPNAGATVK
KLFVGALKDDHDEQSIRDYFQHLLLLLLLDLLLLDLLLLDLLLFVEFDDYDPVDKVVLQK
QHQLNGKMVDVKKALPKNDQQGGGGGRGGPGGRAGGNRGNMGGGNYGNQNGGGNW
NNGGNNWGNNRGNDNWGNNSFGGGGGGGGGYGGGNNSWGNNNPWDNGNGGGNFGG
GGNNWNGGNDFGGYQQNYGGGPQRGGGNFNNNRMQPYQGGGGFKAGGGNQGNYGN
NQGFNNGGNNRRY
8.
9.
Step 3: choose the databasenr = non-redundant (most general database)
dbest = database of expressed sequence tags
dbsts = database of sequence tag sites
gss = genomic survey sequences
protein databases
nucleotide databases
10.
Step 4a: Select optional search parametersorganism
Entrez!
algorithm
page 107
11.
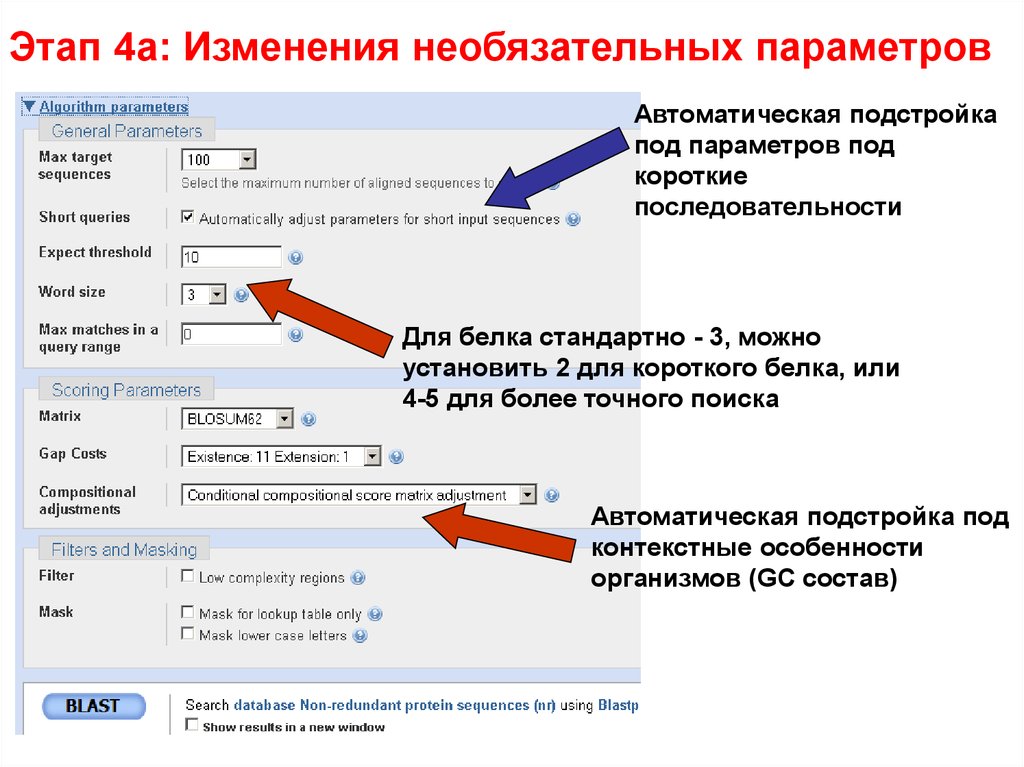
Этап 4a: Изменения необязательных параметровМаксимальное кол-во
выравниваний в выдаче
Вероятность нахождения
последовательностей с таким же
скором по случайным причинам
Scoring matrix
12.
Этап 4a: Изменения необязательных параметровАвтоматическая подстройка
под параметров под
короткие
последовательности
Для белка стандартно - 3, можно
установить 2 для короткого белка, или
4-5 для более точного поиска
Автоматическая подстройка под
контекстные особенности
организмов (GC состав)
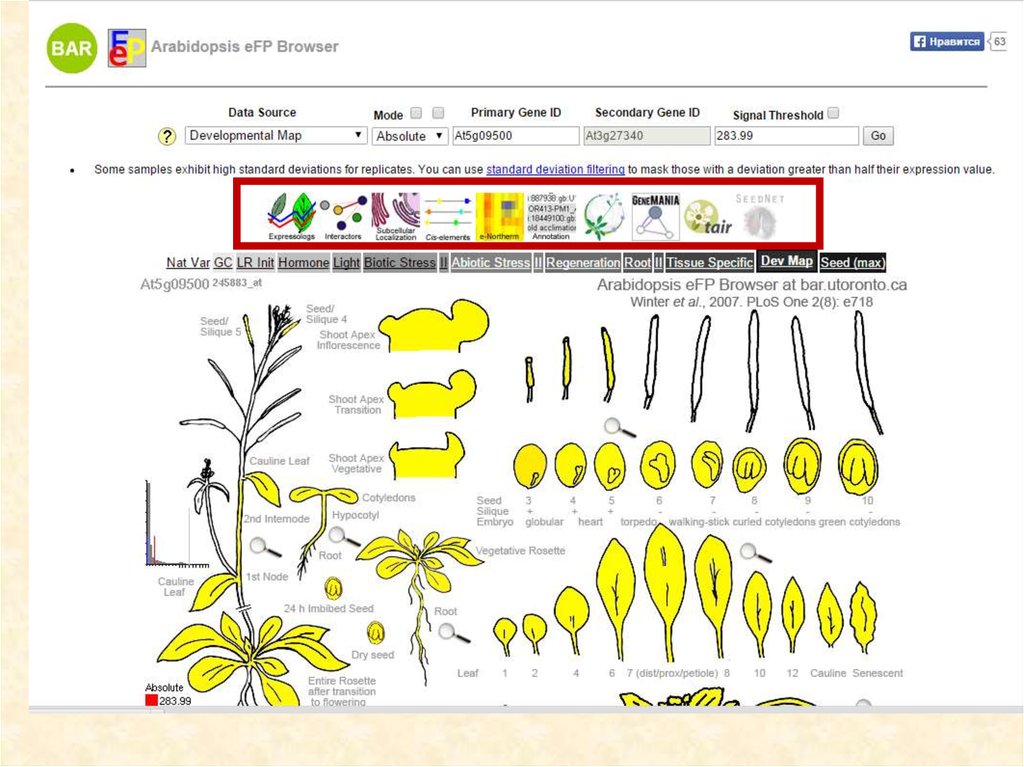
13. АЛГОРИТМ РАБОТЫ (анализ последовательности)
• Поиск в базах данных всей известной информации:• http://bar.utoronto.ca/efp/cgi-bin/efpWeb.cgi
14.
15.
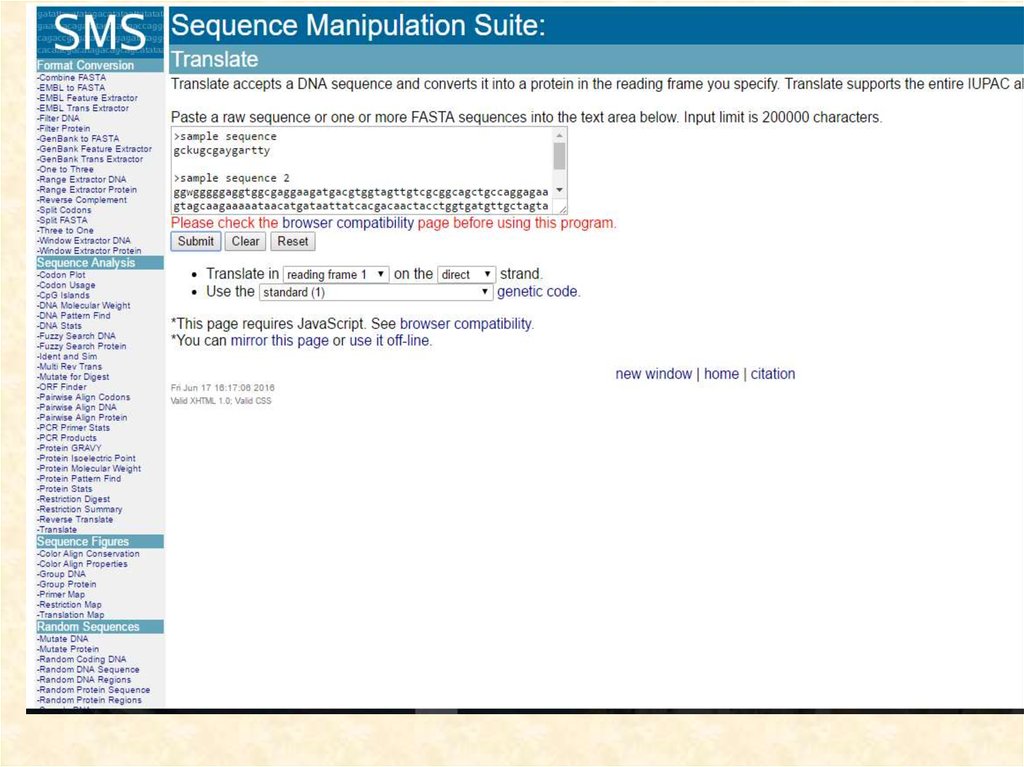
16. Дополнительный анализ последовательности
• http://www.bioinformatics.org/sms2/17.
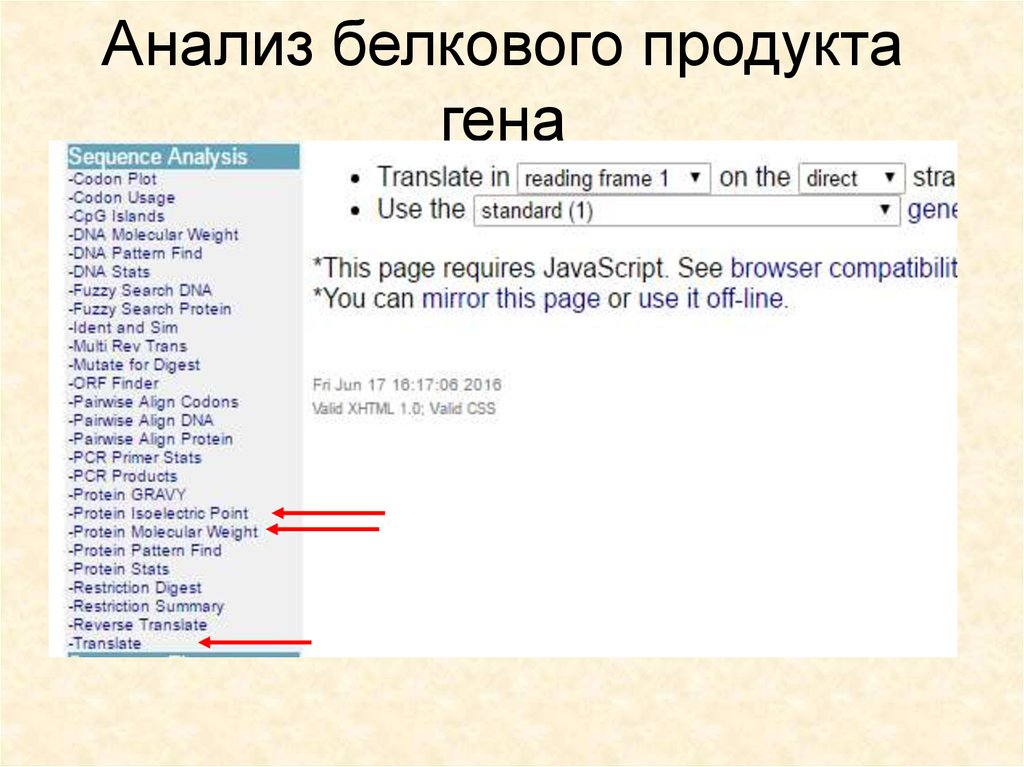
18. Анализ белкового продукта гена
19. АЛГОРИТМ РАБОТЫ
НАЙТИ ПОСЛЕДОВАТЕЛЬНОСТИ САЙТОВ В
ПОЛИЛИНКЕРЕ ВЕКТОРА.
ПРОВЕСТИ ПОИСК ЭТИХ САЙТОВ В
ПОСЛЕДОВАТЕЛЬНОСТИ ЦЕЛЕВОГО ГЕНА.
ВЫБРАТЬ САЙТЫ В ПОЛИЛИНКЕРЕ ВЕКТОРА ДЛЯ
КЛОНИРОВАНИЯ ЦЕЛЕВОГО ГЕНА(ВЫБИРАЮТ ТЕ,
КОТОРЫХ НЕТ В ПОСЛЕДОВАТЕЛЬНОСТИ ЦЕЛЕВОГО
ГЕНА). Учитывать направление промотора!!!
ПОДОБРАТЬ ПРАЙМЕРЫ К ПОСЛЕДОВАТЕЛЬНОСТИ
ЦЕЛЕВОГО ГЕНА (ПРЯМОЙ И ОБРАТНЫЙ).
ВВЕСТИ В СОСТАВ ПРАЙМЕРОВ ПОСЛЕДОВАТЕЛЬНОСТИ
ВЫБРАННЫХ САЙТОВ ДЛЯ КЛОНИРОВАНИЯ.
СОСТАВИТЬ СХЕМУ КЛОНИРОВАНИЯ ЦЕЛЕВОЙ
ПОСЛЕДОВАТЕЛЬНОСТИ В ВЕКТОР.
20. Вектора – что нужно учитывать
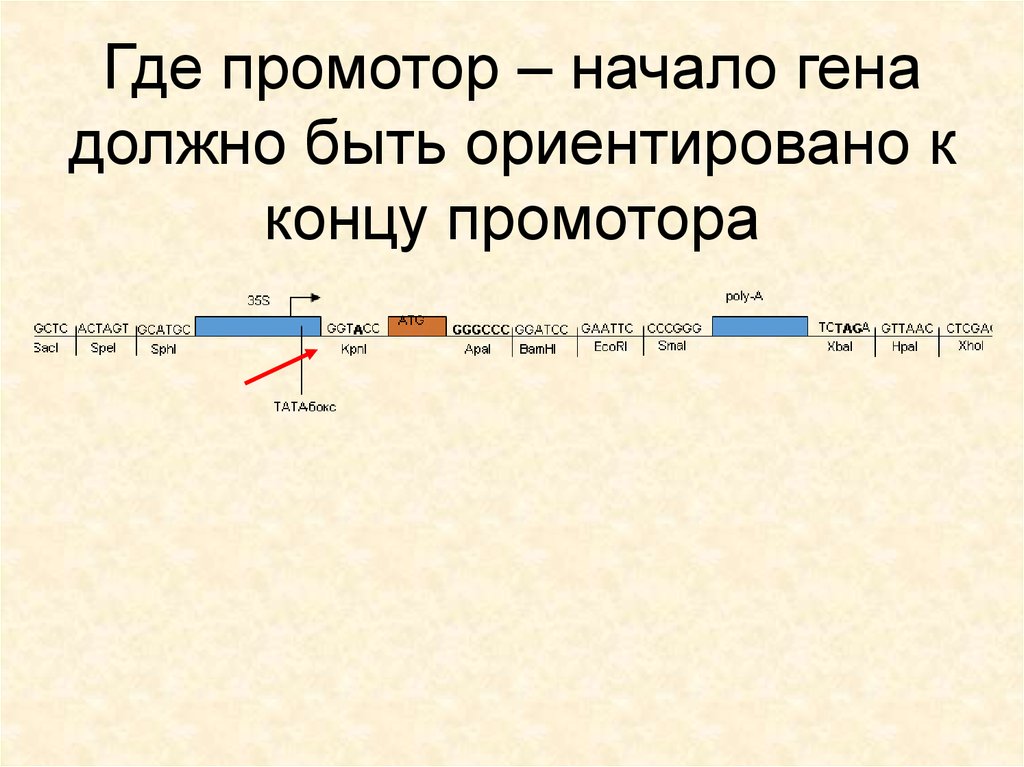
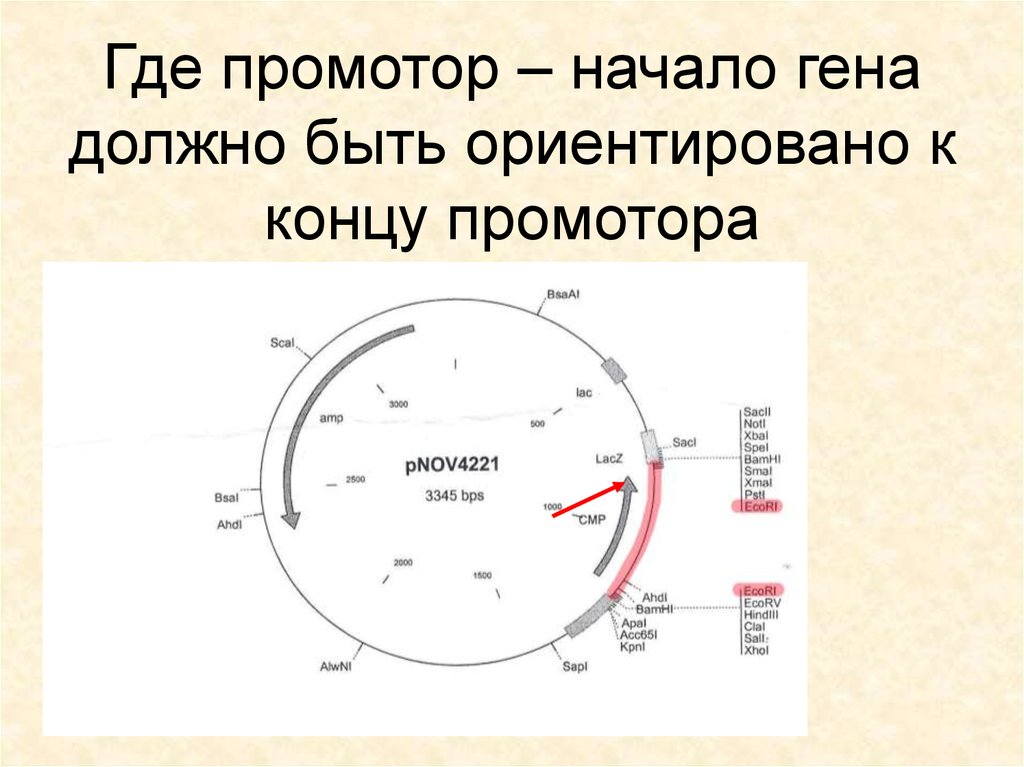
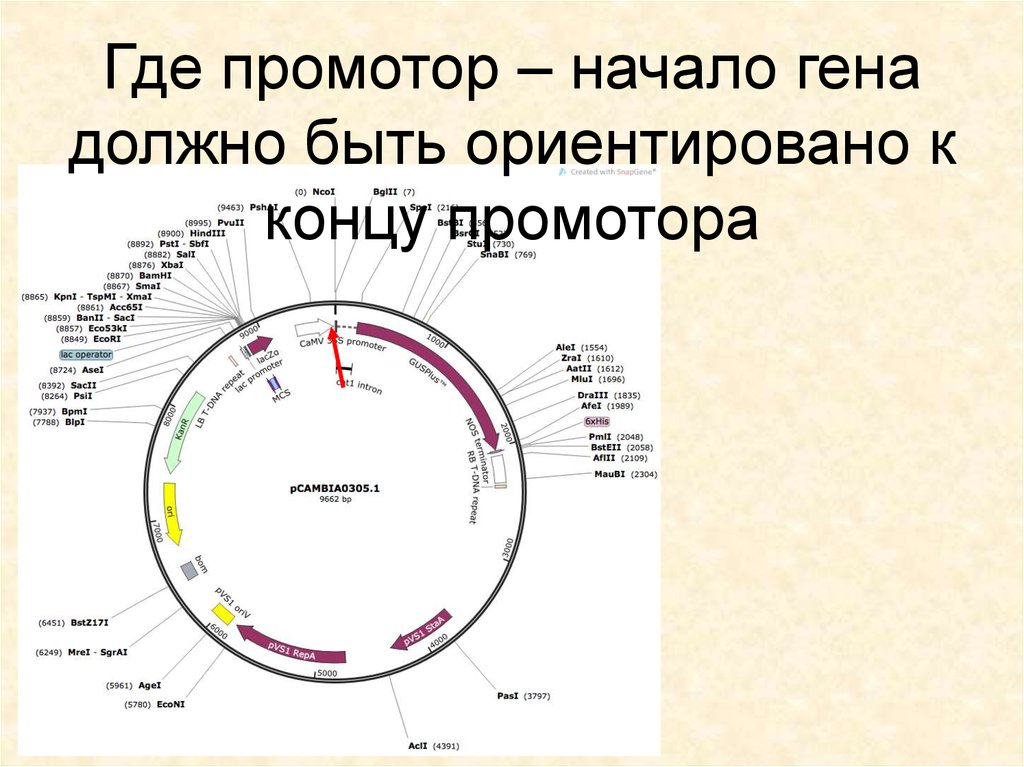
21. Где промотор – начало гена должно быть ориентировано к концу промотора
22. Где промотор – начало гена должно быть ориентировано к концу промотора
23. Где промотор – начало гена должно быть ориентировано к концу промотора
24. Где промотор – начало гена должно быть ориентировано к концу промотора
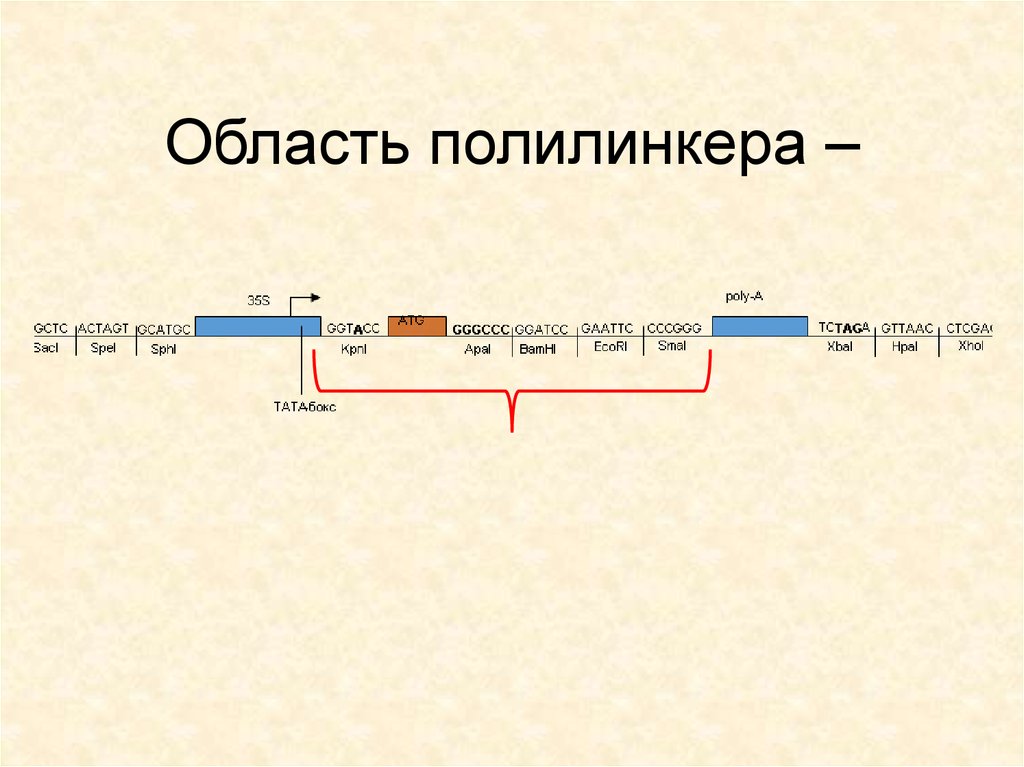
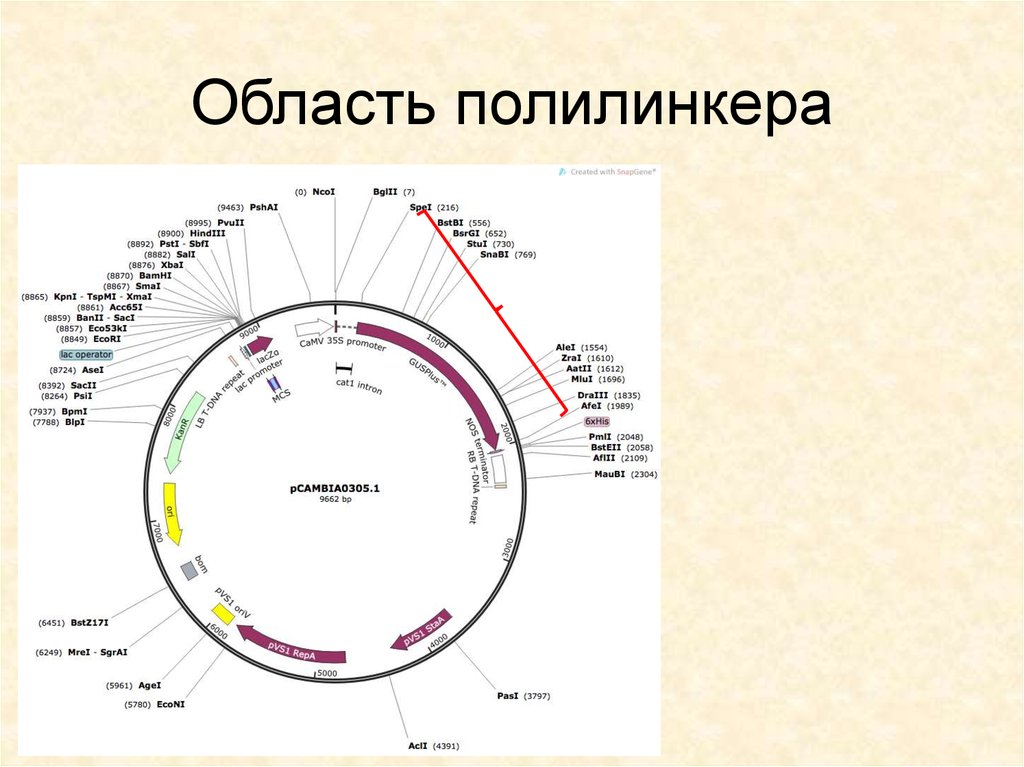
25. Область полилинкера –
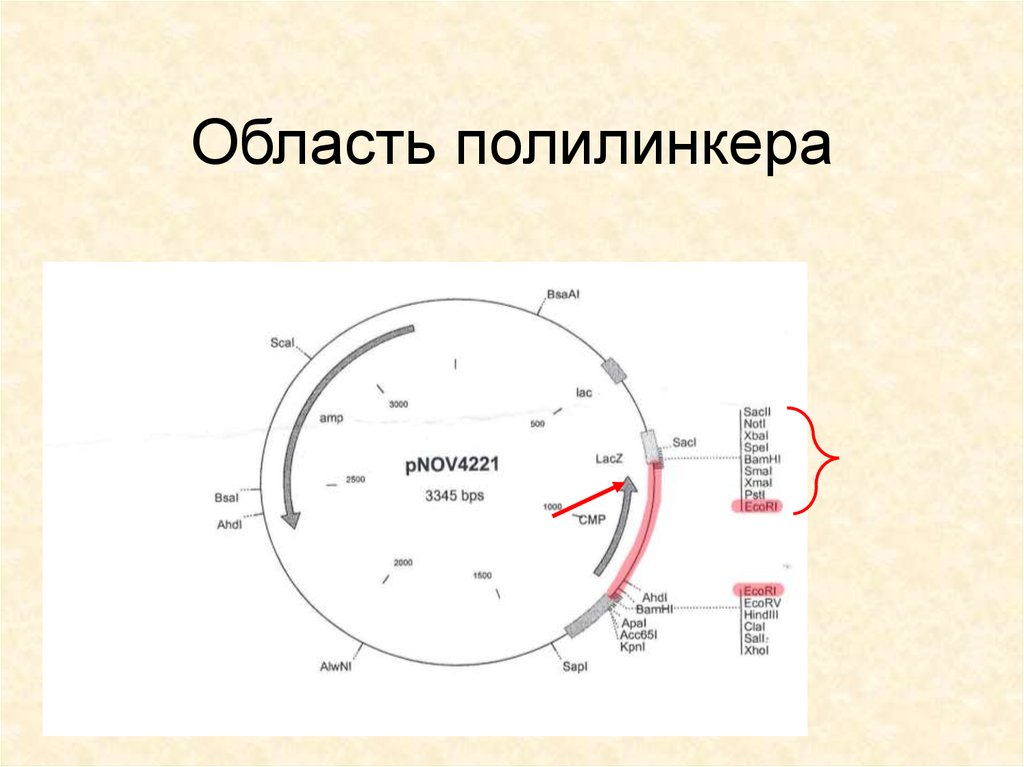
26. Область полилинкера
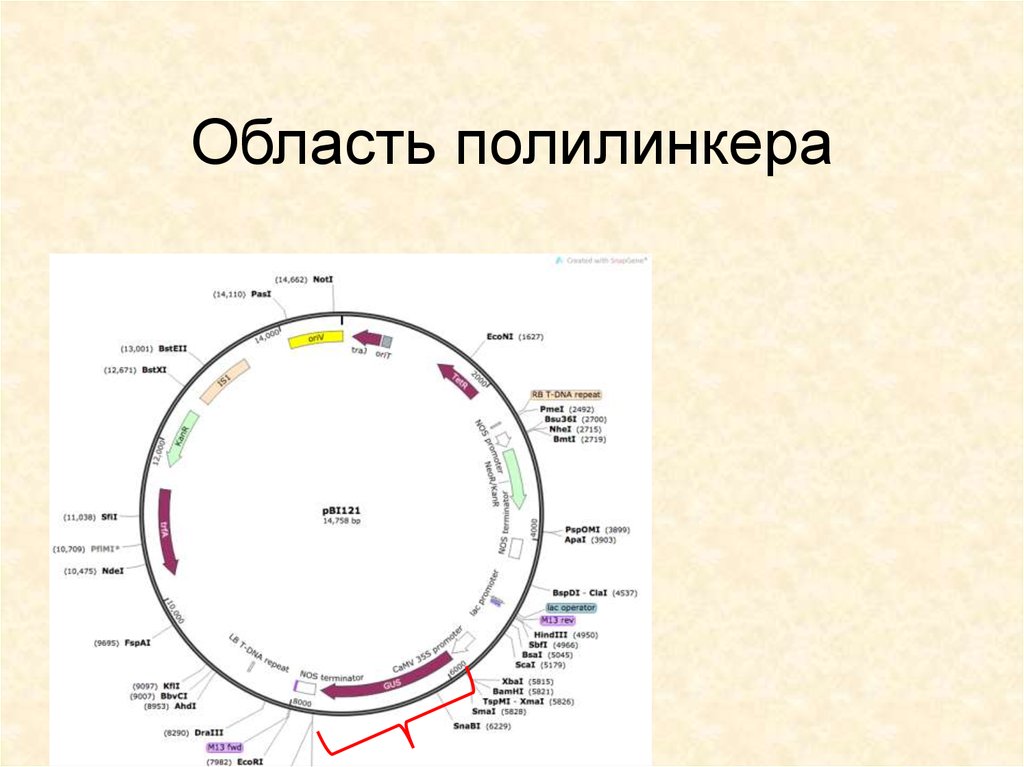
27. Область полилинкера
28. Область полилинкера
29. 1 этап
1.НАЙТИ ПОСЛЕДОВАТЕЛЬНОСТИ САЙТОВ В
ПОЛИЛИНКЕРЕ ВЕКТОРА. Bioinformatics.org/SMS/
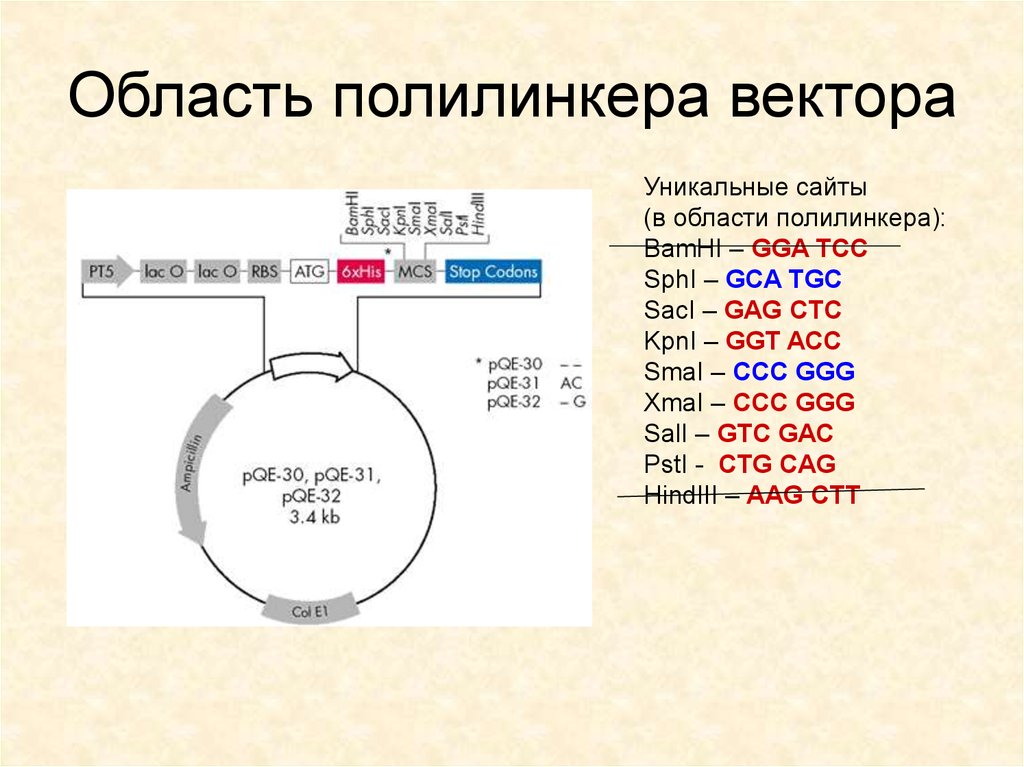
30. Область полилинкера вектора
Уникальные сайты(в области полилинкера):
BamHI – GGA TCC
SphI – GCA TGC
SacI – GAG CTC
KpnI – GGT ACC
SmaI – CCC GGG
XmaI – CCC GGG
SalI – GTC GAC
PstI - CTG CAG
HindIII – AAG CTT
31. АЛГОРИТМ РАБОТЫ
• ПРОВЕСТИ ПОИСК ЭТИХСАЙТОВ В
ПОСЛЕДОВАТЕЛЬНОСТИ
ЦЕЛЕВОГО ГЕНА.
Bioinformatics.org/SMS/
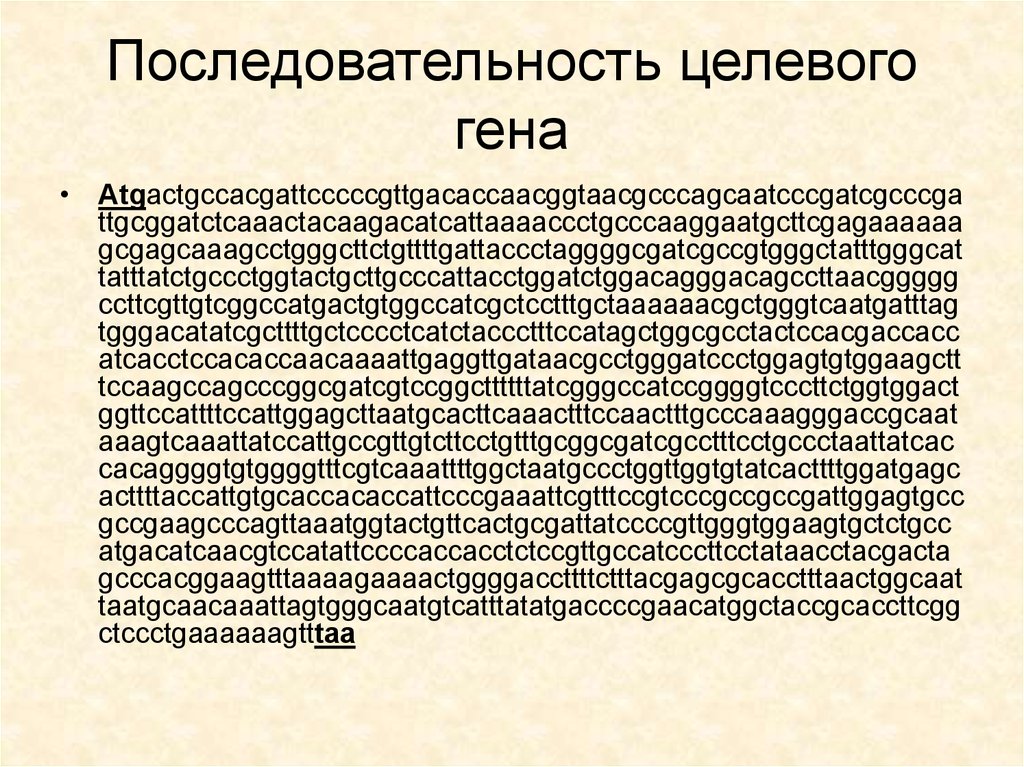
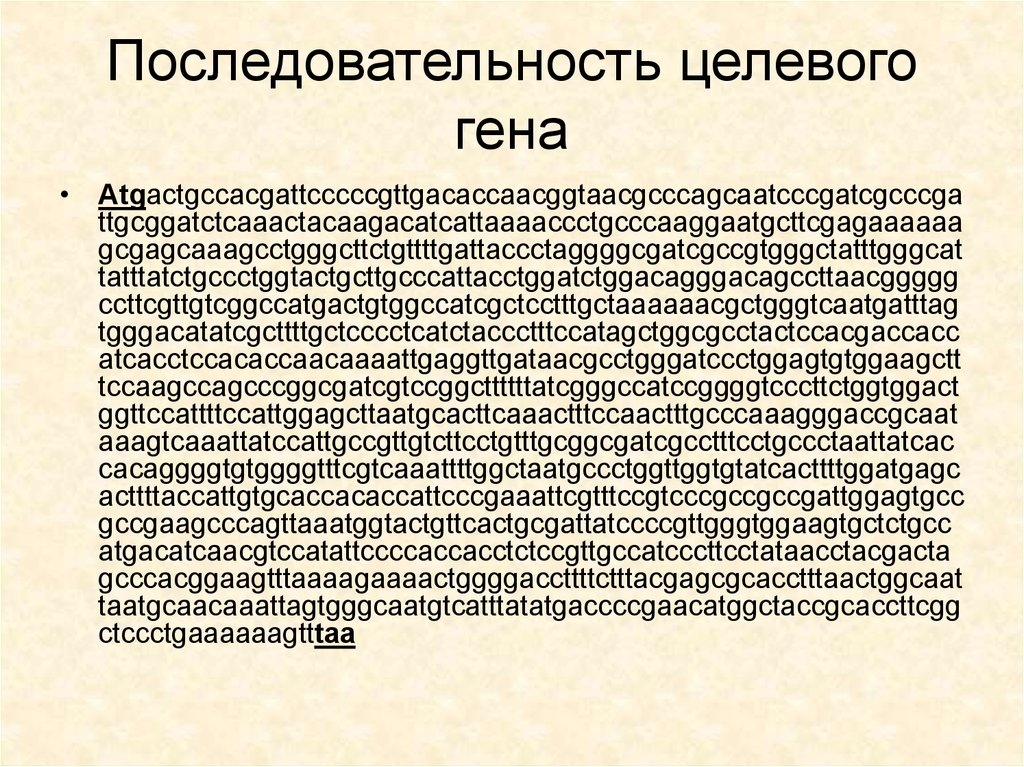
32. Последовательность целевого гена
• Atgactgccacgattcccccgttgacaccaacggtaacgcccagcaatcccgatcgcccgattgcggatctcaaactacaagacatcattaaaaccctgcccaaggaatgcttcgagaaaaaa
gcgagcaaagcctgggcttctgttttgattaccctaggggcgatcgccgtgggctatttgggcat
tatttatctgccctggtactgcttgcccattacctggatctggacagggacagccttaacggggg
ccttcgttgtcggccatgactgtggccatcgctcctttgctaaaaaacgctgggtcaatgatttag
tgggacatatcgcttttgctcccctcatctaccctttccatagctggcgcctactccacgaccacc
atcacctccacaccaacaaaattgaggttgataacgcctgggatccctggagtgtggaagctt
tccaagccagcccggcgatcgtccggcttttttatcgggccatccggggtcccttctggtggact
ggttccattttccattggagcttaatgcacttcaaactttccaactttgcccaaagggaccgcaat
aaagtcaaattatccattgccgttgtcttcctgtttgcggcgatcgcctttcctgccctaattatcac
cacaggggtgtggggtttcgtcaaattttggctaatgccctggttggtgtatcacttttggatgagc
acttttaccattgtgcaccacaccattcccgaaattcgtttccgtcccgccgccgattggagtgcc
gccgaagcccagttaaatggtactgttcactgcgattatccccgttgggtggaagtgctctgcc
atgacatcaacgtccatattccccaccacctctccgttgccatcccttcctataacctacgacta
gcccacggaagtttaaaagaaaactggggaccttttctttacgagcgcacctttaactggcaat
taatgcaacaaattagtgggcaatgtcatttatatgaccccgaacatggctaccgcaccttcgg
ctccctgaaaaaagtttaa
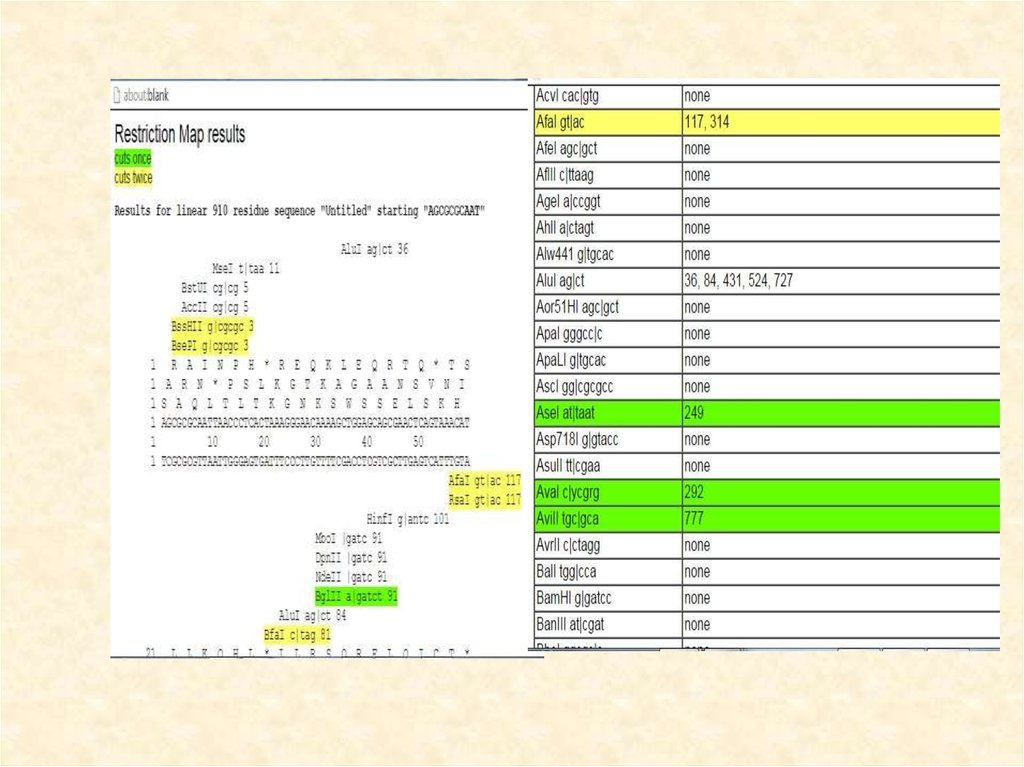
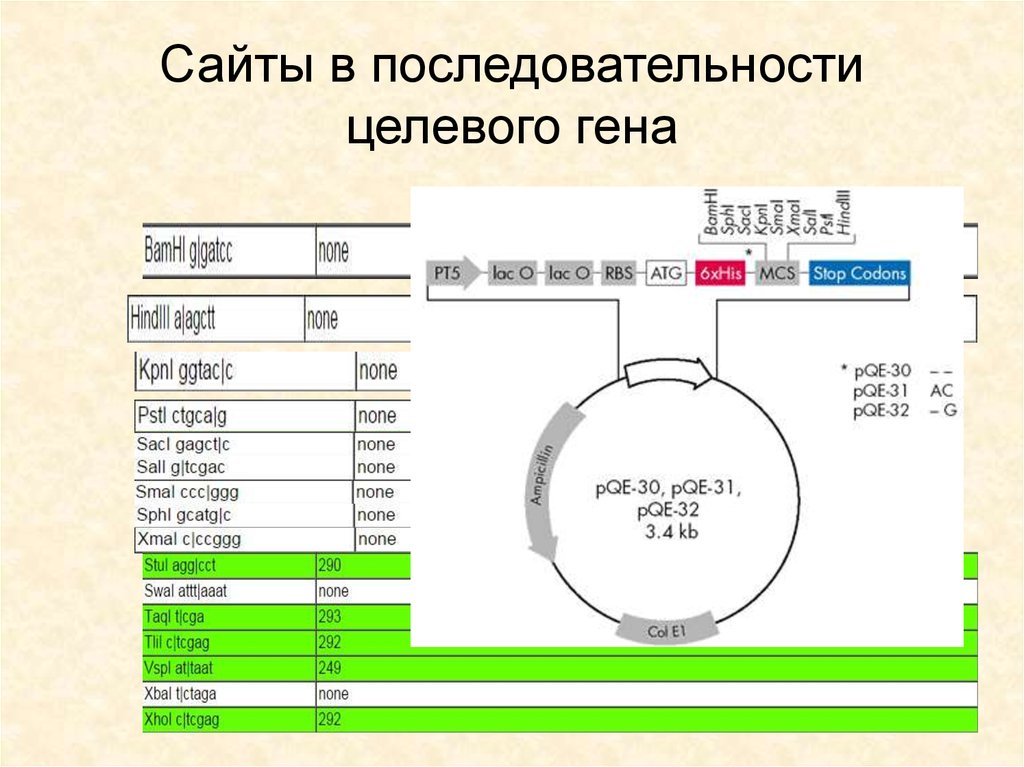
33. Сайты в последовательности целевого гена
34. Анализ сайтов рестрикции
35.
36. Сайты в последовательности целевого гена
37. АЛГОРИТМ РАБОТЫ
• ВЫБРАТЬ САЙТЫ ВПОЛИЛИНКЕРЕ ВЕКТОРА ДЛЯ
КЛОНИРОВАНИЯ ЦЕЛЕВОГО
ГЕНА(ВЫБИРАЮТ ТЕ, КОТОРЫХ
НЕТ В ПОСЛЕДОВАТЕЛЬНОСТИ
ЦЕЛЕВОГО ГЕНА).
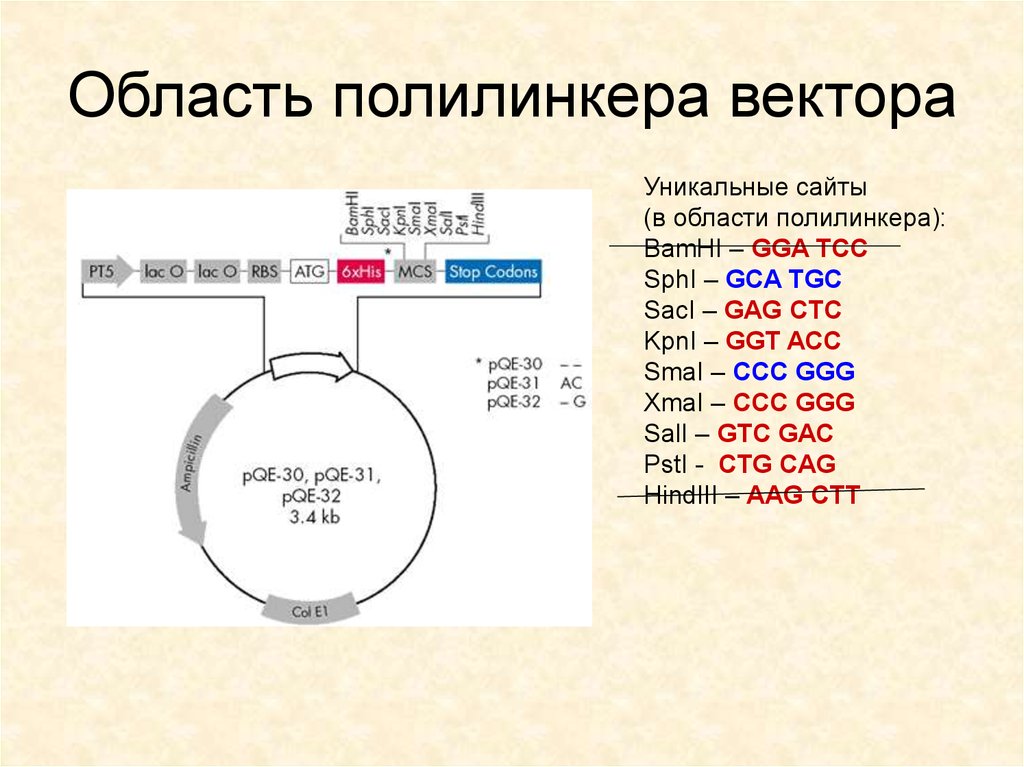
38. Область полилинкера вектора
Уникальные сайты(в области полилинкера):
BamHI – GGA TCC
SphI – GCA TGC
SacI – GAG CTC
KpnI – GGT ACC
SmaI – CCC GGG
XmaI – CCC GGG
SalI – GTC GAC
PstI - CTG CAG
HindIII – AAG CTT
39. АЛГОРИТМ РАБОТЫ
• ПОДОБРАТЬ ПРАЙМЕРЫ КПОСЛЕДОВАТЕЛЬНОСТИ
ЦЕЛЕВОГО ГЕНА (ПРЯМОЙ И
ОБРАТНЫЙ).
Bioinformatics.org/SMS/
40. Последовательность целевого гена
• Atgactgccacgattcccccgttgacaccaacggtaacgcccagcaatcccgatcgcccgattgcggatctcaaactacaagacatcattaaaaccctgcccaaggaatgcttcgagaaaaaa
gcgagcaaagcctgggcttctgttttgattaccctaggggcgatcgccgtgggctatttgggcat
tatttatctgccctggtactgcttgcccattacctggatctggacagggacagccttaacggggg
ccttcgttgtcggccatgactgtggccatcgctcctttgctaaaaaacgctgggtcaatgatttag
tgggacatatcgcttttgctcccctcatctaccctttccatagctggcgcctactccacgaccacc
atcacctccacaccaacaaaattgaggttgataacgcctgggatccctggagtgtggaagctt
tccaagccagcccggcgatcgtccggcttttttatcgggccatccggggtcccttctggtggact
ggttccattttccattggagcttaatgcacttcaaactttccaactttgcccaaagggaccgcaat
aaagtcaaattatccattgccgttgtcttcctgtttgcggcgatcgcctttcctgccctaattatcac
cacaggggtgtggggtttcgtcaaattttggctaatgccctggttggtgtatcacttttggatgagc
acttttaccattgtgcaccacaccattcccgaaattcgtttccgtcccgccgccgattggagtgcc
gccgaagcccagttaaatggtactgttcactgcgattatccccgttgggtggaagtgctctgcc
atgacatcaacgtccatattccccaccacctctccgttgccatcccttcctataacctacgacta
gcccacggaagtttaaaagaaaactggggaccttttctttacgagcgcacctttaactggcaat
taatgcaacaaattagtgggcaatgtcatttatatgaccccgaacatggctaccgcaccttcgg
ctccctgaaaaaagtttaa
41. Подбор праймеров
5’- Atgactgccacgattcc…………----……………………………………………………cgcaccttcggctccctgaaaaaagtttaa – 3’
Прямой праймер – такой же как
последовательность в 5’ области
целевого гена.
Почему?
42. Подбор праймеров
5’- Atg act gcc acg att cc…………----3’ Tac tga cgg tgc taa gg (комплементарная цепь)……………………………………………………
ctt cgg ctc cct gaa aaa agt tta a – 3’
gaa gcc gag gga ctt ttt tca aat t – 5’
Прямой праймер – «садится» на комплементарную цепь (3’ –
5’), следовательно, его последовательность совпадает. В
нашем случае: 5’- atg
act gcc acg att cc – 3’
43. Подбор праймеров
5’- Atgactgccacgattcc…………----……………………………………………………cgcaccttcggctccctgaaaaaagtttaa – 3’
Обратный праймер – пишется с учетом
правила комплементарности.
Почему?
44. Подбор праймеров
5’- Atg act gcc acg att cc…………----3’ Tac tga cgg tgc taa gg (комплементарная цепь)……………………………………………………
ctt cgg ctc cct gaa aaa agt tta a – 3’
gaa gcc gag gga ctt ttt tca aat t – 5’
Обратный праймер – «садится» на первую цепь (3’ – 5’),
следовательно, его последовательность должна быть ей
комплементарна. В нашем случае – 3’- gag gga ctt ttt tca
aat t – 5’
При заказе на синтез – праймер «переворачивают и пишут
следующим образом: 5’- t taa act ttt ttc agg gag - 3’
45. Прямое клонирование
• Сайты рестрикции можно ввести в 5’область праймера• Провести ПЦР
• Сделать гидролиз ПЦР-продукта и
вектора соответствующими
эндонуклеазами рестрикции
• Провести лигирование целевого
фрагмента и вектора
46. АЛГОРИТМ РАБОТЫ
• ВВЕСТИ В СОСТАВ ПРАЙМЕРОВПОСЛЕДОВАТЕЛЬНОСТИ
ВЫБРАННЫХ САЙТОВ ДЛЯ
КЛОНИРОВАНИЯ.
• Были выбраны сайты для SphI –
GCA TGC и SmaI – CCC GGG
47. Область полилинкера вектора
Уникальные сайты(в области полилинкера):
BamHI – GGA TCC
SphI – GCA TGC
SacI – GAG CTC
KpnI – GGT ACC
SmaI – CCC GGG
XmaI – CCC GGG
SalI – GTC GAC
PstI - CTG CAG
HindIII – AAG CTT
48. Введение сайтов рестрикции в праймеры
5’- Atg act gcc acg att cc…………----3’ Tac tga cgg tgc taa gg (комплементарная цепь)……………………………………………………
ctt cgg ctc cct gaa aaa agt tta a – 3’
gaa gcc gag gga ctt ttt tca aat t – 5’
Прямой праймер: 5’- atc gat gca tgc atg
– 3’
act gcc acg att cc
SphI – GCA TGC
Обратный праймер: 5’- aca ttc ccc ggg t
gag - 3’
SmaI – CCC GGG
taa act ttt ttc agg
49. Расчет температуры отжига праймеров
(A+T)*2+(C+G)*4 (для праймеров до 20-25 п.н.)Для праймеров большей длины более сложное:
22+1,46(2*(G+C) +(A+T))
Прямой праймер: 5’- atc gat gca tgc atg act gcc acg att cc
– 3’
(14)*2+(15)*4= 88 градусов
Обратный праймер: 5’- aca ttc ccc ggg t
gag - 3’
(16)*2+(14)*4= 88 градусов
taa act ttt ttc agg
50. Прямое клонирование
• Две пары праймеров:• Одна пара строго на
последовательность целевого гена
Прямой праймер: 5’- atg
att ccg att– 3’
(11)*2+(10)*4= 62 градуса
act gcc acg
Обратный праймер: 5’- t
agg gag ggc - 3’
(13)*2+(9)*4= 62 градуса
taa act ttt ttc
51. Прямое клонирование
• Провести ПЦР с первой парой• Подобрать вторую пару праймеров с
введенными сайтами рестрикции
(уменьшая число нуклеотидов,
соответствующих целевой
последовательности для оптимизации
температуры отжига)
52. Прямое клонирование
• Провести ПЦР, используя ПЦР-продуктпервой реакции в качестве матрицы
• Сделать гидролиз второго ПЦРпродукта и вектора соответствующими
эндонуклеазами рестрикции
• Провести лигирование целевого
фрагмента и вектора
53. Прямое клонирование
• Через Т-вектор (в случае использованияTaq-полимеразы)
• Тогда сайты рестрикции можно ввести в
5’-концевую часть праймера без
добавления нуклеотидов
54. Введение сайтов рестрикции в праймеры
5’- Atg act gcc acg att cc…………----3’ Tac tga cgg tgc taa gg (комплементарная цепь)……………………………………………………
ctt cgg ctc cct gaa aaa agt tta a – 3’
gaa gcc gag gga ctt ttt tca aat t – 5’
Прямой праймер: 5’- atc gat gca tgc atg
– 3’
act gcc acg att cc
SphI – GCA TGC
Обратный праймер: 5’- aca ttc ccc ggg t
gag - 3’
SmaI – CCC GGG
taa act ttt ttc agg
55. АЛГОРИТМ РАБОТЫ
• СОСТАВИТЬ СХЕМУКЛОНИРОВАНИЯ ЦЕЛЕВОЙ
ПОСЛЕДОВАТЕЛЬНОСТИ В
ВЕКТОР.
56.
Схема клонирования целевого гена в вектор.Вектор
1. Гидролизуем вектор SphI и SmaI.
2. Проводим разделение
в агарозном геле.
3. ДНК выделяем из геля и
4. Используем для лигирования с
последовательностью целевого гена.
Целевая последовательность
1. Проводим ПЦР с подобранными
праймерами.
2. Клонируем (ПЦР-продукт в Т-вектор).
3. Гидролизуем Т-вектор со вставкой
целевого гена SphI и SmaI.
4. Проводим разделение
в агарозном геле.
5. Фрагмент ДНК соответствующего
размера выделяем из геля и
6. Используем для лигирования в
экспрессионный вектор.



































 medicine
medicine biology
biology software
software