Similar presentations:
Компьютерные методы анализа нуклеотидных последовательностей
1. Компьютерные методы анализа нуклеотидных последовательностей
Биоинформационные подходы,позволяющие планировать эксперименты.
ПЦР. Дизайн праймеров для ПЦР и для
секвенирования ДНК.
2. ПЦР
Метод основан на многократном избирательномкопировании определённого участка ДНК in vitro. C
помощью ПЦР амплифицируются относительно
короткие участки ДНК. В обычном ПЦР-процессе
длина продукта составляет не более 3 т.п.н. С
помощью смеси различных полимераз и при
определённых условиях длина ПЦР-фрагмента
может достигать 20—40 тысяч пар нуклеотидов.
3. ПЦР
4. ПЦР
Компоненты реакции:• ДНК-матрица, содержащая участок ДНК, который требуется
амплифицировать.
• Два праймера, комплементарные противоположным
концам разных цепей требуемого фрагмента ДНК (ForwardReverse, Sense Antisense, Upper-Lower).
• Термостабильная ДНК-полимераза
• Дезоксирибонуклеозидтрифосфаты (dATP, dGTP, dCTP, dTTP).
• Ионы Mg2+, необходимые для работы полимеразы.
• Буферный раствор, обеспечивающий необходимые условия
реакции
Специфичность ПЦР основана на образовании комплементарных
комплексов между матрицей и праймерами, короткими
синтетическими олигонуклеотидами длиной 18—30 оснований.
Каждый из праймеров комплементарен одной из цепей
двуцепочечной матрицы и ограничивает начало и конец
амплифицируемого участка.
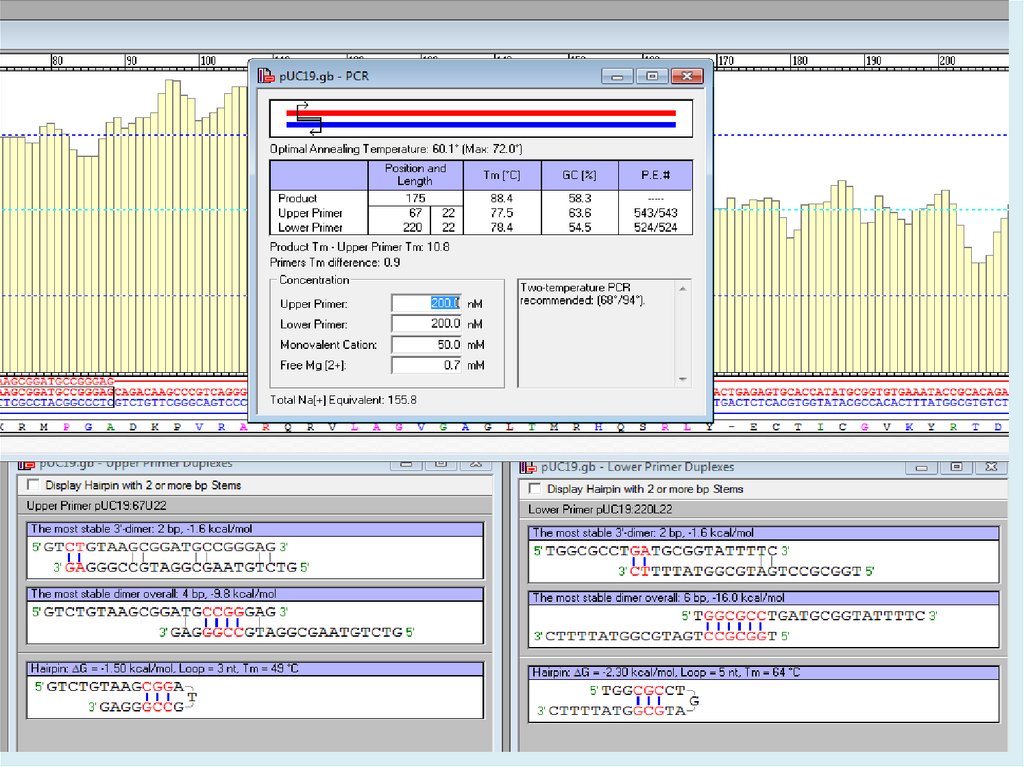
5. Tm
• Важнейшая характеристика праймеров — температураплавления (Tm) комплекса праймер-матрица.
• Tm — температура, при которой половина ДНК-матриц
образует комплекс с олигонуклеотидным праймером.
Простой расчет оптимальной температуры отжига
праймера:
Tm = [(A+T) x 2 °C] + [(G+C) x 4 °C](если суммарная длина
олигонуклеотида не превышает 20 оснований)
Tm = 22 + 1.46([2 x (G+C)] + (A+T))(если суммарная длина
олигонуклеотида составляет 20-30 оснований) или
Tm= 64.9 +41 x (G+C-16.4)/(A+T+G+C)
Обычно мы выбираем температуру отжига на 3-5oС
меньше, чем температура плавления олигонуклеотидов.
6. Tm
хорошие программы требуют для определения Tm заданиядвух параметров:
• концентрации праймеров;
• концентрации соли
L=G+C+A+T
Tm= 100.5 + (41 x (G+C)/(A+T+G+C)) - (820/(A+T+G+C)) + 16.6 x
log10([Na+]) или для длинных олигонуклеотидов:
Tm= 81.5 + (41 x (G+C)/(A+T+G+C)) - (500/(A+T+G+C)) + 16.6 x
log10([Na+])
Концентрация соли = [Na+]+[K+]+0.7[Tris].
7. Tа (annealing)
Более низкая температура отжига появление неспецифических продуктов.Более высокая температура отжига - более
высокая специфичность. Но как только она
превышает некую критическую (для данной
пары праймеров), количество продукта
начинает резко снижаться.
8.
При выборе праймеров также желательнопридерживаться следующих критериев:
• GC-состав ~ 40—60 %;
• близкие Tm праймеров (отличия не более, чем на 5
°C);
• желательно, чтобы на 3’-конце был гуанин или
цитозин / последние несколько нуклеотидов 3' конца праймера содержали GC-основания
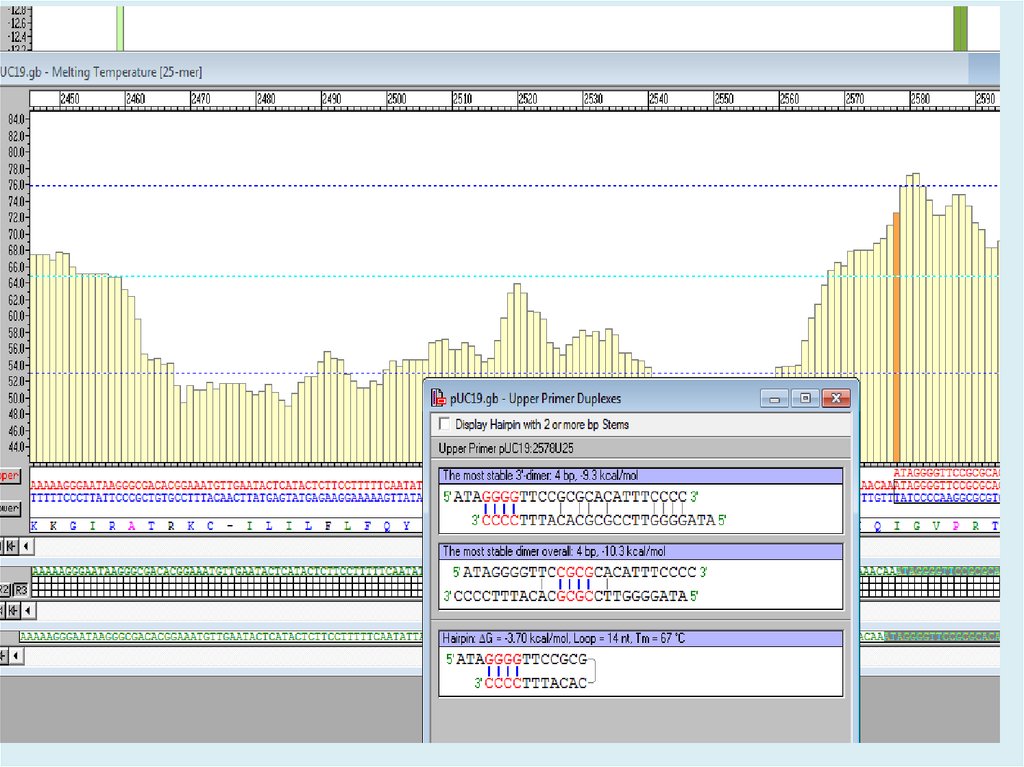
• отсутствие неспецифических вторичных структур —
шпилек и димеров (особенно на 3’-конце);
9.
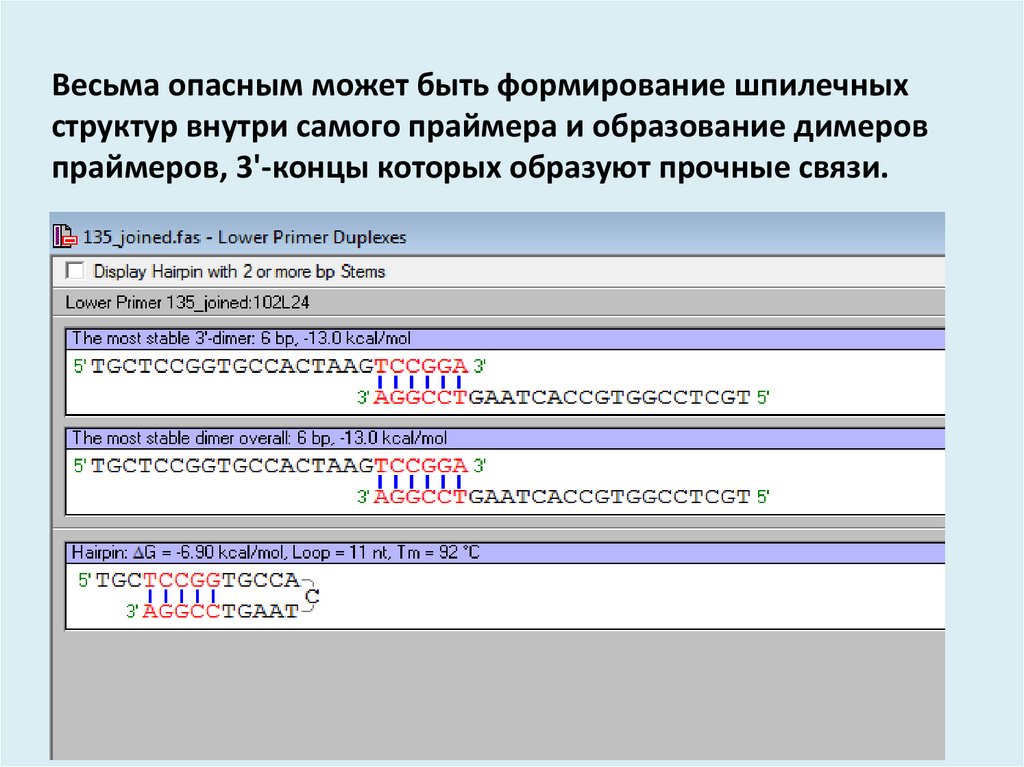
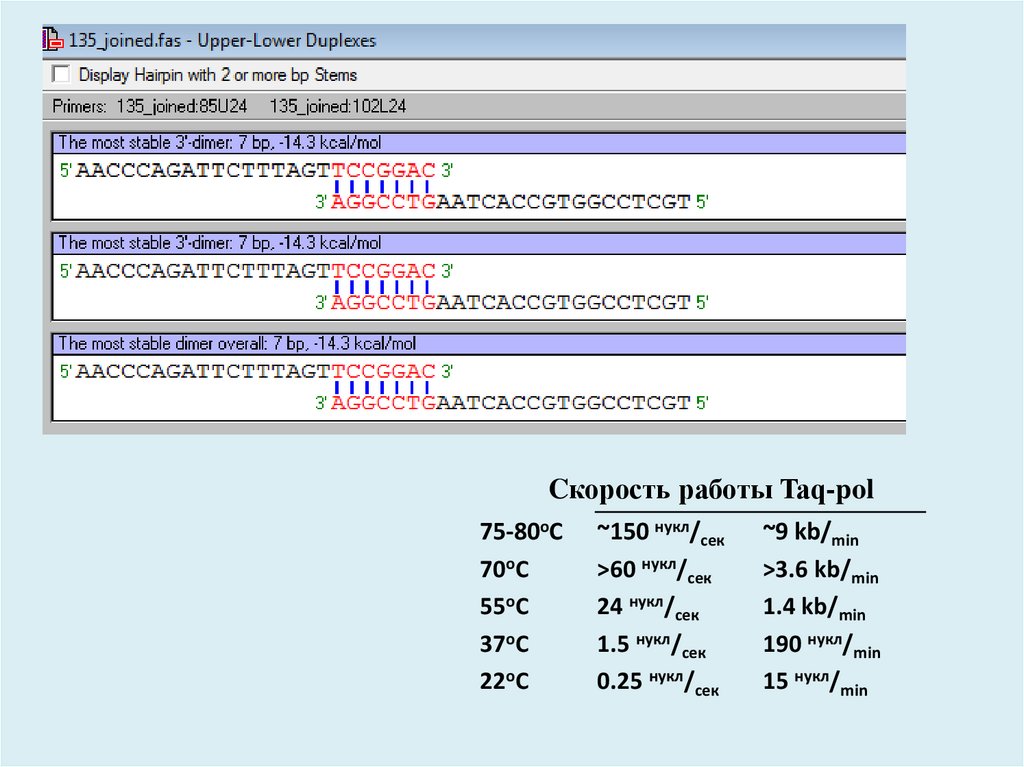
Весьма опасным может быть формирование шпилечныхструктур внутри самого праймера и образование димеров
праймеров, 3'-концы которых образуют прочные связи.
10.
Cкорость работы Taq-pol75-80oС
70oС
55oС
37oС
22oС
~150 нукл/сек
>60 нукл/сек
24 нукл/сек
1.5 нукл/сек
0.25 нукл/сек
~9 kb/min
>3.6 kb/min
1.4 kb/min
190 нукл/min
15 нукл/min
11.
12.
Разновидности ПЦРПЦР с использованием горячего старта (Hot-start
PCR) .
Ступенчатая ПЦР (Touchdown PCR )
ПЦР длинных фрагментов ( Long-range PCR) .
Вложенная ПЦР (Nested PCR )
ПЦР с обратной транскрипцией (Reverse Transcription
PCR, RT-PCR )
RAPD ( Random Amplification of Polymorphic DNA).
Асимметричная ПЦР (Asymmetric PCR).
Мультиплексная ПЦР.
Количественная ПЦР (Quantitative PCR, Q-PCR )
или ПЦР в реальном времени .
Инвертированная ПЦР (Inverse PCR )
Метод молекулярных колоний (ПЦР в геле, Colony PCR Colony) .
ПЦР с быстрой амплификацией концов кДНК ( Rapid
amplification of cDNA ends, RACE-PCR).
Групп-специфическая ПЦР ( group-specific PCR) .
и т.д.
13. Разновидности ПЦР для рещения проблемы «плохих» праймеров
• ПЦР с использованием горячего старта (Hot-startPCR) .
• Ступенчатая ПЦР (Touchdown PCR )
Параметры обычной PCR реакции.
Td=95oC, 1';
25-30 циклов:
Td=94oC, 10'',
Ta=55 oC, 30'',
Te=72oC, 1';
T=72oC, 5';
T=4oC.
14.
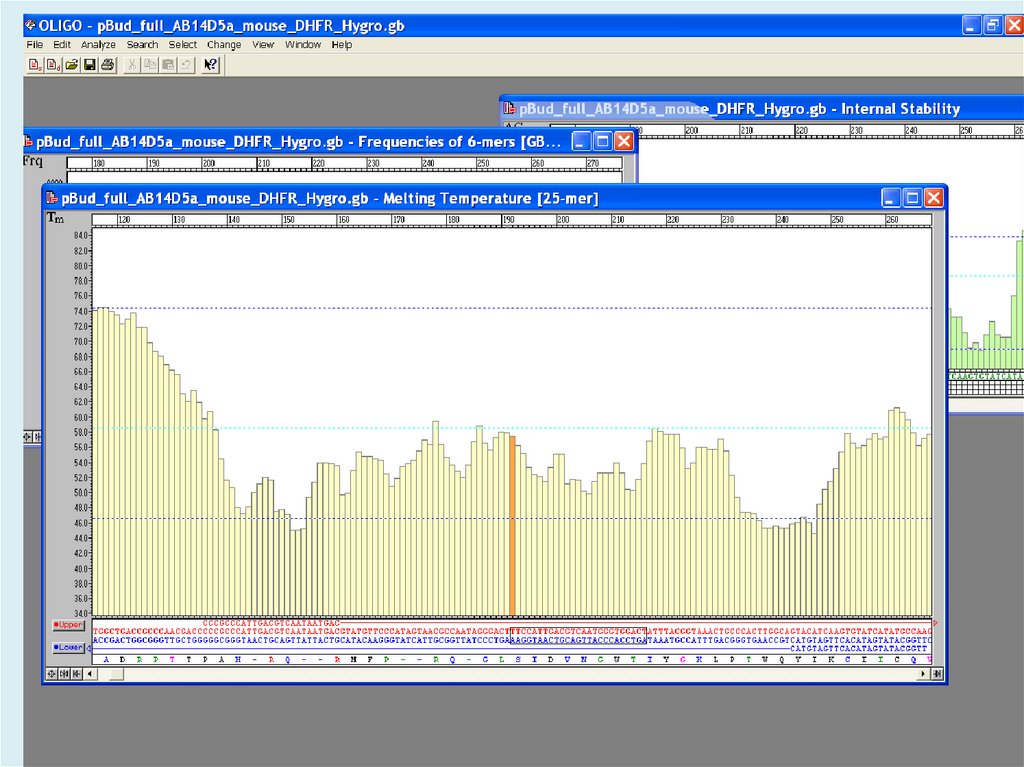
Vector NTIOligo
BioEdit
Oligo Calc: Oligonucleotide Properties Calculator
http://biotools.nubic.northwestern.edu/OligoCalc.html
и т.д.
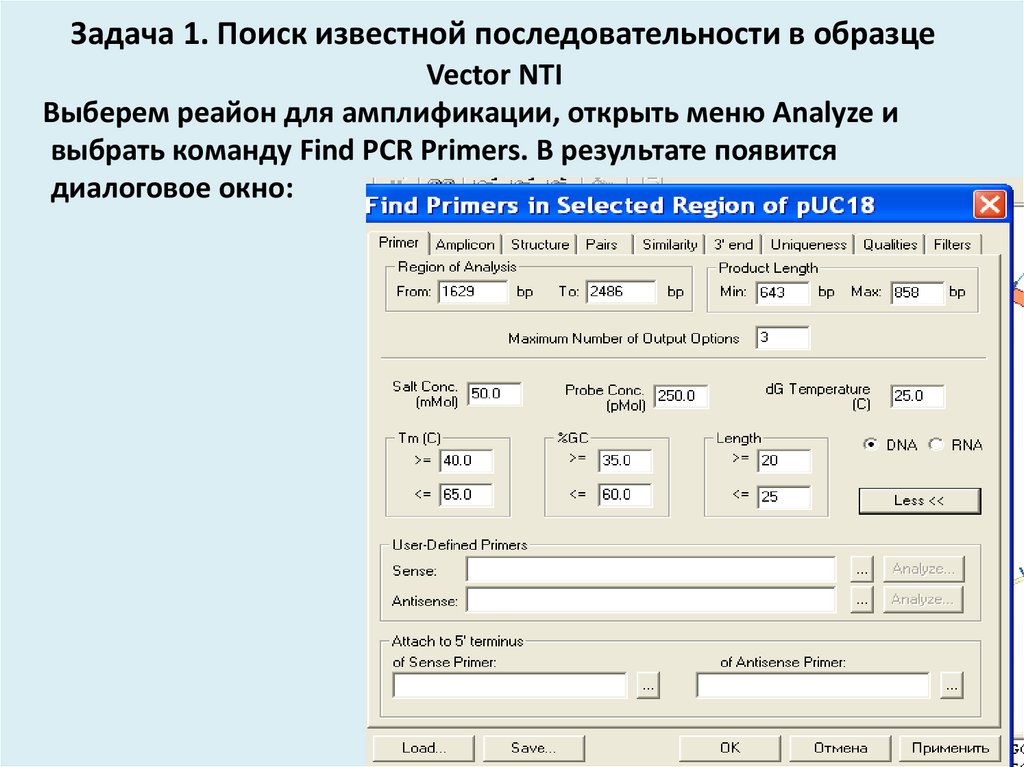
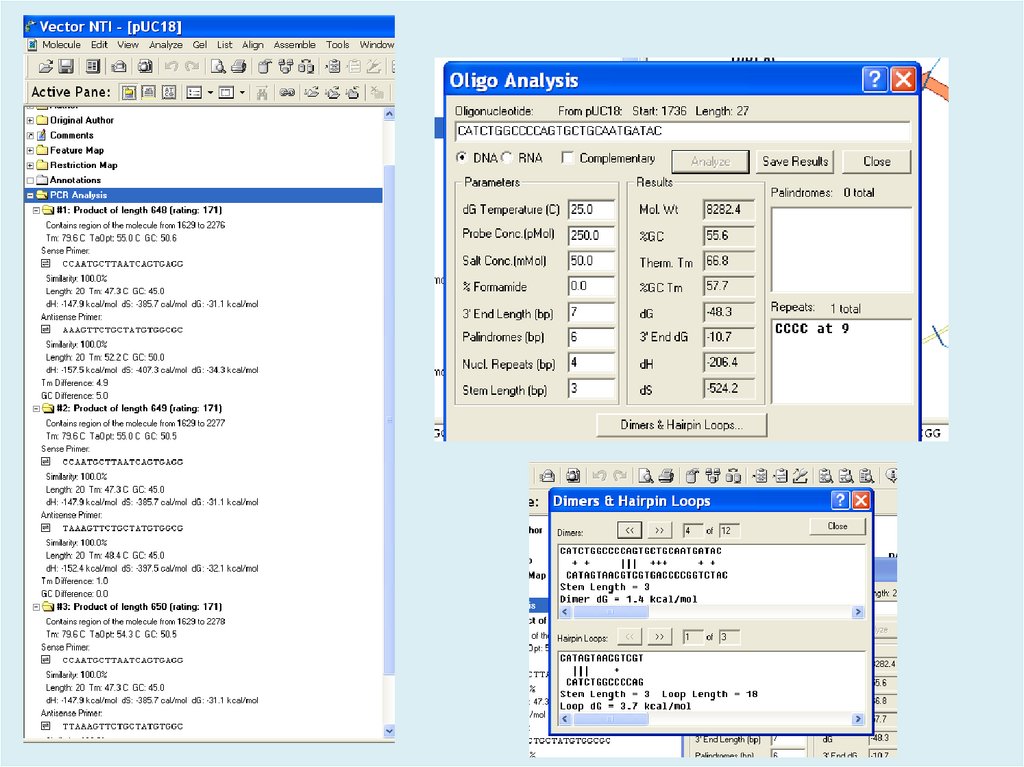
15. Задача 1. Поиск известной последовательности в образце
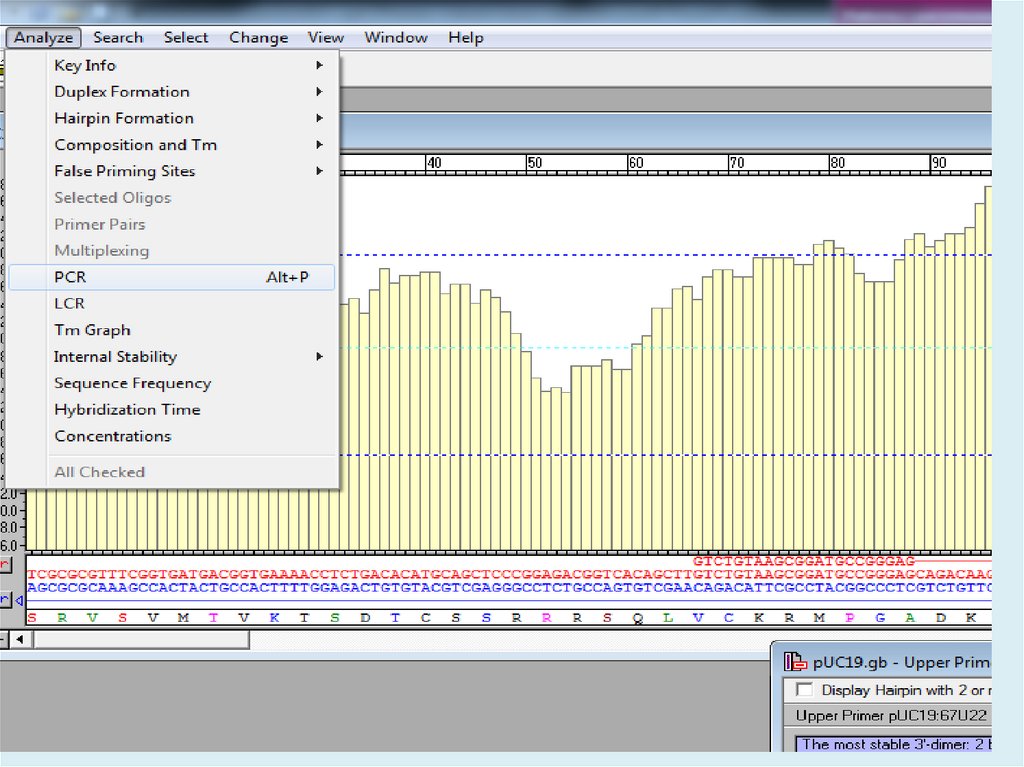
Vector NTIВыберем реайон для амплификации, открыть меню Analyze и
выбрать команду Find PCR Primers. В результате появится
диалоговое окно:
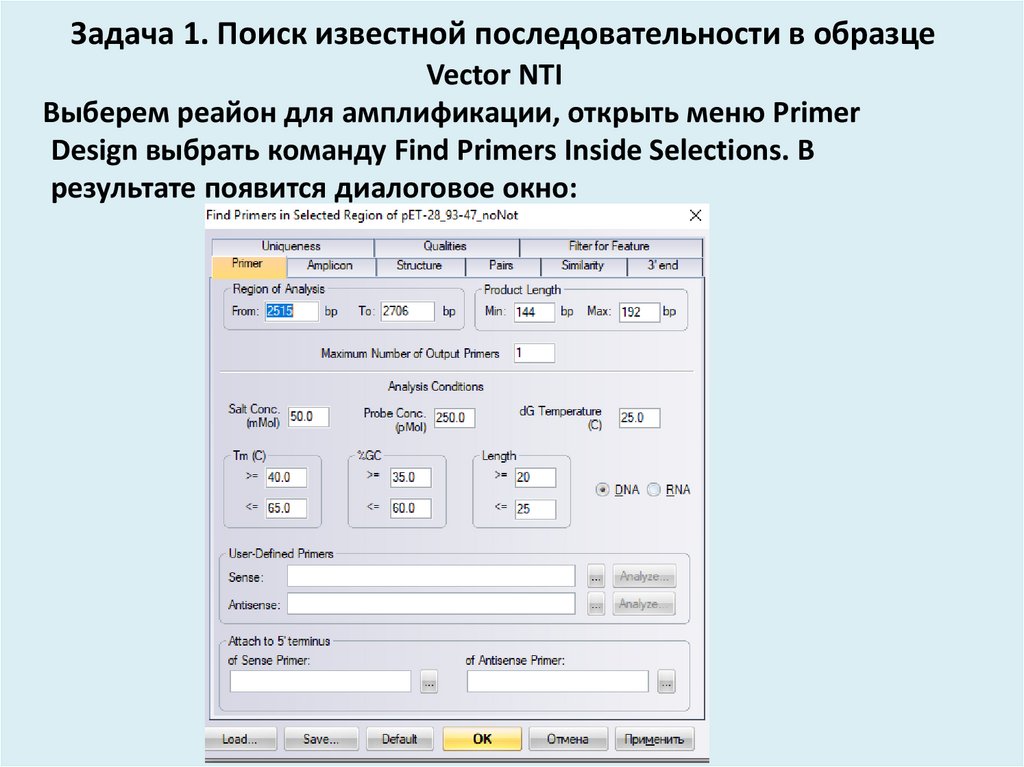
16. Задача 1. Поиск известной последовательности в образце
Vector NTIВыберем реайон для амплификации, открыть меню Primer
Design выбрать команду Find Primers Inside Selections. В
результате появится диалоговое окно:
17.
Диалоговое окно позволяет сделать следующее:Автоматически подобрать соответствующие
праймеры;
Задать праймеры собственноручно;
Добавить к 5’ концу праймера короткую
последовательность;
Сформулировать требования к гомологии
праймера (Primer Similarity);
Задать биохимические и структурные параметры
праймеров;
Определить требования к качеству праймеров.
18.
19.
20.
21.
22.
23.
24.
25.
26.
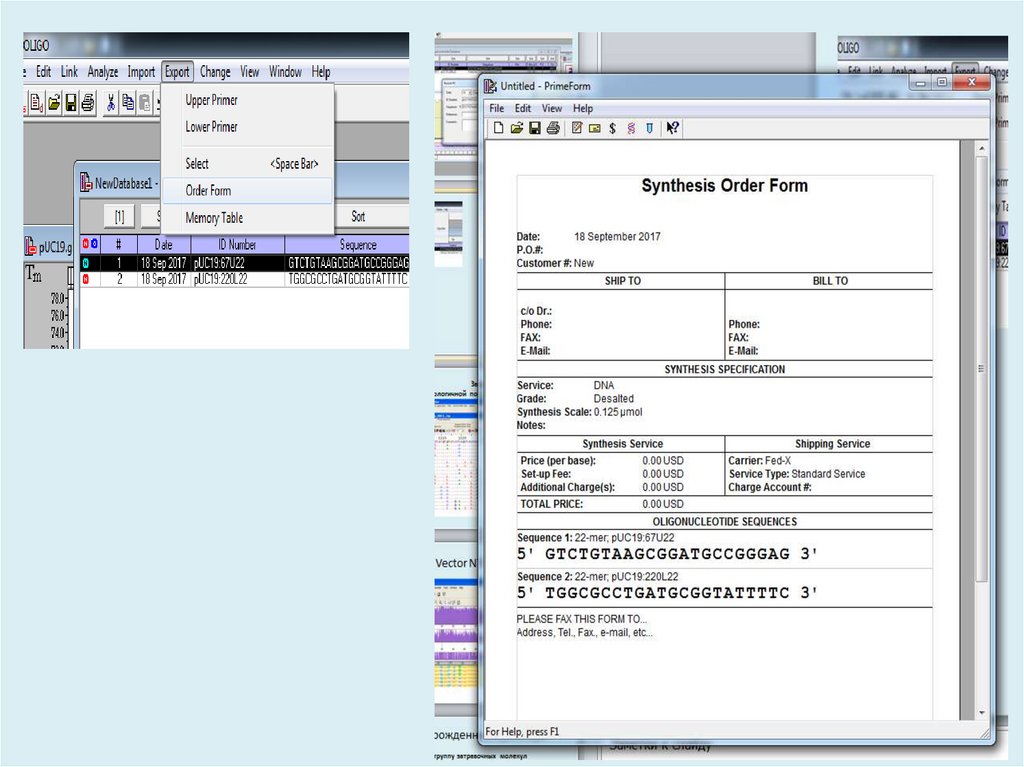
27. Заявка на синтез праймеров
• Последовательности от 5’ к 3’ концу(Antisense праймер необходимо записать
как обратно комплиментарный)
• «Буквы» только английские
• Масштаб синтеза и требуемый вид
очистки
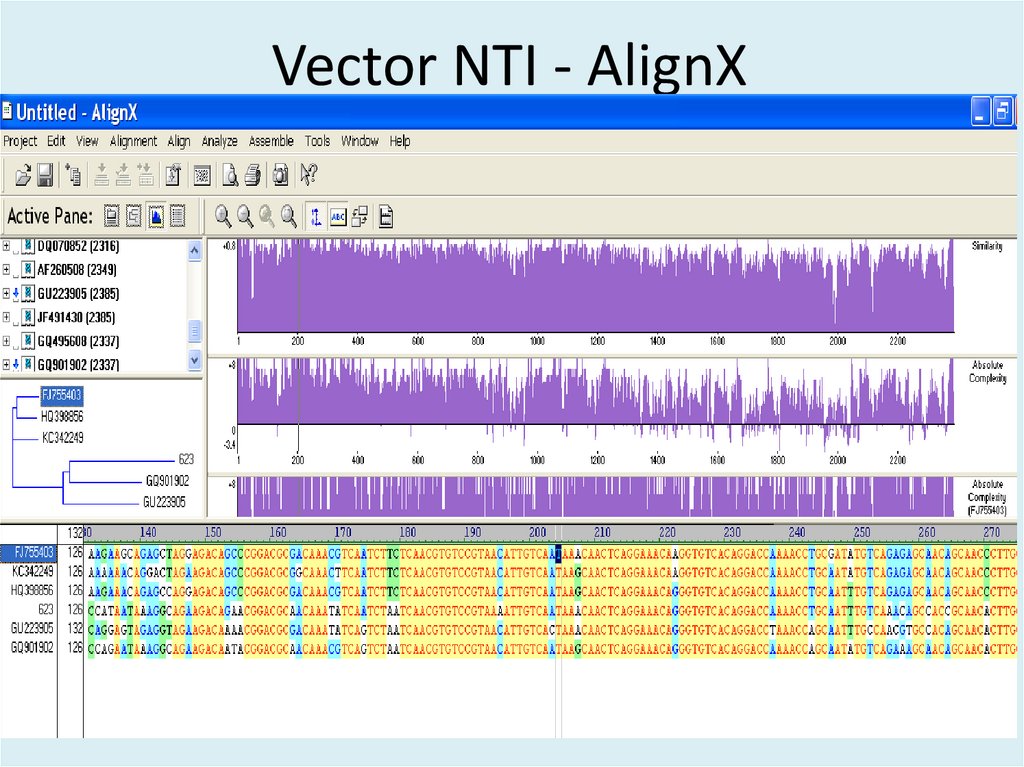
28. Задача 2. Поиск гомологичной последовательности в образце
29. Vector NTI - AlignX
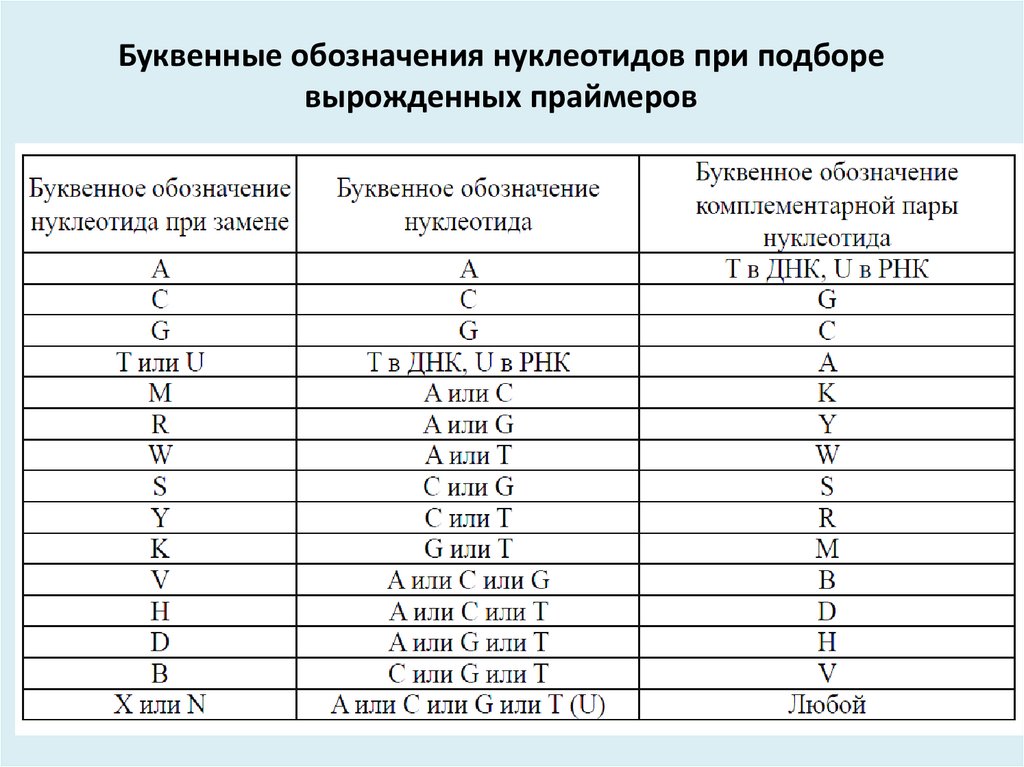
30. Вырожденные праймеры
Отдельную группу затравочных молекулпредставляют собой так называемые вырожденные
праймеры, составленные или на основе
относительно консервативной последовательности
нуклеотидов какого-либо гена из мультигенного
семейства. Зачастую такой праймер будет
представлять собой весьма гетерогенную смесь
олигонуклеотидов. Применение остатков инозина,
образующего комплементарные пары с любым из
четырех дезоксинуклеотидов, в вырожденных
местах таких праймеров в значительной степени
решает эту проблему
31. Буквенные обозначения нуклеотидов при подборе вырожденных праймеров
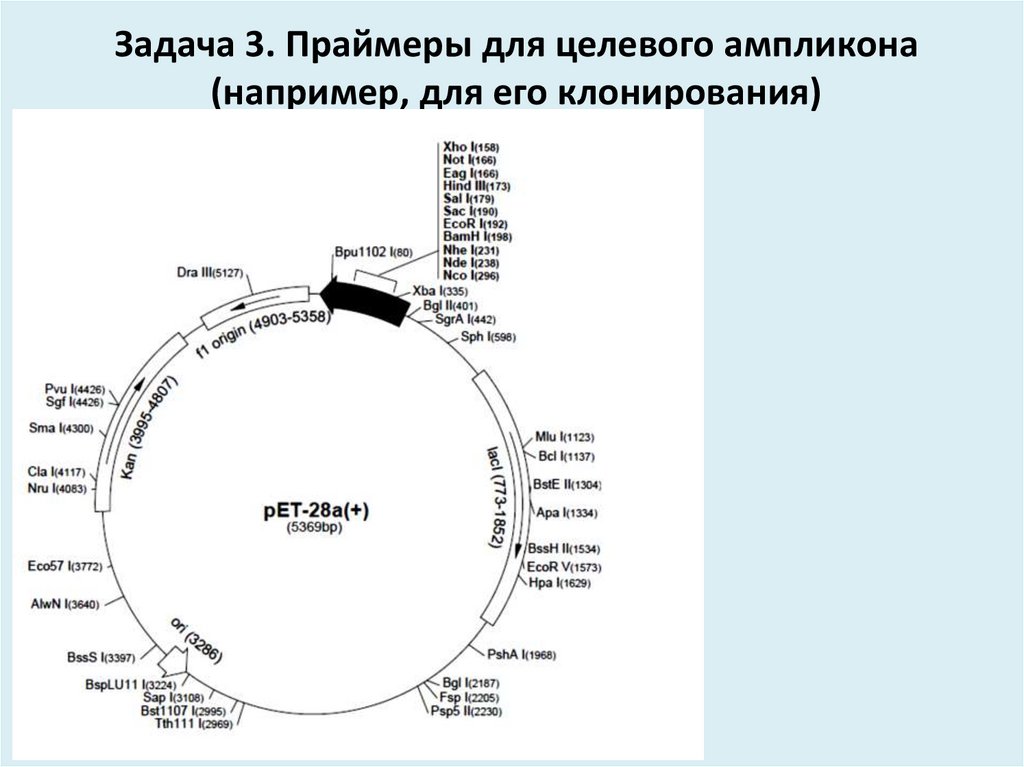
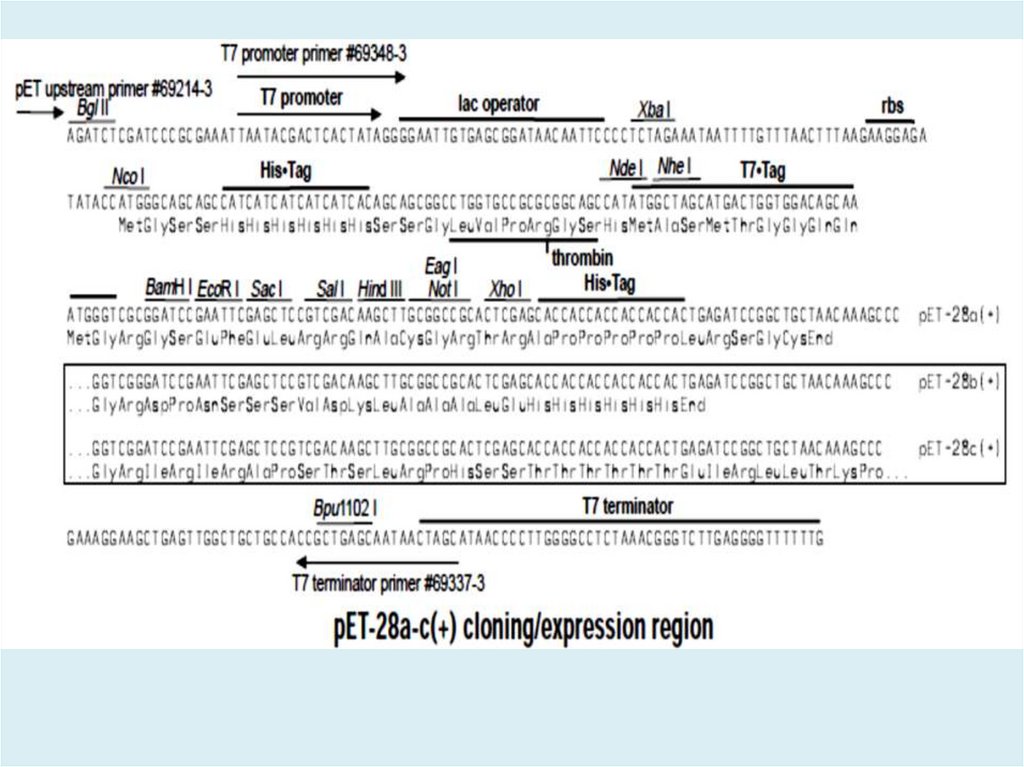
32. Задача 3. Праймеры для целевого ампликона (например, для его клонирования)
33.
1. Выбор вектора.2. Подбор сайтов рестрикции в векторе.
3. Анализ этих сайтов в целевом фрагменте.
4. В случае слитых последовательностей –
проверка непрерывности ОРТ.
5. Расчет праймеров и введение
последовательностей сайтов рестрикции на 5'
концах праймеров (дополнительные
нуклеотиды для работы ферментов.
https://www.neb.com/
34.
35.
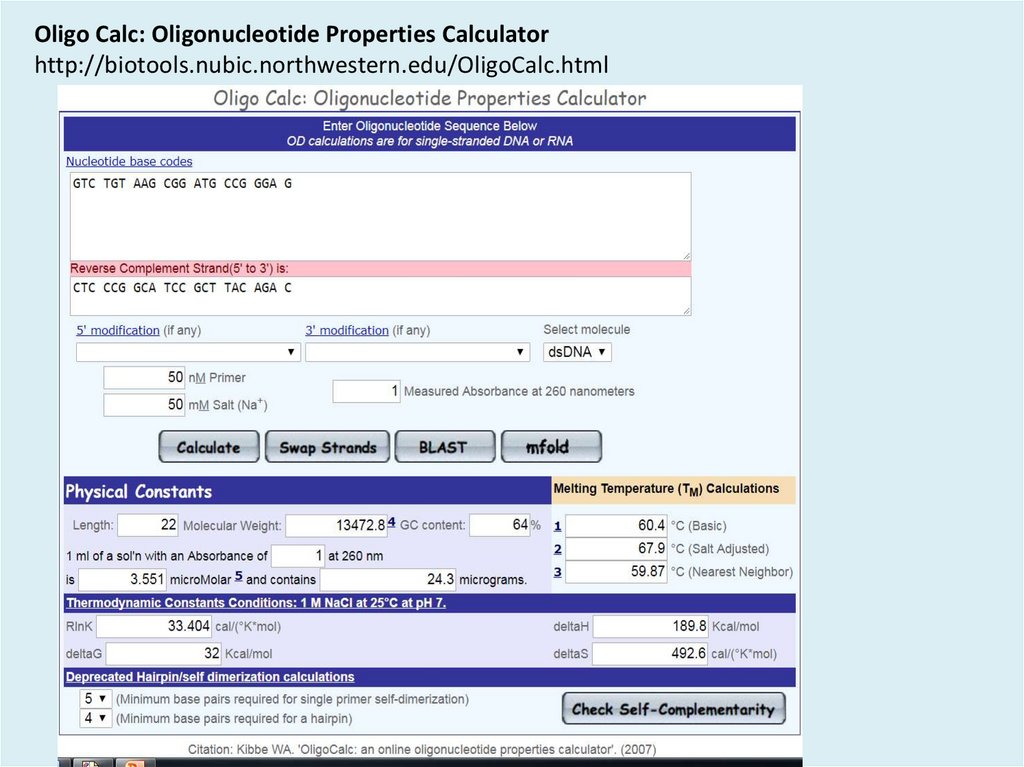
Oligo Calc: Oligonucleotide Properties Calculatorhttp://biotools.nubic.northwestern.edu/OligoCalc.html
36.
Oligo Calc: Oligonucleotide Properties Calculatorhttp://biotools.nubic.northwestern.edu/OligoCalc.html
37.
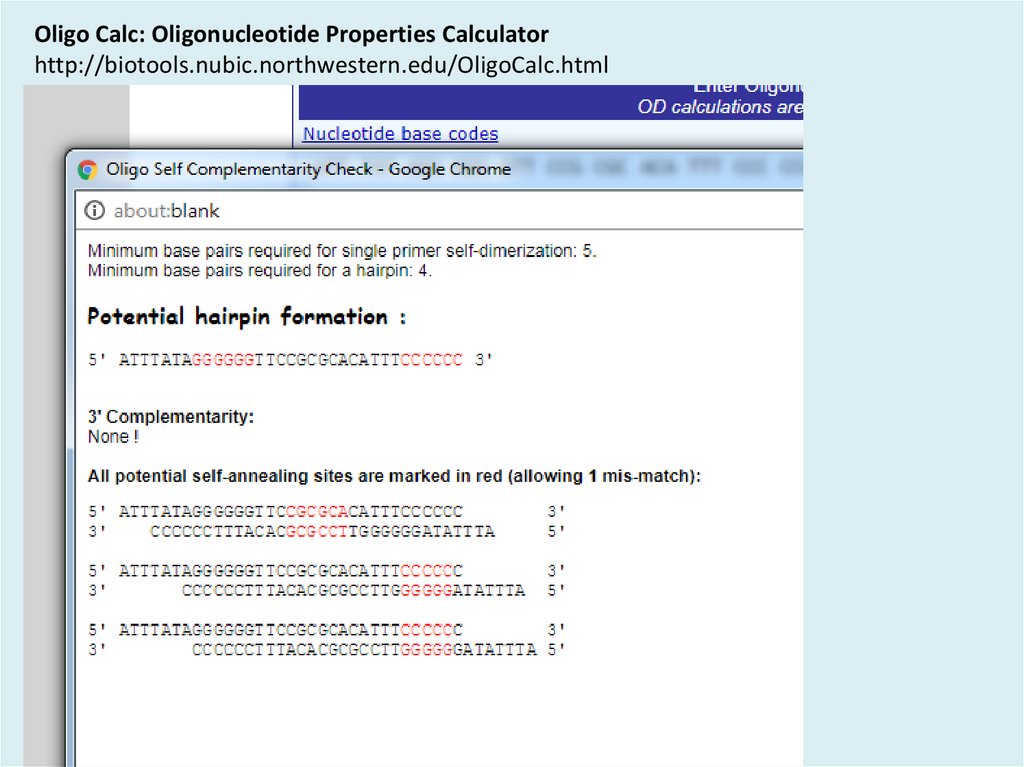
Oligo Calc: Oligonucleotide Properties Calculatorhttp://biotools.nubic.northwestern.edu/OligoCalc.html
38.
39. Праймеры для секвенирования
• Оптимальная температура плавления 50600С• Расположение праймеров на обеих цепях
изучаемой последовательности
(перекрывание, длина чтения)
40.
• Primers should be at least 18 bases long to ensure goodhybridization.
• Avoid runs of an identical nucleotide, especially guanine, where
runs of four or more Gs should be avoided.
Избегать 4 и более G
• Keep the G-C content in the range 30–80%.
• For cycle sequencing, primers with melting temperatures (Tm)
above 45 °C produce better results than primers with lower Tm.
• For primers with a G-C content less than 50%, it may be
necessary to extend the primer sequence beyond 18 bases to
keep the Tm>45 °C.
• Use of primers longer than 18 bases also minimizes the chance
of having a secondary hybridization site on the target DNA.
• Avoid primers that have secondary structure or that can
hybridize to form dimers.
• Several computer programs for primer selection are available.
41.
ABI recommended guidelines when designing primers/probes for Quantitative assay· select the probe first and design the primers as close as possible to the probe without
overlapping it. Amplicons of 50 to 150 bp are strongly recommended.
· keep primer/probe GC content within 30-80%
· avoid runs of identical nucleotides, this is especially true for guanine, where runs of
four or more Gs should be avoided
When designing primers:
· Tm should be within 58°C to 60°C
· the last 5 bases at the 3 prime end should have no more than two G's or C's
· keep the annealing Ts of the primers as close as possible
· select primer pairs with minimal number of potential primer dimers and primer
hairpins as possible
When designing probes:
· Tm should be within 68°C to 70°C
· no Gs on the 5’ end
· select the strand that gives the probe more C than G bases
· make the TaqMan probes as short as possible, without being shorter than 13
nucleotides
42.
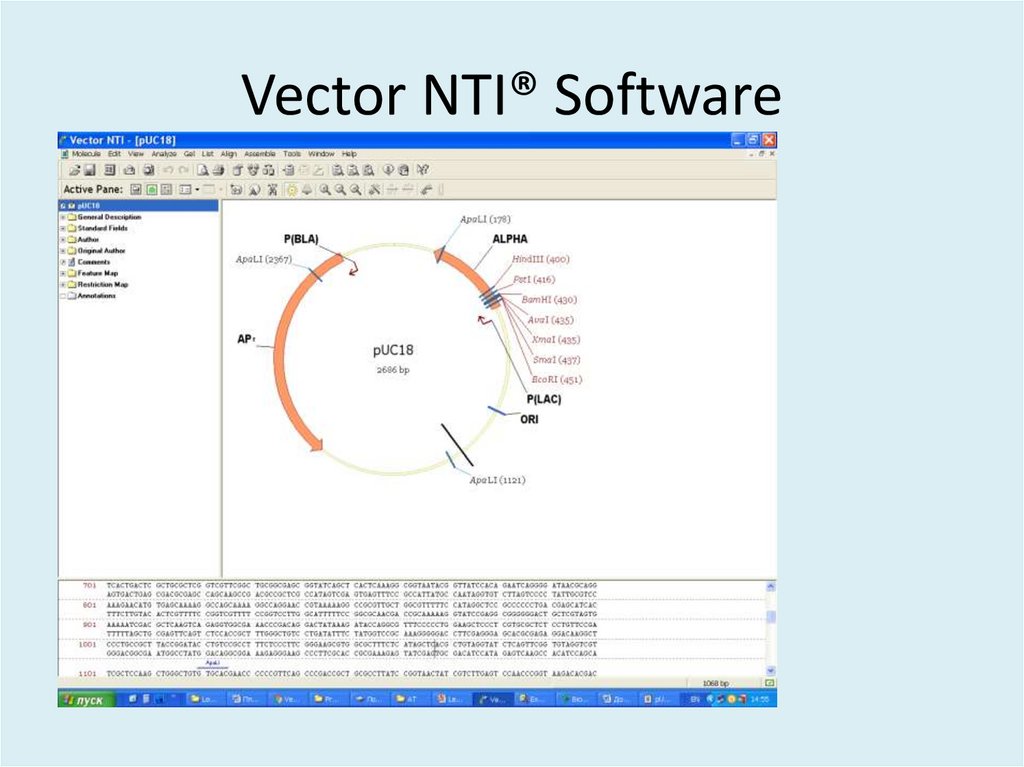
43. Vector NTI® Software
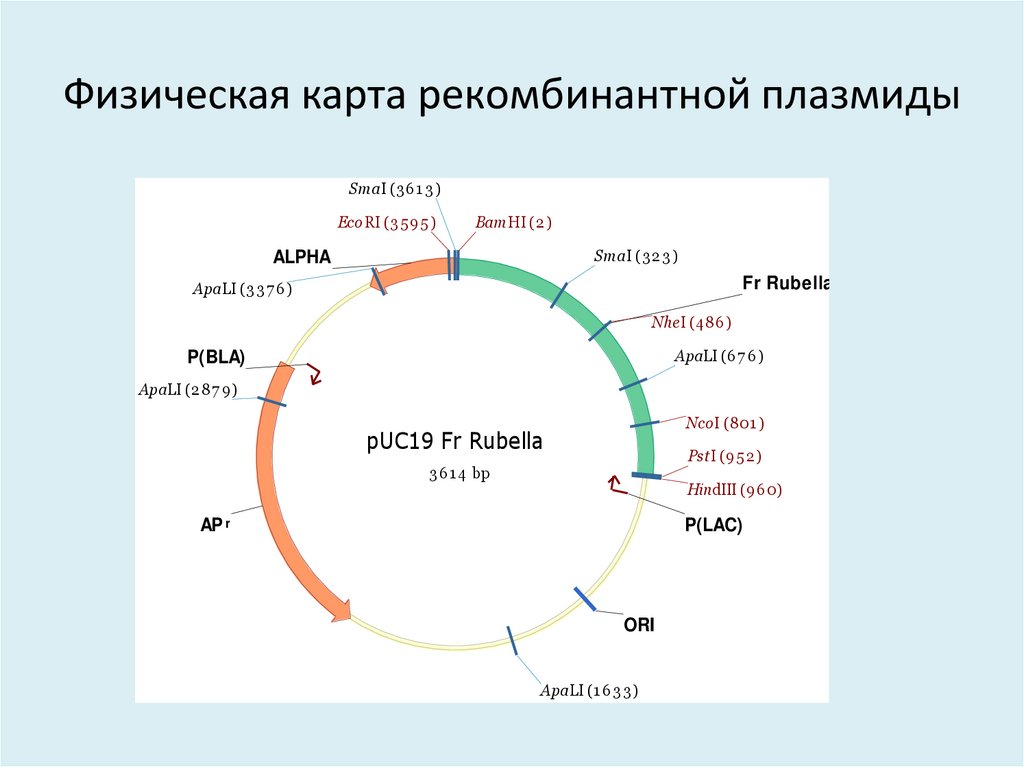
44. Физическая карта рекомбинантной плазмиды
SmaI (3 6 1 3 )Eco RI (3 5 9 5 )
BamHI (2 )
SmaI (3 2 3 )
ALPHA
Fr Rubella
ApaLI (3 3 7 6)
NheI (486 )
ApaLI (6 7 6 )
P(BLA)
ApaLI (2 87 9)
NcoI (801 )
pUC19 Fr Rubella
PstI (95 2 )
3 6 1 4 bp
HindIII (9 6 0)
AP r
P(LAC)
ORI
ApaLI (1 6 3 3 )
45.
Полезные Интернет-сайты:1. http://www.ncbi.nlm.nih.gov/
2. http://www.ebi.ac.uk/
3. http://web.expasy.org/
4. http://evolution.genetics.washington.edu/phylip/software.html#methods
5. http://blast.ncbi.nlm.nih.gov/Blast.cgi
6. http://mafft.cbrc.jp/alignment/server/index.html
7. http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form
8. http://molbiol.ru/
























 biology
biology








