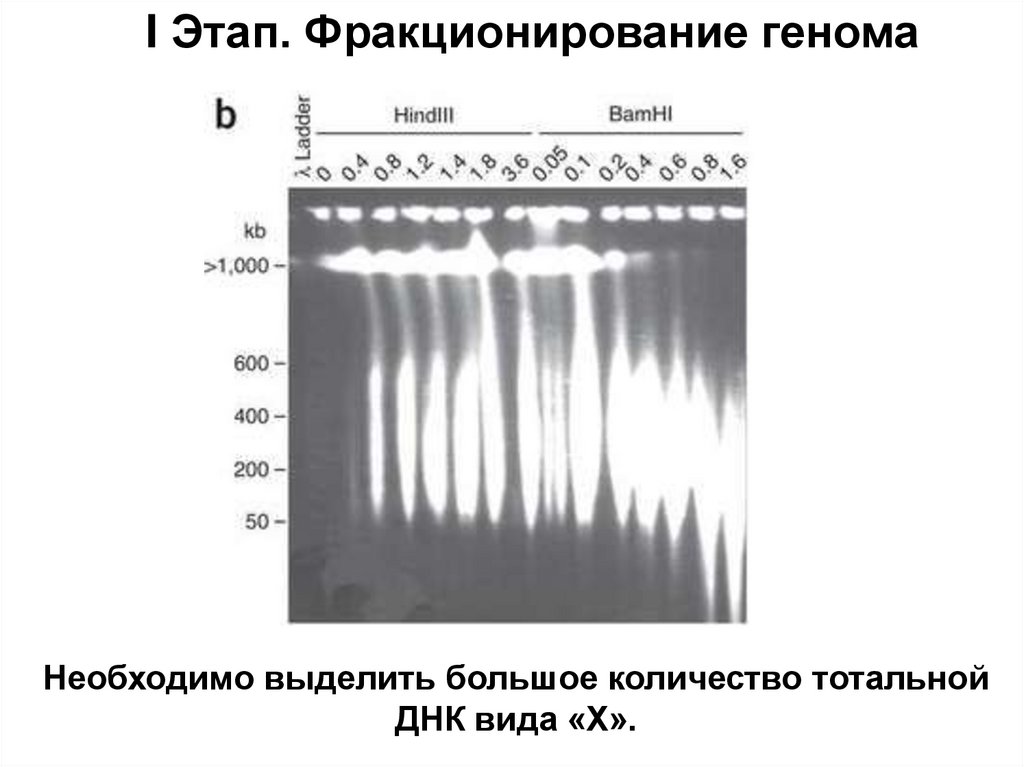
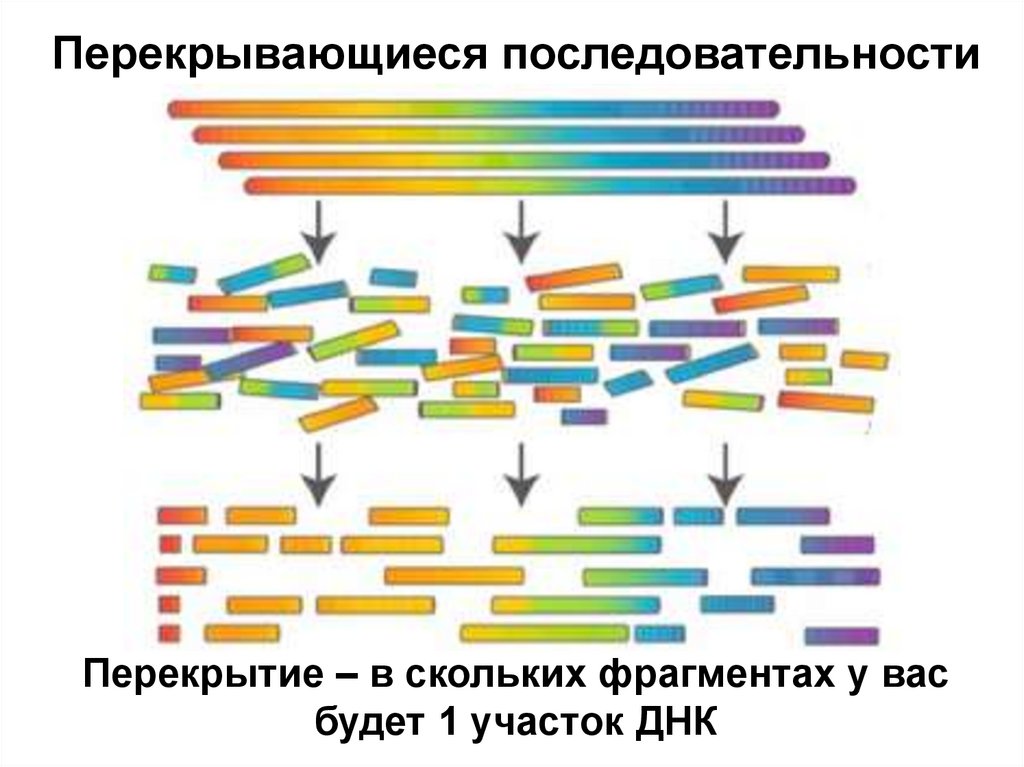
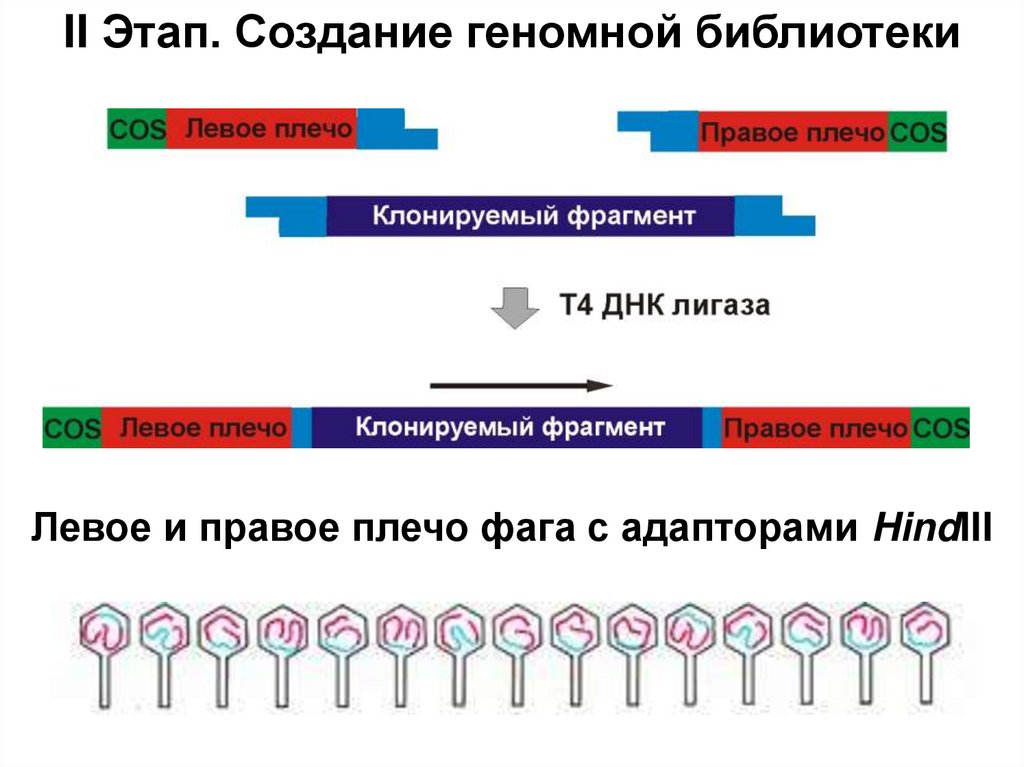
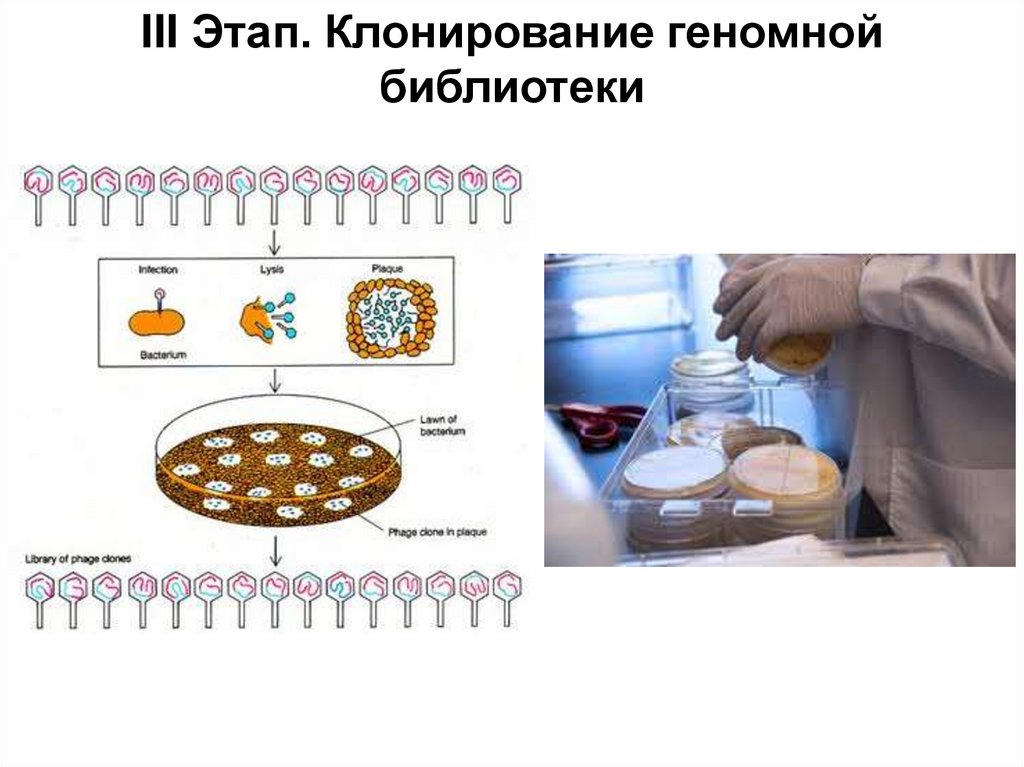
Similar presentations:
Основы генетической инженерии
1. Основы генетической инженерии
Герасимов Андрей Сергеевич, канд. биол. наук,доцент кафедры Биотехнологии ИББТ
2.
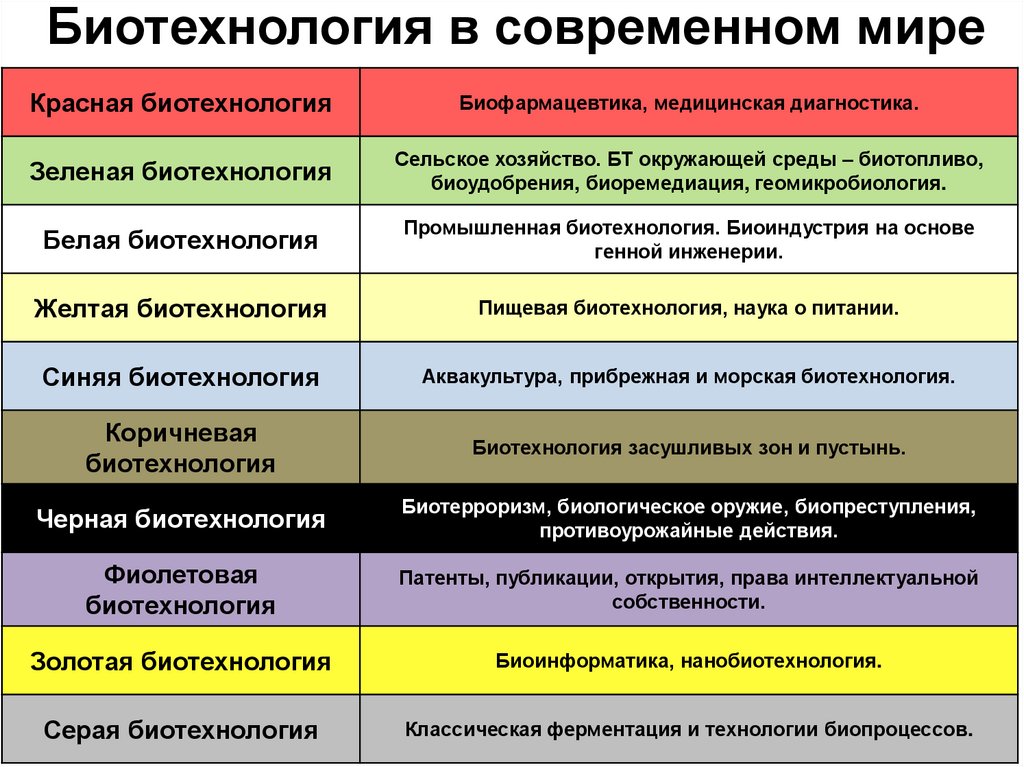
ЛИТЕРАТУРА3. Биотехнология в современном мире
Красная биотехнологияБиофармацевтика, медицинская диагностика.
Зеленая биотехнология
Сельское хозяйство. БТ окружающей среды – биотопливо,
биоудобрения, биоремедиация, геомикробиология.
Белая биотехнология
Промышленная биотехнология. Биоиндустрия на основе
генной инженерии.
Желтая биотехнология
Пищевая биотехнология, наука о питании.
Синяя биотехнология
Аквакультура, прибрежная и морская биотехнология.
Коричневая
биотехнология
Биотехнология засушливых зон и пустынь.
Черная биотехнология
Биотерроризм, биологическое оружие, биопреступления,
противоурожайные действия.
Фиолетовая
биотехнология
Патенты, публикации, открытия, права интеллектуальной
собственности.
Золотая биотехнология
Биоинформатика, нанобиотехнология.
Серая биотехнология
Классическая ферментация и технологии биопроцессов.
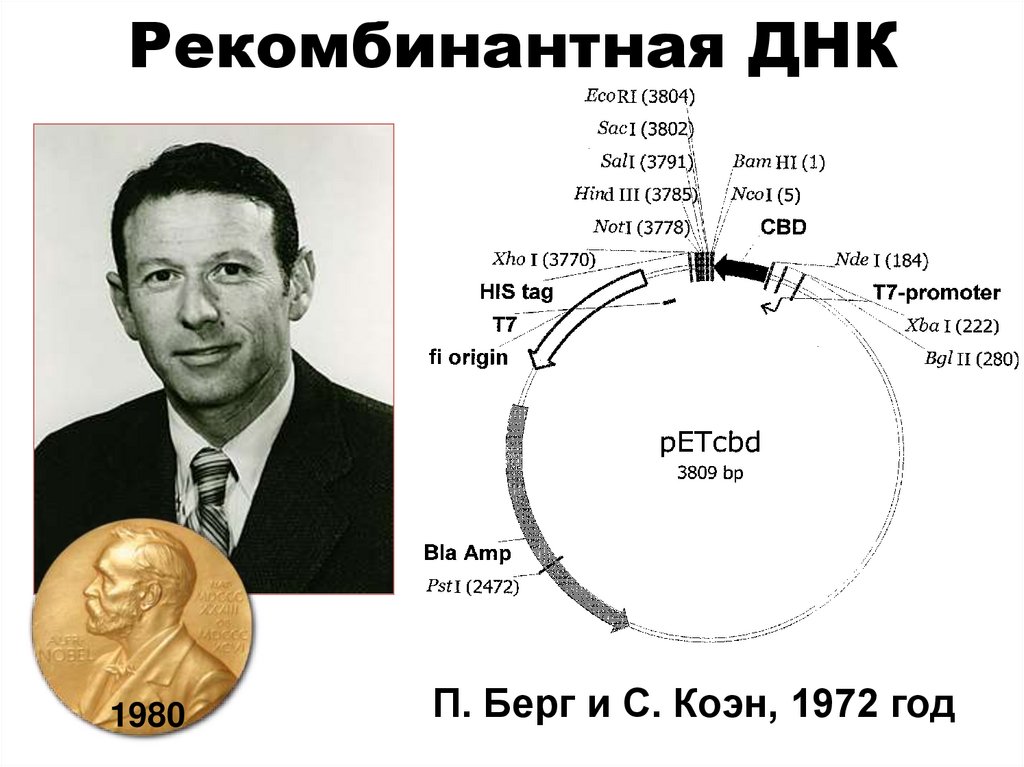
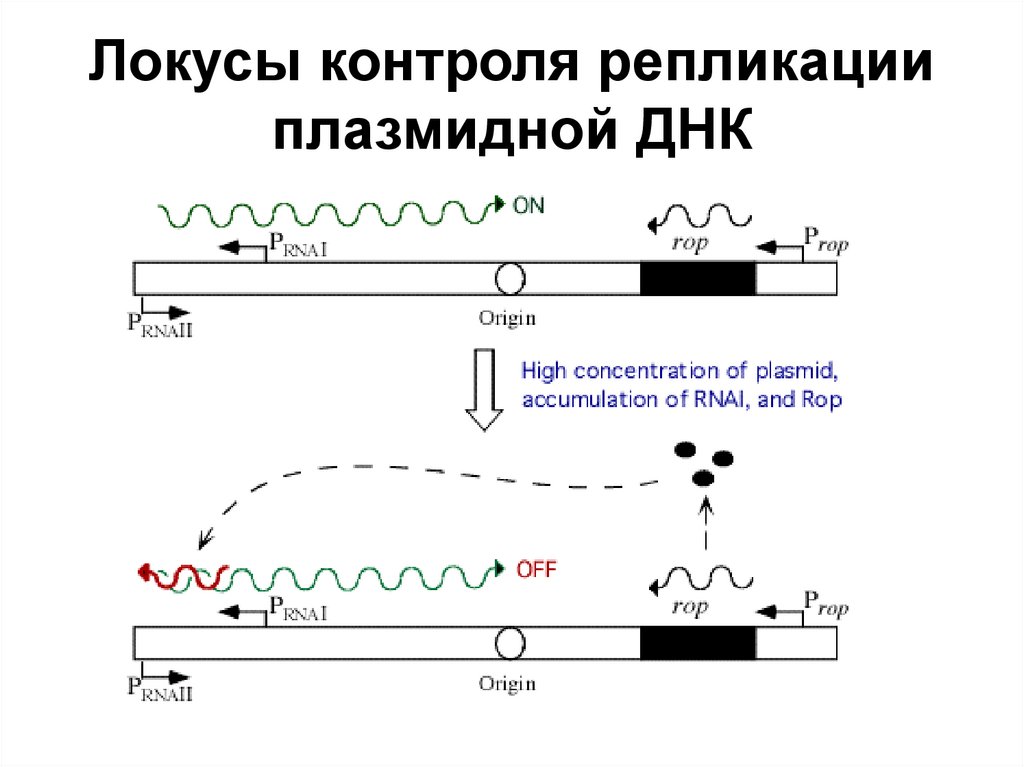
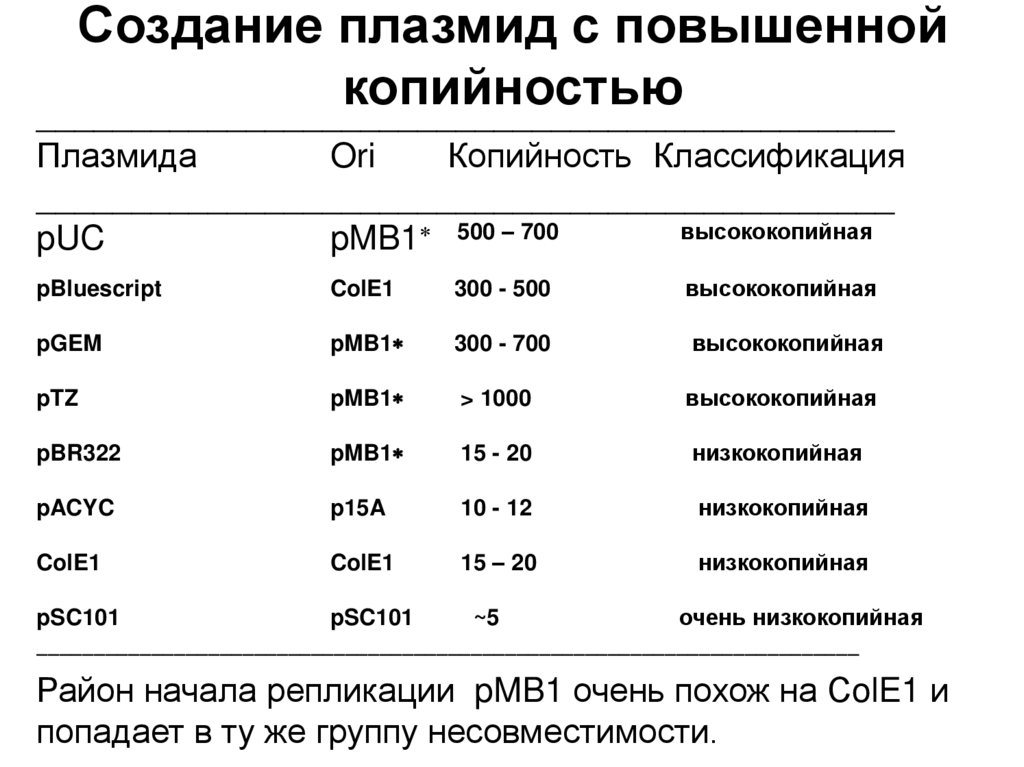
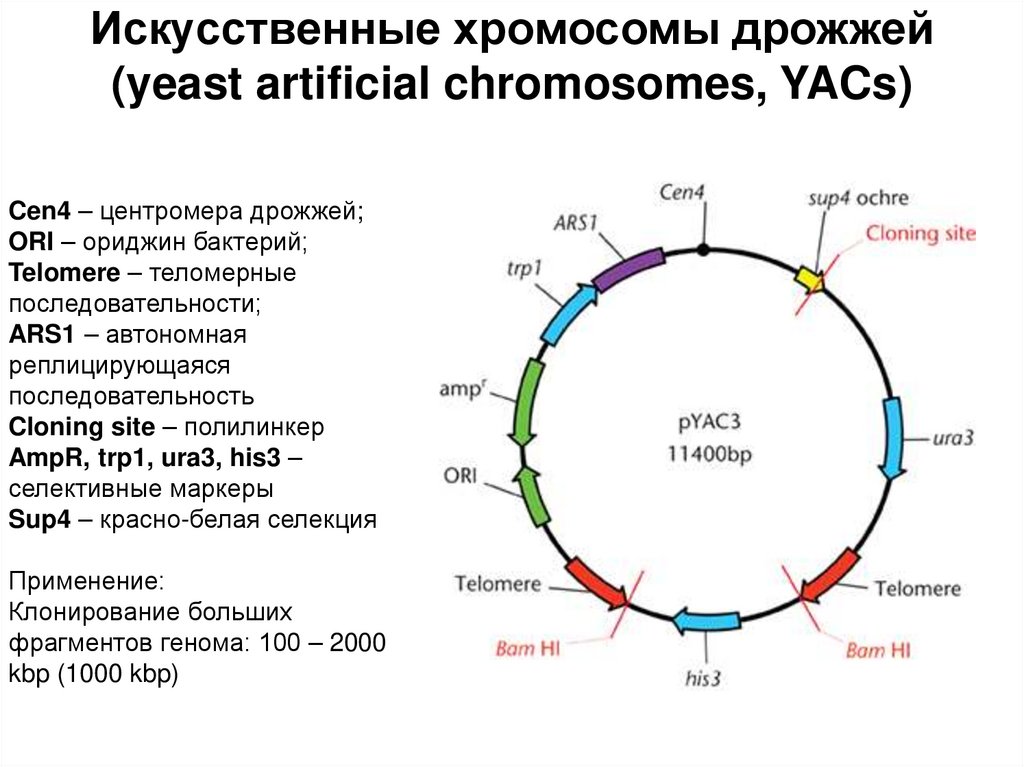
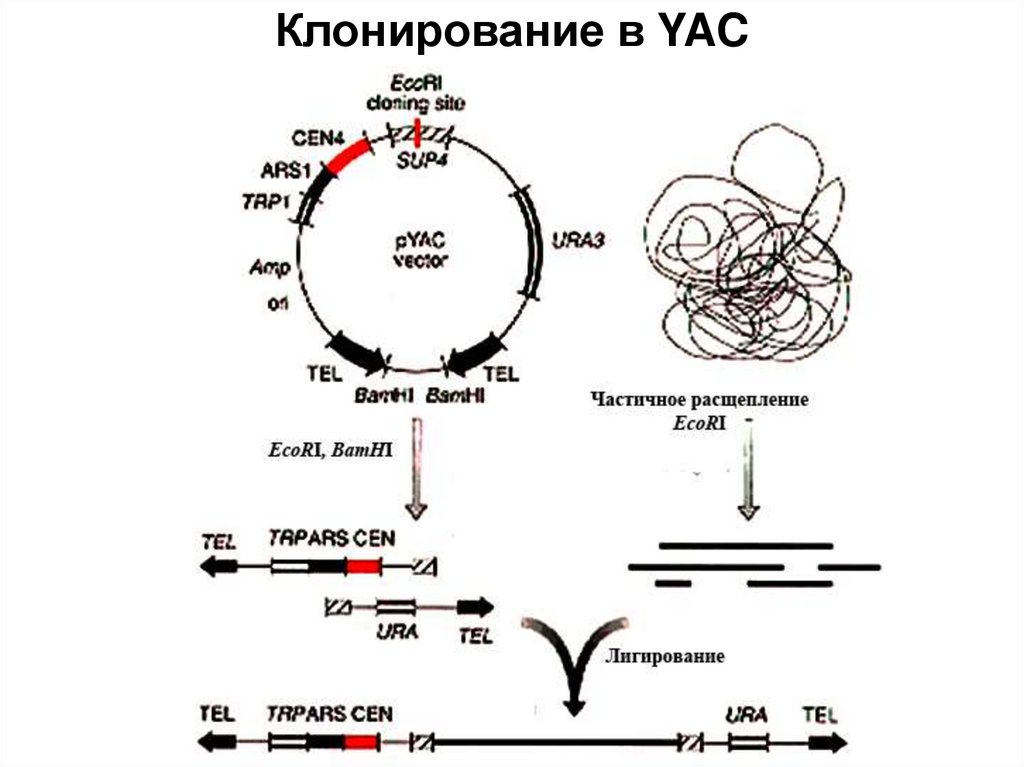
4. Рекомбинантная ДНК
1980П. Берг и С. Коэн, 1972 год
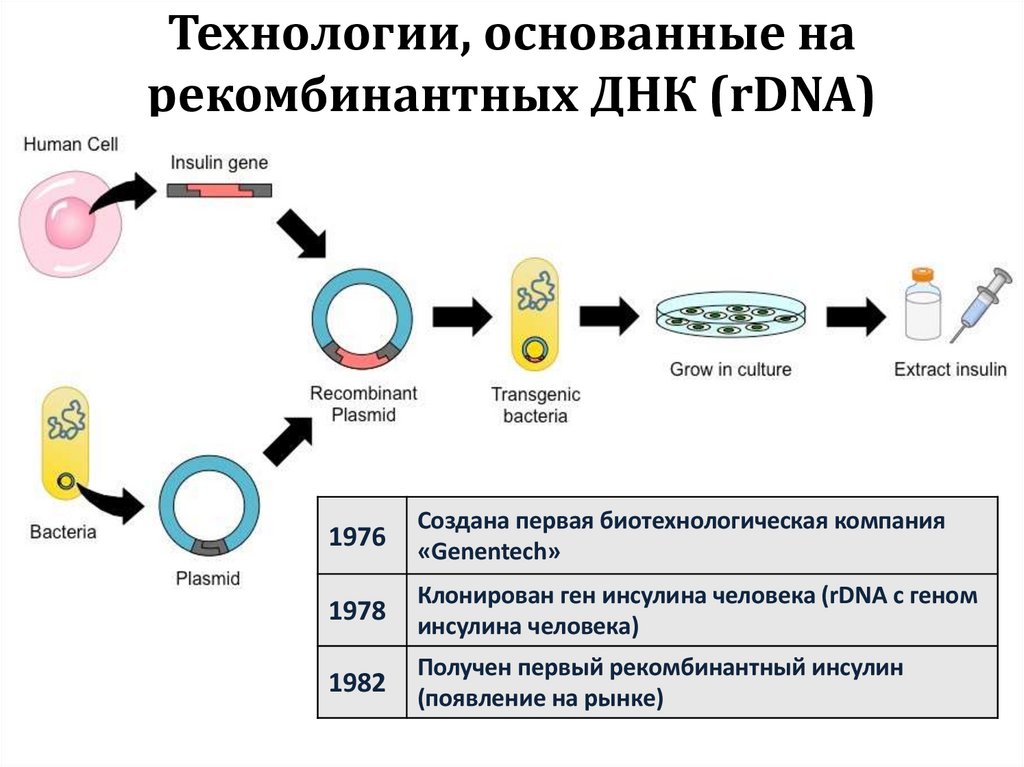
5. Технологии, основанные на рекомбинантных ДНК (rDNA)
1976Создана первая биотехнологическая компания
«Genentech»
1978
Клонирован ген инсулина человека (rDNA с геном
инсулина человека)
1982
Получен первый рекомбинантный инсулин
(появление на рынке)
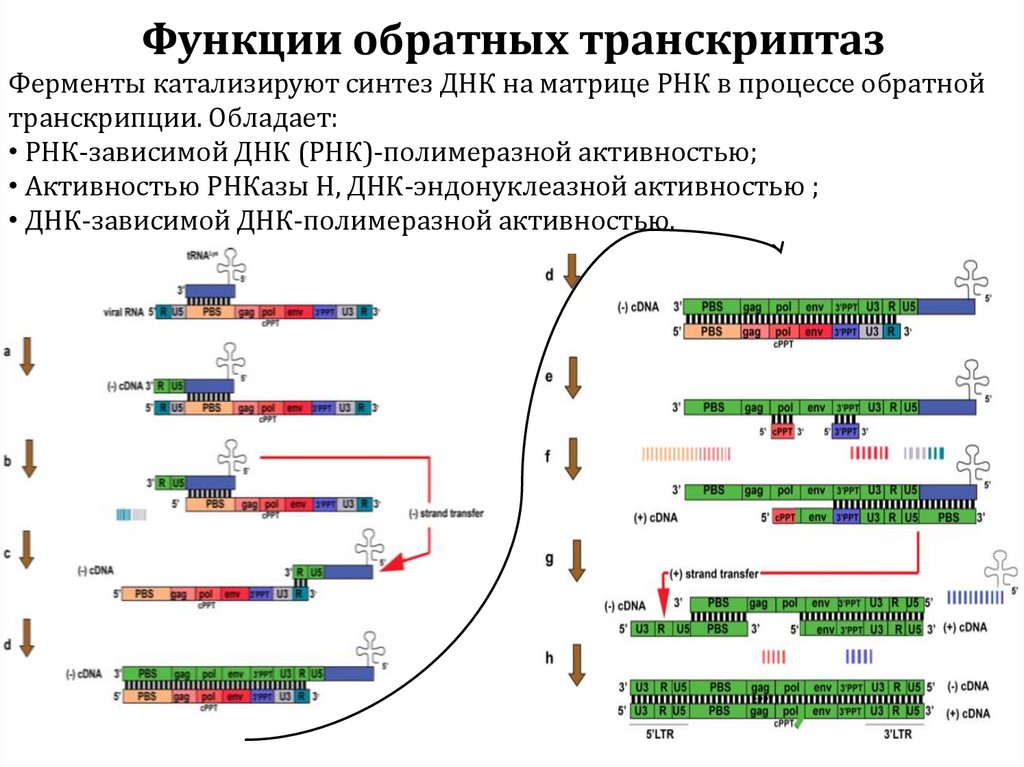
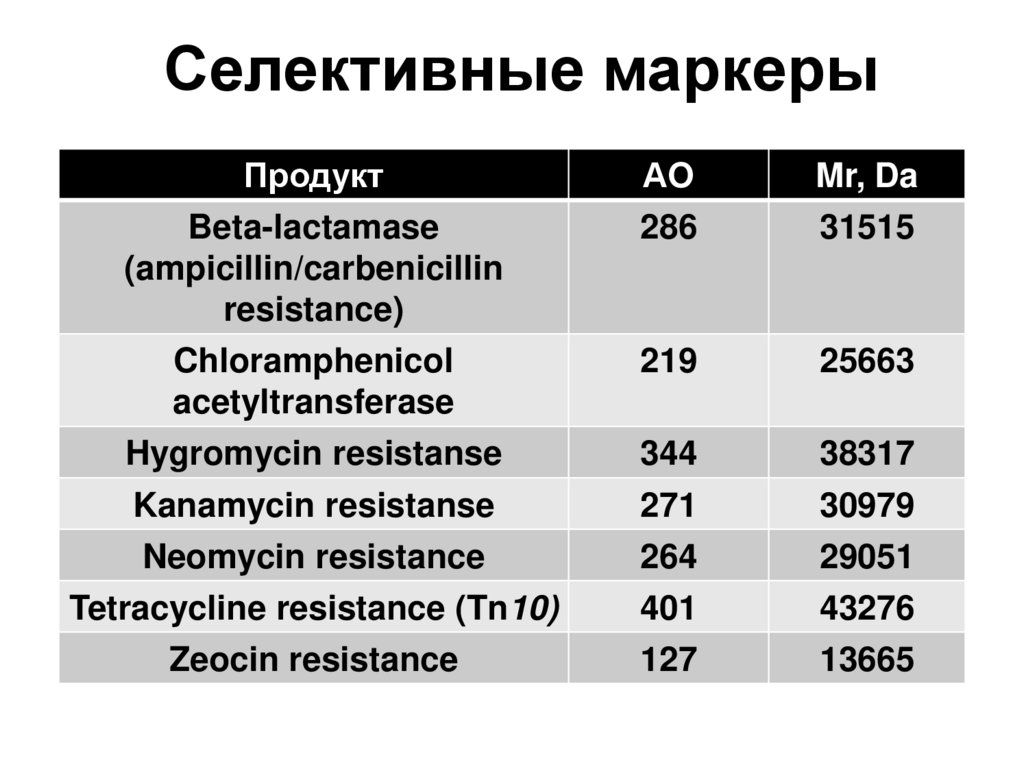
6. Ферменты, применяемые в генетической инженерии
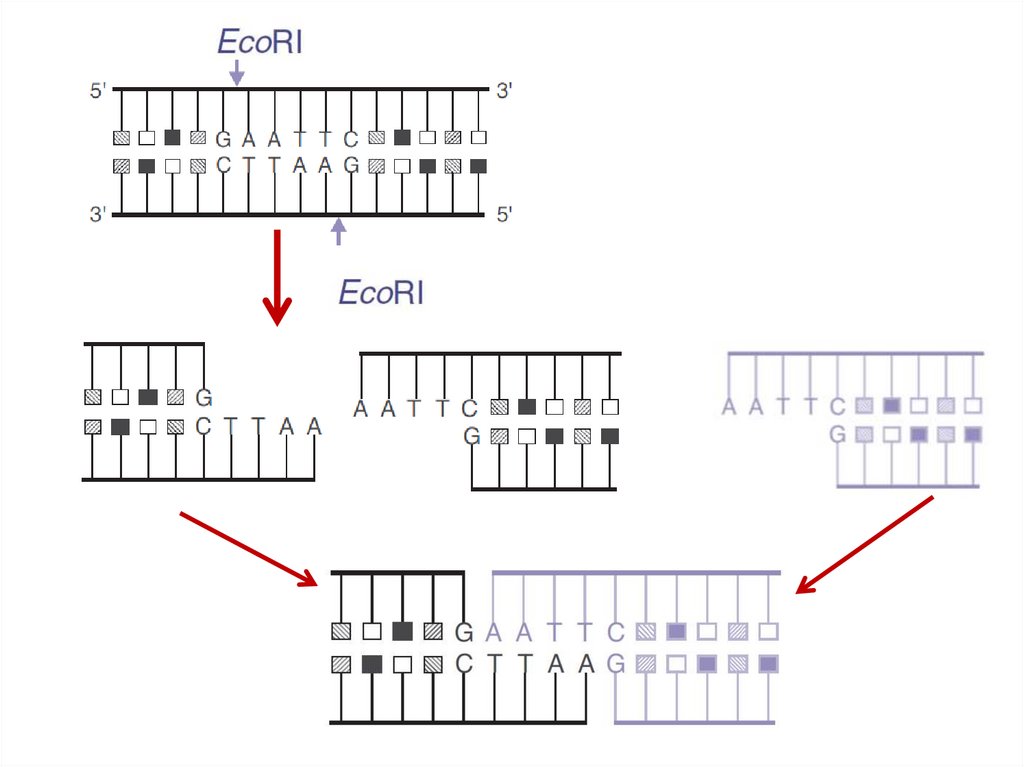
7. I. РЕСТРИКТАЗЫ (ЕС 3.1.21.) ЭНДОНУКЛЕАЗЫ РЕСТРИКЦИИ)
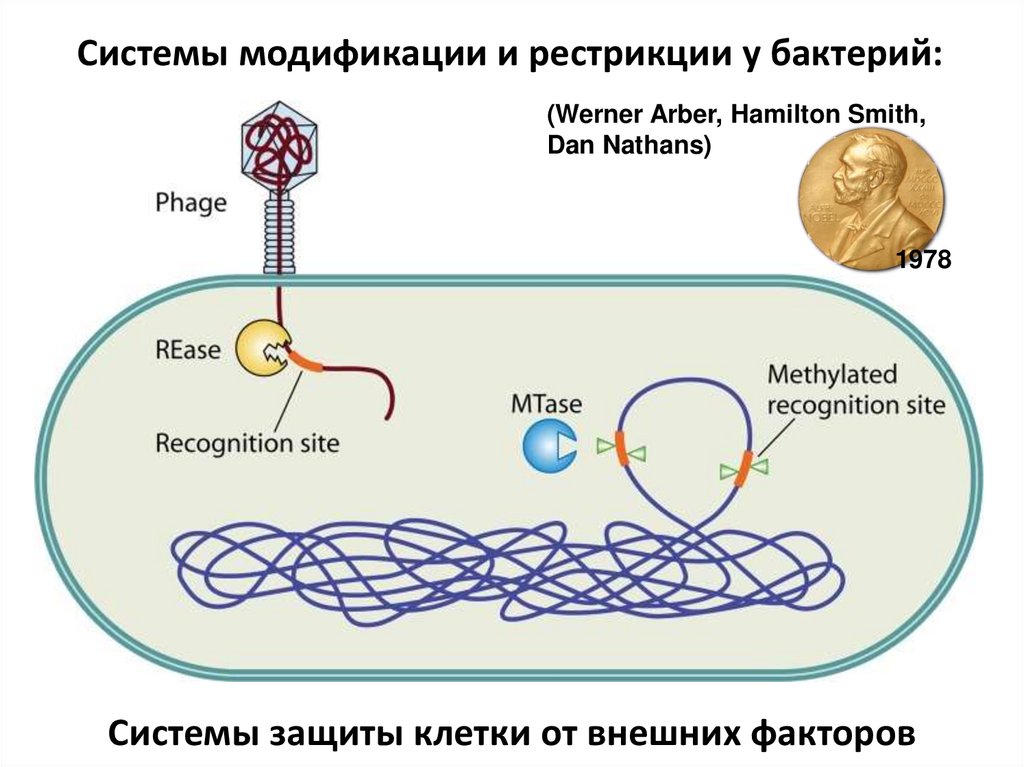
8.
Системы модификации и рестрикции у бактерий:(Werner Arber, Hamilton Smith,
Dan Nathans)
1978
Системы защиты клетки от внешних факторов
9.
Системы модификации и рестрикции у бактерий:Системы защиты клетки от внешних факторов
M. Мезельсон, Р. Юань выделили первые рестриктазы EcoK и EcoB, 1968 г.
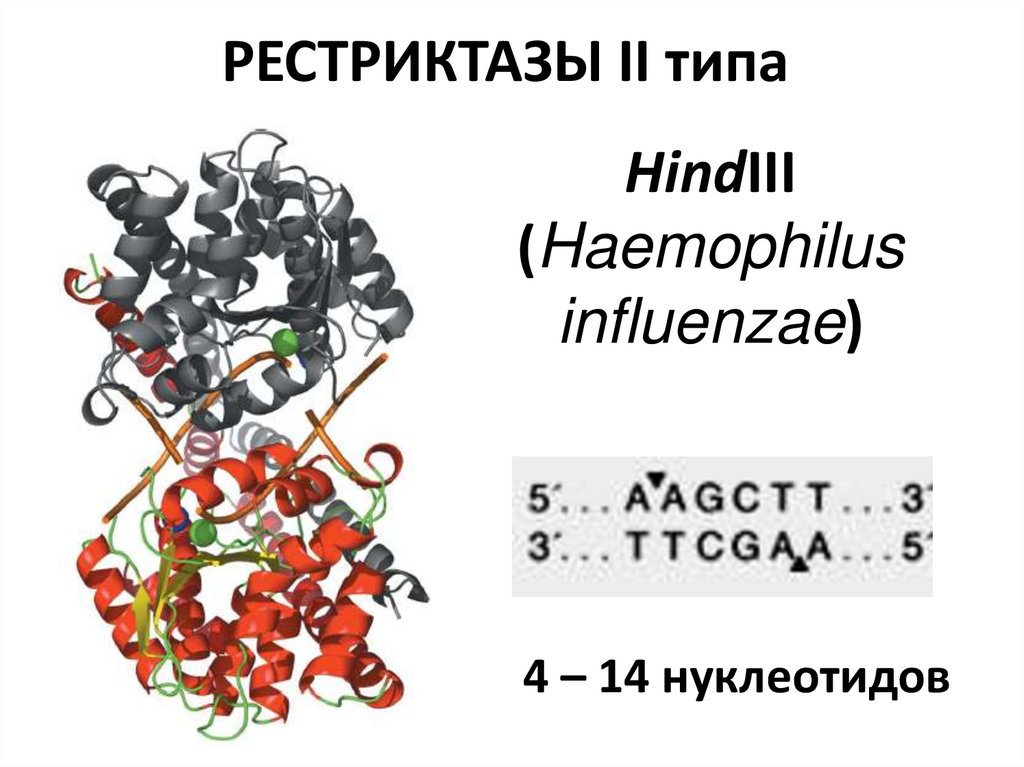
10. РЕСТРИКТАЗЫ II типа
HindIII(Haemophilus
influenzae)
4 – 14 нуклеотидов
11. ПАЛИНДРОМЫ
А РОЗА УПАЛА НА ЛАПУ АЗОРАПо обеим цепям ДНК в одном
направлении читаются одинаково
12. Рестриктазы в генной инженерии
EcoRI(E. coli RY13)
NdeI (Neisseria denitrificans)
BamHI (Bacillus amyloliquefaciens H)
BglII
(Bacillus globigii)
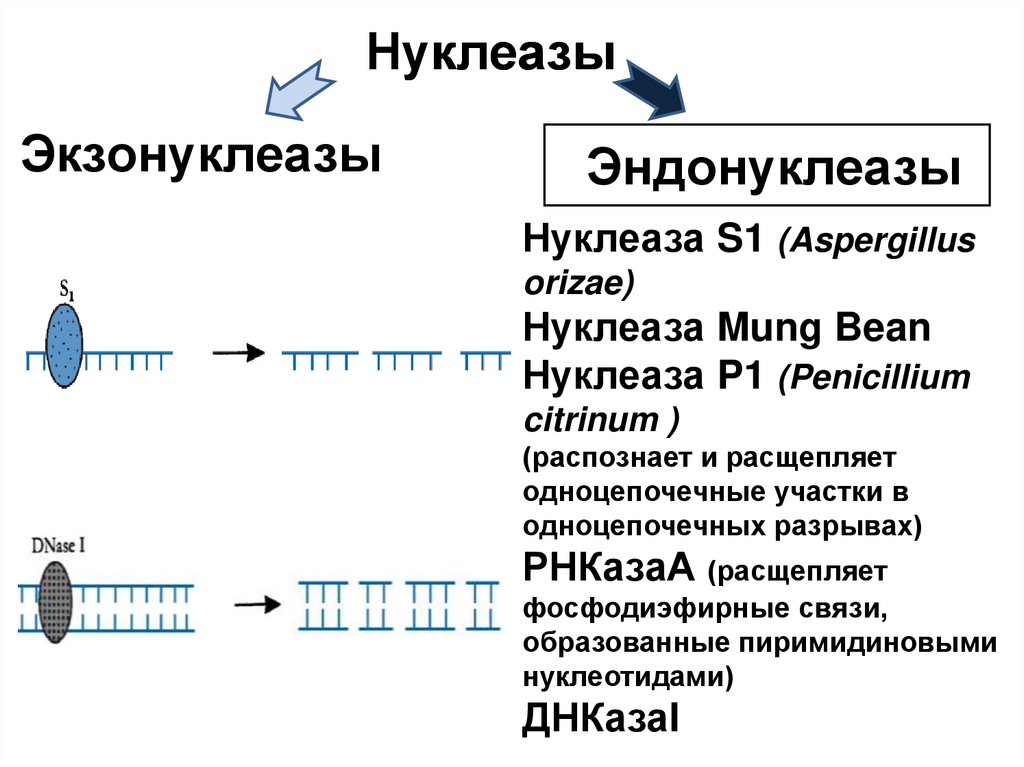
PmeI (Pseudomonas mendocina)
13. Классификация рестриктаз
ПалиндромЧастота встречаемости
4 нуклеотида
(мелкощепящие)
1:250
6 нуклеотидов (среднещепящие)
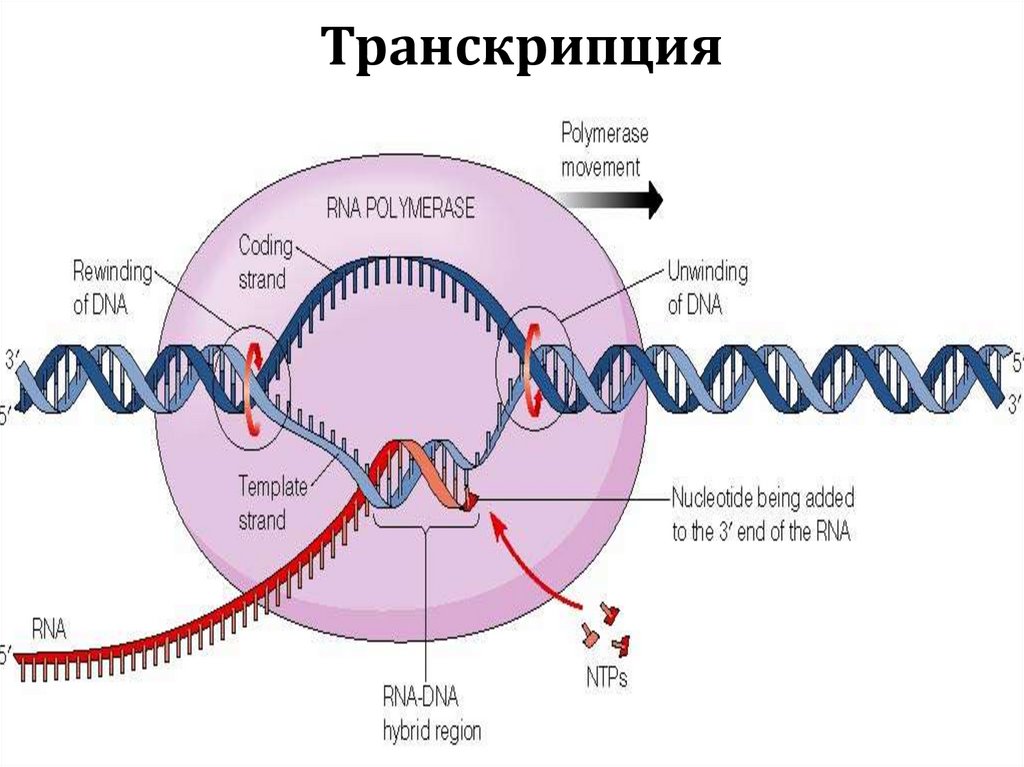
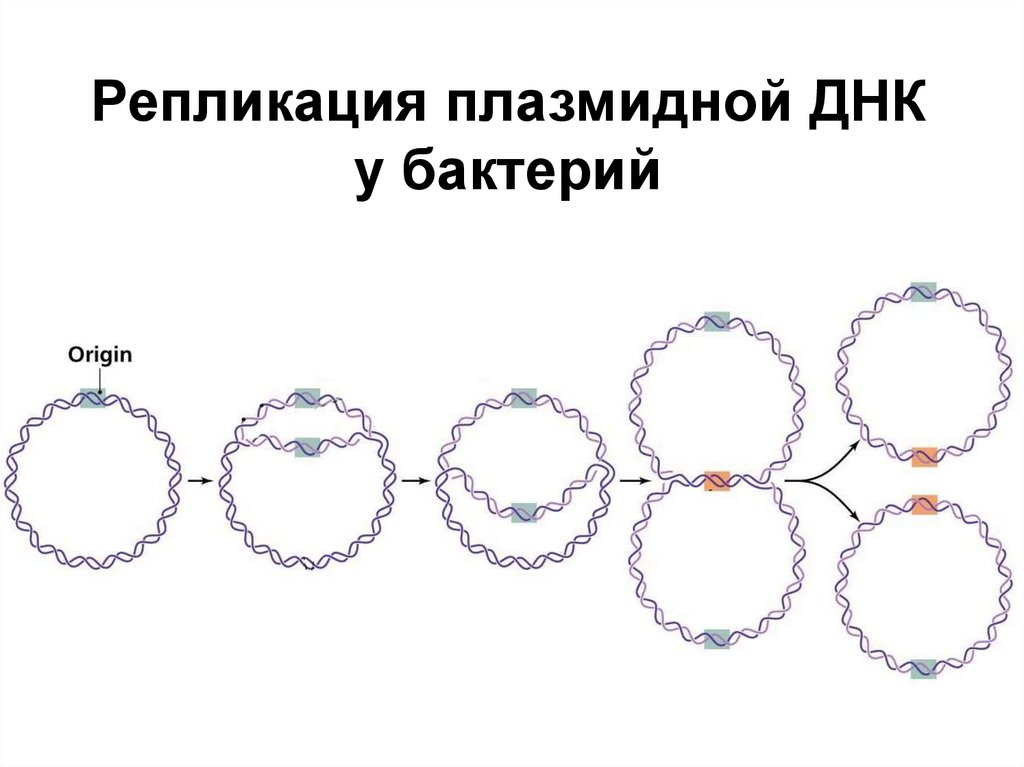
1:4000
8 нуклеотидов (среднещепящие)
1:64000
10 нуклеотидов ( крупнощепящие)
редко
14 нуклеотидов ( крупнощепящие)
редко
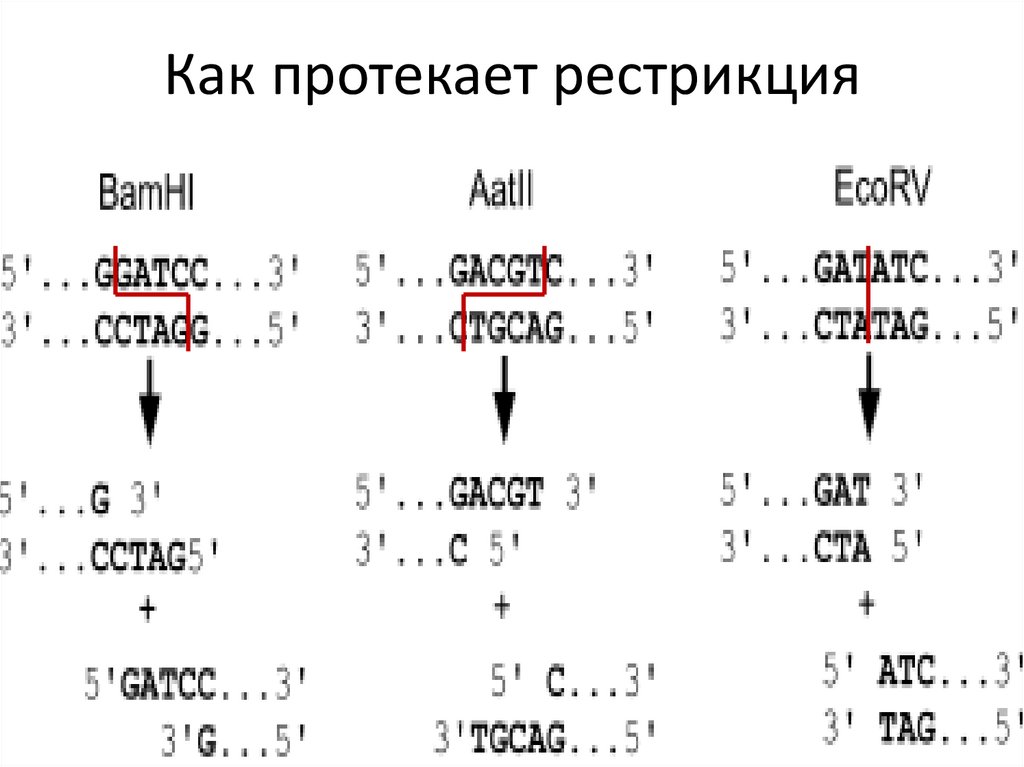
14. Как протекает рестрикция
15. Изошизомеры
PmeI (Pseudomonas mendocina)MssI (Methylobacterium species)
Неошизомеры
SmaI (Serratia marcescens)
XmaI (Xanthomonas malvacearum)
Изокаудомеры
BamHI (Bacillus amyloliquefaciens H)
BglII
(Bacillus globigii)
16. Активность рестриктаз
Единица активности рестриктазысоответствует количеству фермента необходимого
для полного расщепления 1 мкг ДНК фага-лямбда
при оптимальных условиях за 1 час
•Mg 2+ (обычно 10мМ)
•рН (для большинства от 7.0 до 8.0)
•Температура (370С, SmaI – 250C, TaqI – 650C)
•Ионная сила (50 – 100 мМ NaCl)
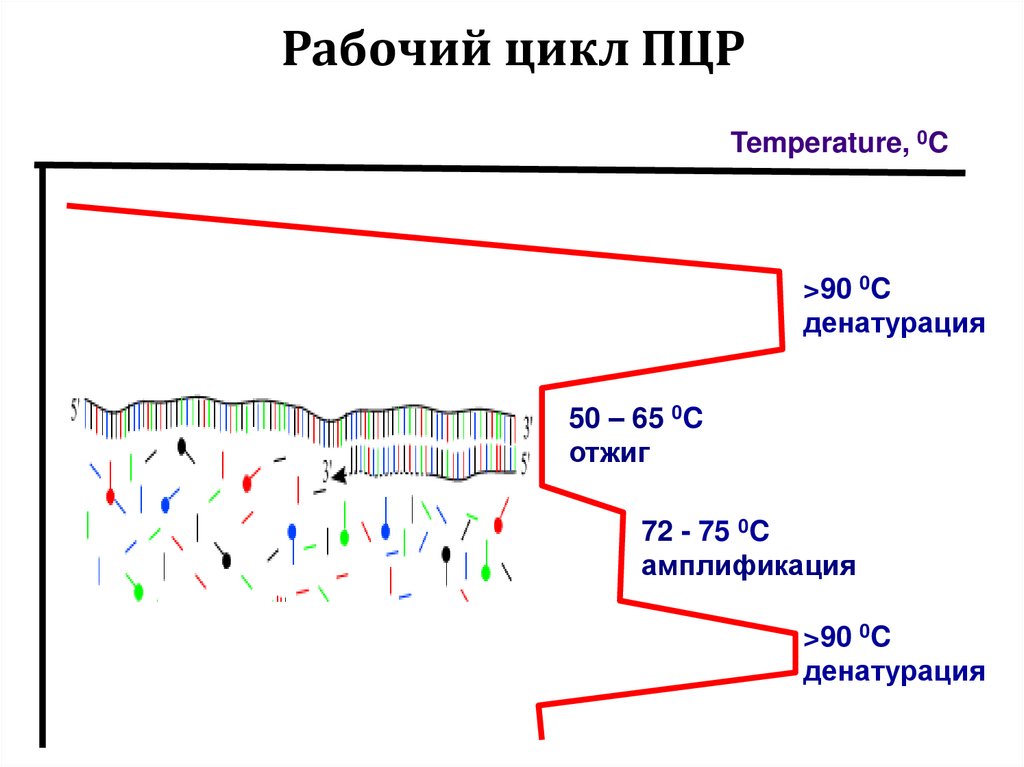
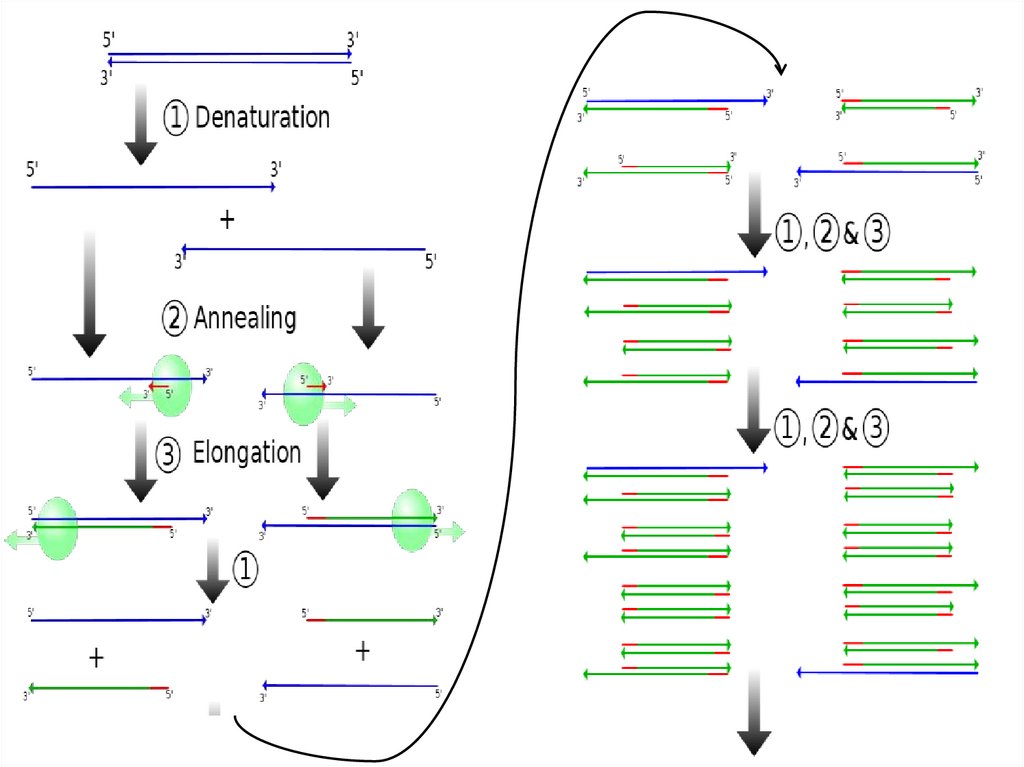
•BSA (bovine serum albumin, 100 мкг/мл)
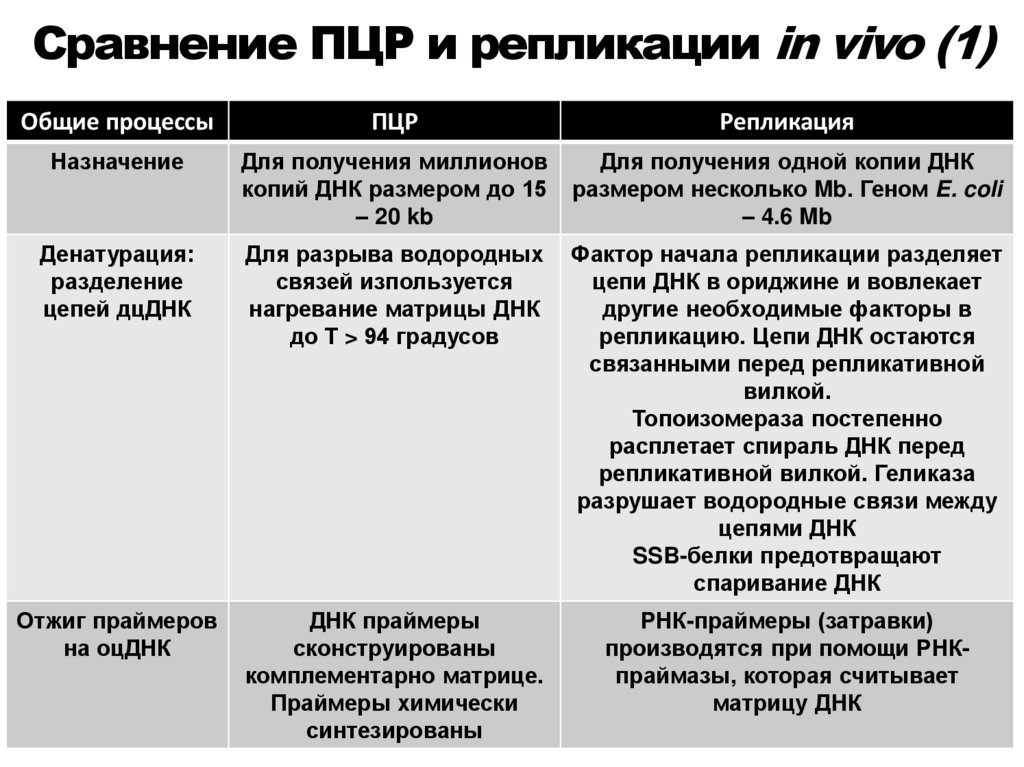
•Восстановители, прочие условия
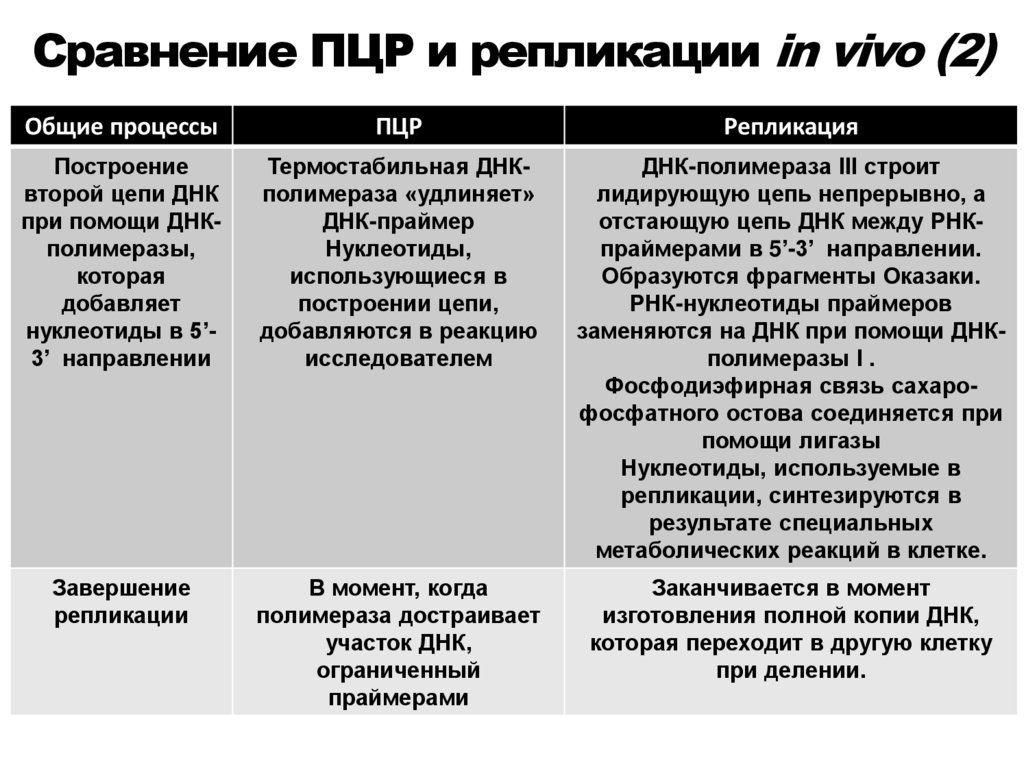
Звездная активность!!!
17.
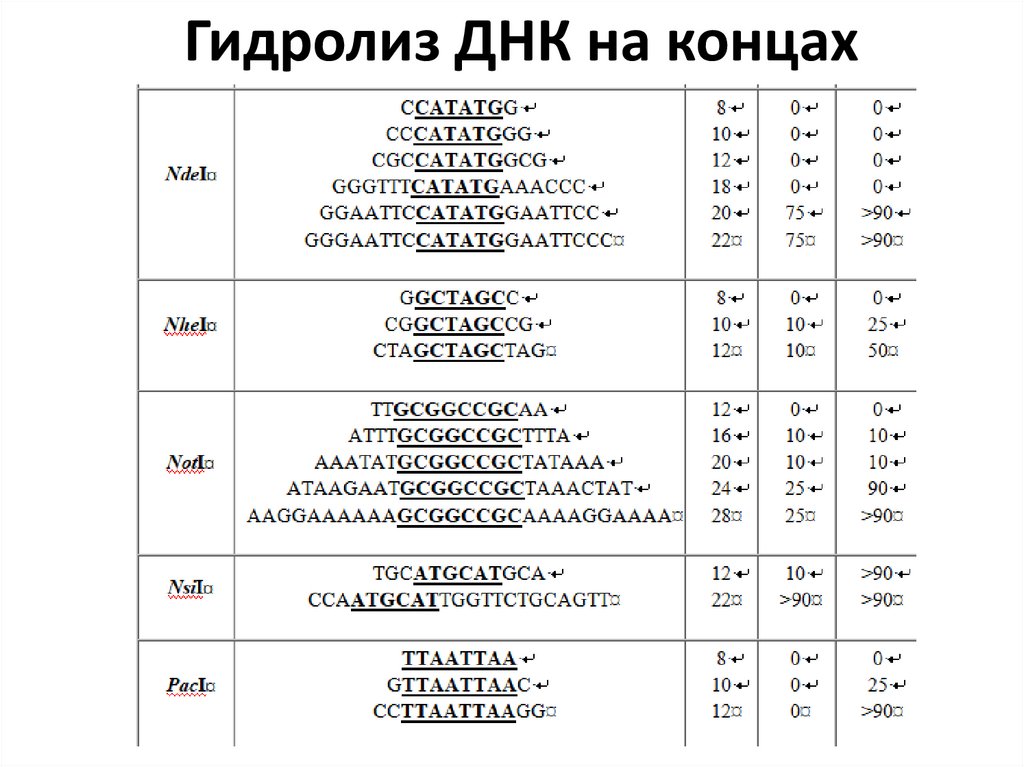
18. Гидролиз ДНК на концах
19.
Рестрикция ДНК!!!Концентрация образца ДНК может влиять на полноту расщепления. В вязких
сильно концентрированных растворах ДНК диффузия затруднена и это может
значительно уменьшить активность фермента. Рекомендуемые конечные
концентрации ДНК в реакции составляют 0.02-0.2 мкг/мкл.
1.Смешать: плазмидную ДНК (конечная концентрация до 0.1 мкг/мл),
1/10 объема 10-кратного рестрикционного буфера, рекомендуемого
фирмой-поставщиком
не более 1/10 объема фермента!!!
2. Довести объем реакции до 10 мкл, если это аналитическое расщепление, или до
желаемого объема, в случае препаративного расщепления, очищенной водой.
3. Инкубировать при 37°С в течение 1 ч.
Типичная рестрикция
Смешать: 7.9 мкл очищенной воды
1 мкл 10 буфера
1 мкл (0.2 мкг) плазмидной ДНК
0.1 мкл (10 ед/мкл) HindIII.
!!!Компоненты следует добавлять в той последовательности, как они даны в
прописи.
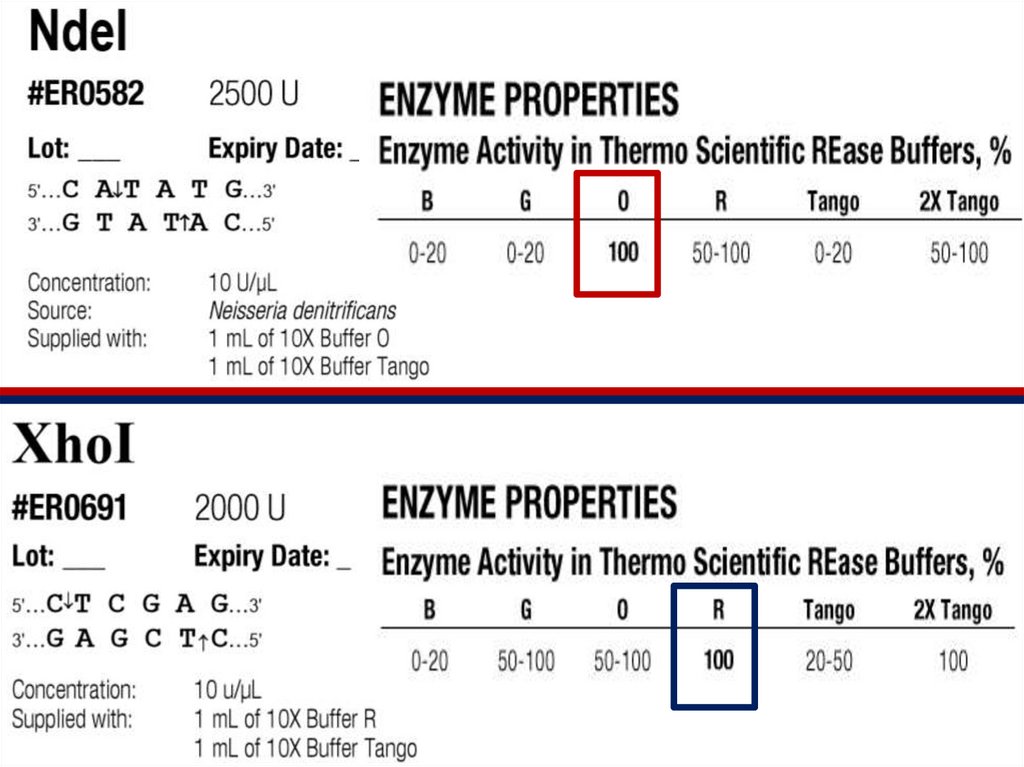
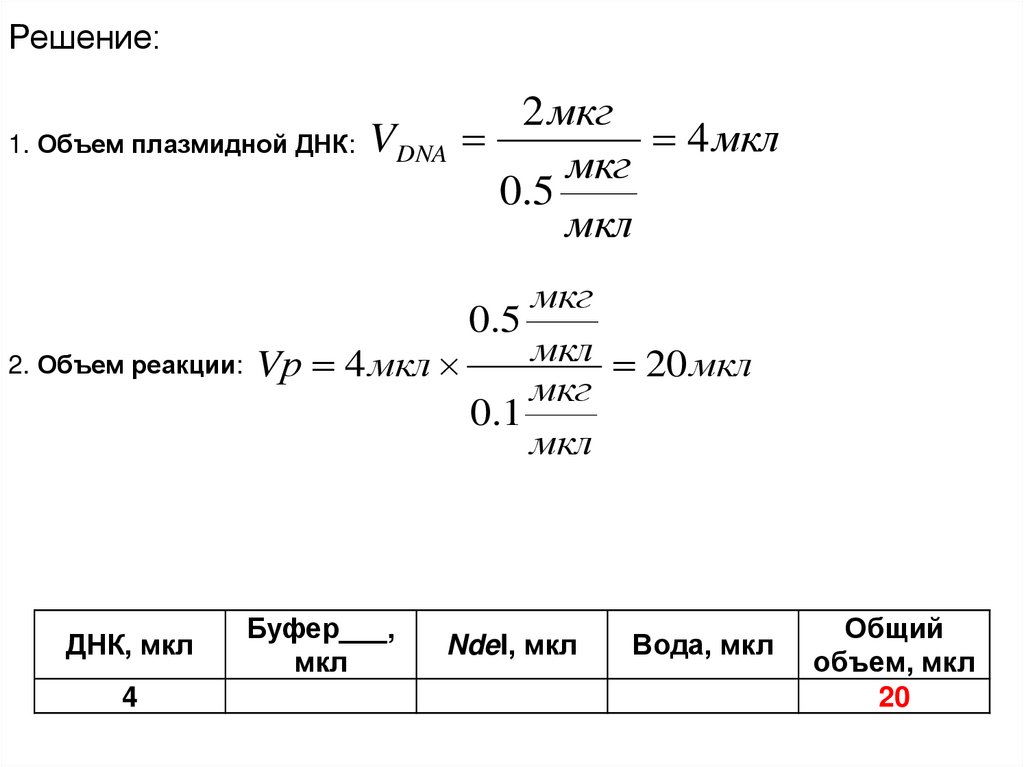
20. Пример №1
Рассчитайте количество фермента NdeI, требуемого длягидролиза 2 мкг плазмидной ДНК. Составьте пропись и
подберите условия для проведения реакции рестрикции.
Исходные данные:
1. Раствор плазмидной ДНК. Концентрация 0,5 мкг/мкл
2. Рестриктаза NdeI (производство NEB). Концентрация
фермента 20000 единиц/мл. Эффективность работы в
буферных растворах производителя: Буфер 1 – 75%, Буфер 2
– 100%, Буфер 3 – 100%, Буфер 4 – 10%. Буферные растворы
поставляются в 10-тикратной концентрации!!!
21.
Решение:2 мкг
4 мкл
1. Объем плазмидной ДНК: VDNA
мкг
0 .5
мкл
мкг
0.5
мкл 20 мкл
2. Объем реакции: Vр 4 мкл
мкг
0.1
мкл
ДНК, мкл
4
Буфер___,
мкл
NdeI, мкл
Вода, мкл
Общий
объем, мкл
20
22.
Расчет количества фермента:По определению активности эндонуклеазы рестрикции:
1 единица фермента гидролизует 1 мкг фага лямбда
5 единиц фермента гидролизует 1 мкг плазмиды, тогда:
2 мкг
EU NdeI 5ед
10ед
1мкг
Согласно инструкции производителя фермента:
В 1 мл ферментного препарата содержится 20000 единиц, тогда:
10ед
VNdeI 1000 мкл
0.5 мкл
20000ед
Следовательно, минимальный объем реакции – 5 мкл (по ферменту):
ДНК, мкл
4
Буфер___,
мкл
NdeI, мкл
0,5
Вода, мкл
Общий
объем, мкл
20
23.
Расчет количества буферного раствора:Согласно инструкции производителя фермент имеет
максимальную активность в буферных растворах №2 и №3.
Выбираем любой из них. Количество десятикратного
концентрата рассчитываем следующим образом:
20 мкл
VБуфер .
2 мкл
10
Расчет количества воды:
Vводы Vобщ Vбуф. Vферм V ДНК 20 2 0,5 4 13,5 мкл
ДНК, мкл
4
Буфер 2 (3),
мкл
2
NdeI, мкл
Вода, мкл
0,5
13,5
Общий
объем, мкл
20
Условия проведения реакции: температура 370С, время – 1 час
24. Задание № 2
Рассчитайте количество ферментов NdeI и XhoI, требуемогодля гидролиза 3 мкг плазмидной ДНК с целью выделения
фрагмента. Составьте пропись и подберите условия для
проведения реакции рестрикции.
Исходные данные:
1. Раствор плазмидной ДНК. Концентрация 0,3 мкг/мкл
2. Рестриктаза NdeI (производство NEB). Концентрация
фермента 20000 единиц/мл. Эффективность работы в
буферных растворах производителя: Буфер 1 – 75%, Буфер
2 – 100%, Буфер 3 – 100%, Буфер 4 – 10%.
3. Рестриктаза XhoI (производство NEB). Концентрация
фермента 20000 единиц/мл. Эффективность работы в
буферных растворах производителя: Буфер 1 – 75%, Буфер
2 – 10%, Буфер 3 – 50%, Буфер 4 – 100%. Буферные
растворы поставляются в 10-тикратной концентрации!!!
25. Совместный гидролиз ДНК
Буфер1
2
3
4
NdeI
XhoI
75%
75%
100%
10%
100%
50%
10%
100%
Выбирается наиболее удачный вариант
для одновременной работы двух
рестриктаз!!!
26.
мкг0.3
мкл 30 мкл
Vр 10 мкл
мкг
0.1
мкл
3 мкг
VDNA
10 мкл
мкг
0 .3
мкл
EU NdeI ( XhoI ) 5ед
3 мкг
15ед
1мкг
VБуфер .
VNdeI ( XhoI ) 1000 мкл
15ед
0.75 мкл
20000ед
30 мкл
3 мкл
10
Vводы Vобщ Vбуф. VNdeI VXhoI V ДНК 30 3 0.75 0.75 10 15,5 мкл
ДНК, мкл Буфер 1, мкл
10
3
NdeI, мкл
XhoI, мкл
Вода, мкл
Общий
объем, мкл
0.75
0.75
15.5
30
Условия проведения реакции: температура 370С, время – 1.5 часа
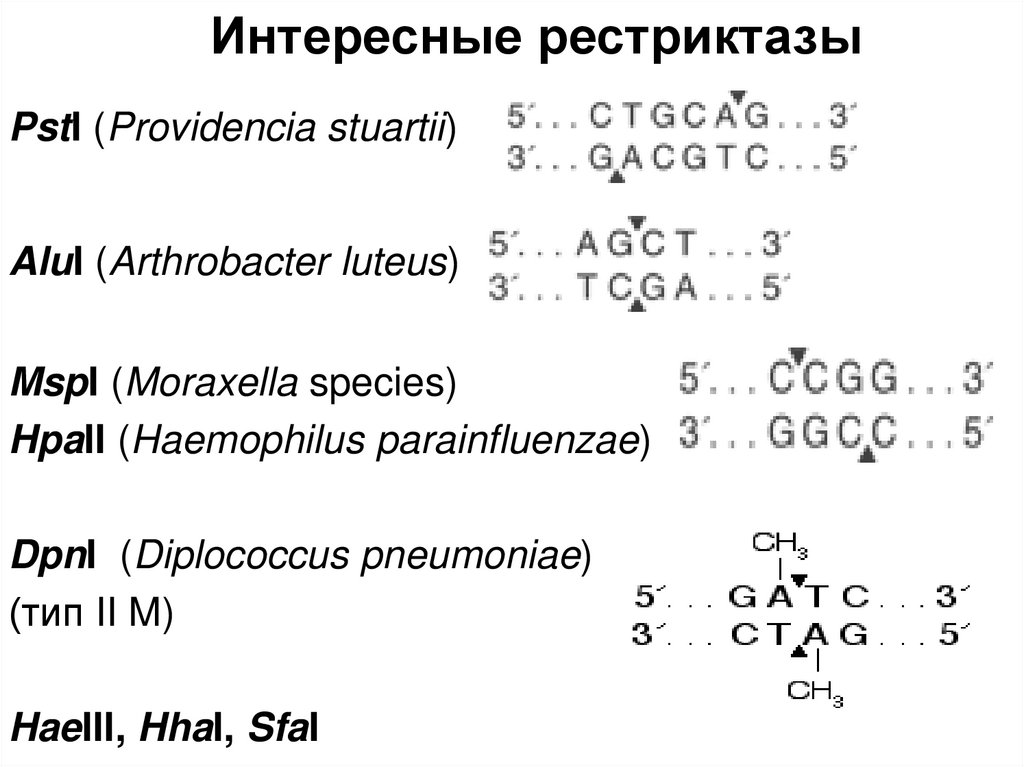
27. Интересные рестриктазы
PstI (Providencia stuartii)AluI (Arthrobacter luteus)
MspI (Moraxella species)
HpaII (Haemophilus parainfluenzae)
DpnI (Diplococcus pneumoniae)
(тип II M)
HaeIII, HhaI, SfaI
28.
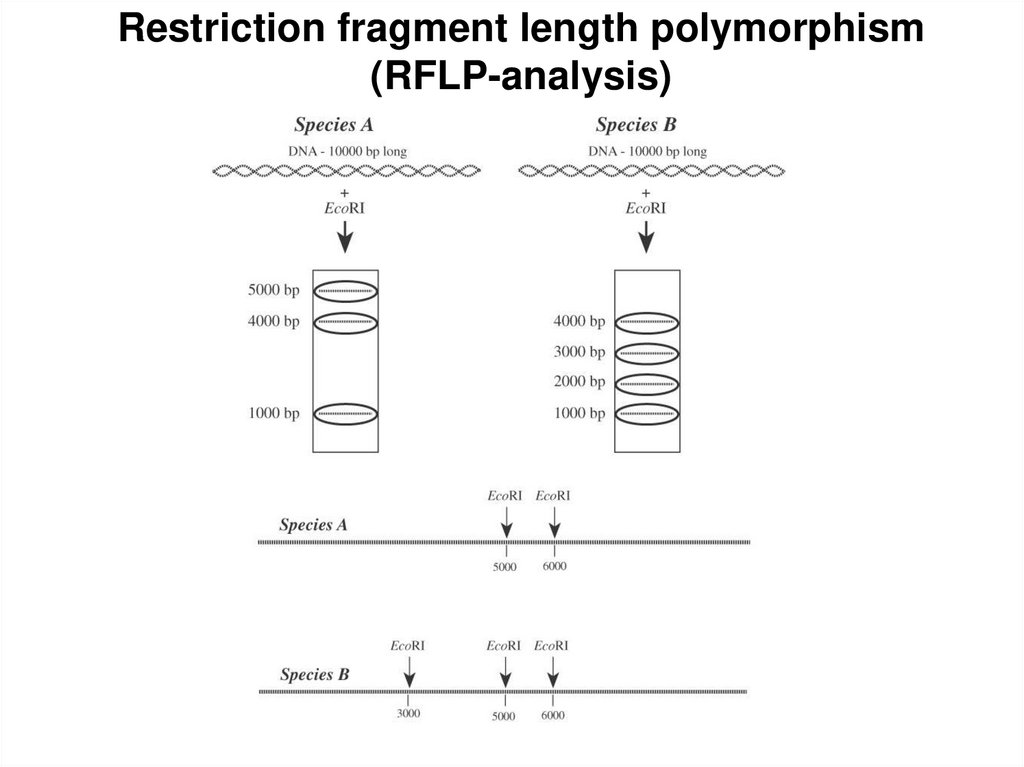
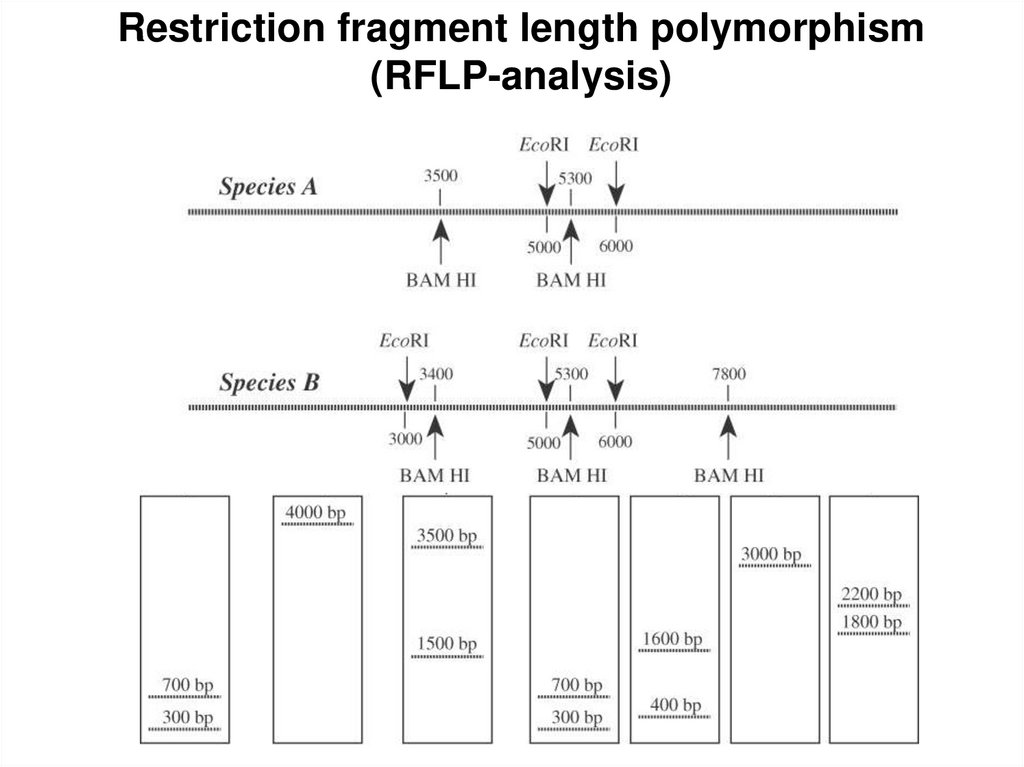
Restriction fragment length polymorphism(RFLP-analysis)
29.
Restriction fragment length polymorphism(RFLP-analysis)
30.
«Молекулярный» отпечаток пальцев(Molecular fingerprint)
31.
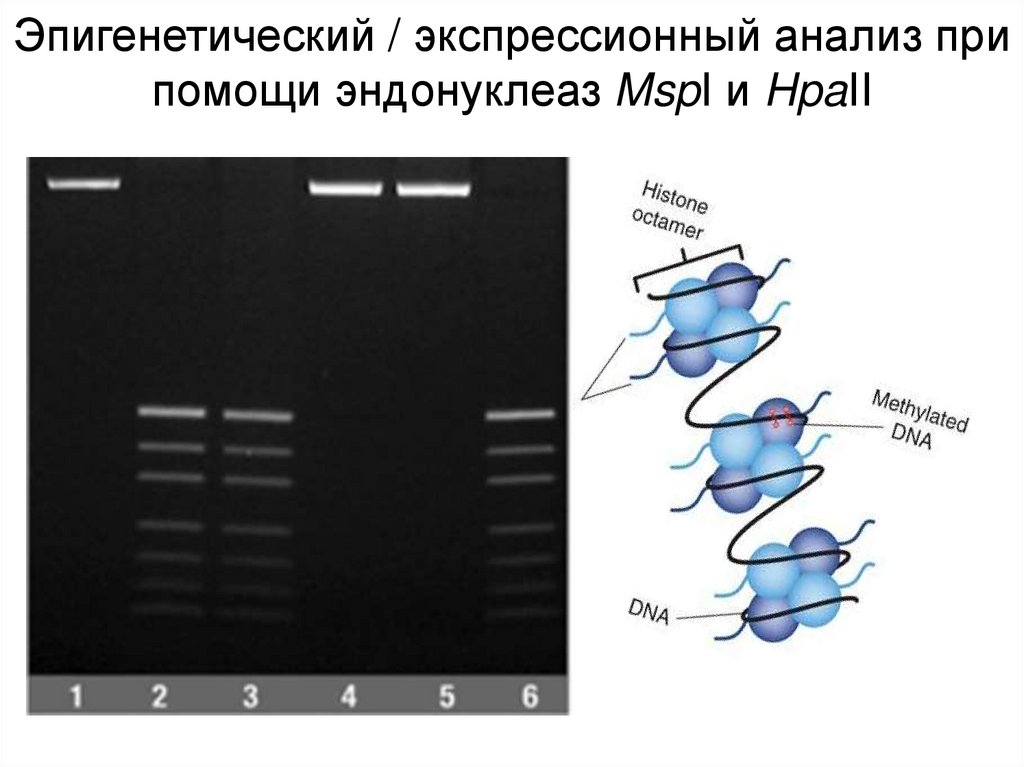
Эпигенетический / экспрессионный анализ припомощи эндонуклеаз MspI и HpaII
32.
33.
II. ЛИГАЗЫ (EC 6.5.1.1)34.
История открытия ДНК-лигазНеобходимость фермента, катализирующего сшивание
сахарофосфатного остова доказано при открытие механизмов
1961рекомбинации и репарации, в ходе которых исправляются
1965
ошибки, возникшие при повреждениях ДНК (Meselson and
Weigle, 1961)
1967
Показано, что лизат E. coli может «сшивать» сахарофосфатный
остов репликативной формы фага-лямбда (псевдокольцо) в
истинную кольцевую молекулу (Marty Gellert)
1967
Была выделена ДНК-лигаза, фермент, который катализирует
образование фосфодиэфирной связи между 5’-фосфатной и 3’гидрокси- группами
1968
Okazaki обнаружил важную роль лигаз в репликации ДНК
1979
Клонирована T4-ДНК лигаза (Wilson and Murray)
1983
Исследована пространственная структура T4-ДНК лигазы
(Armstrong)
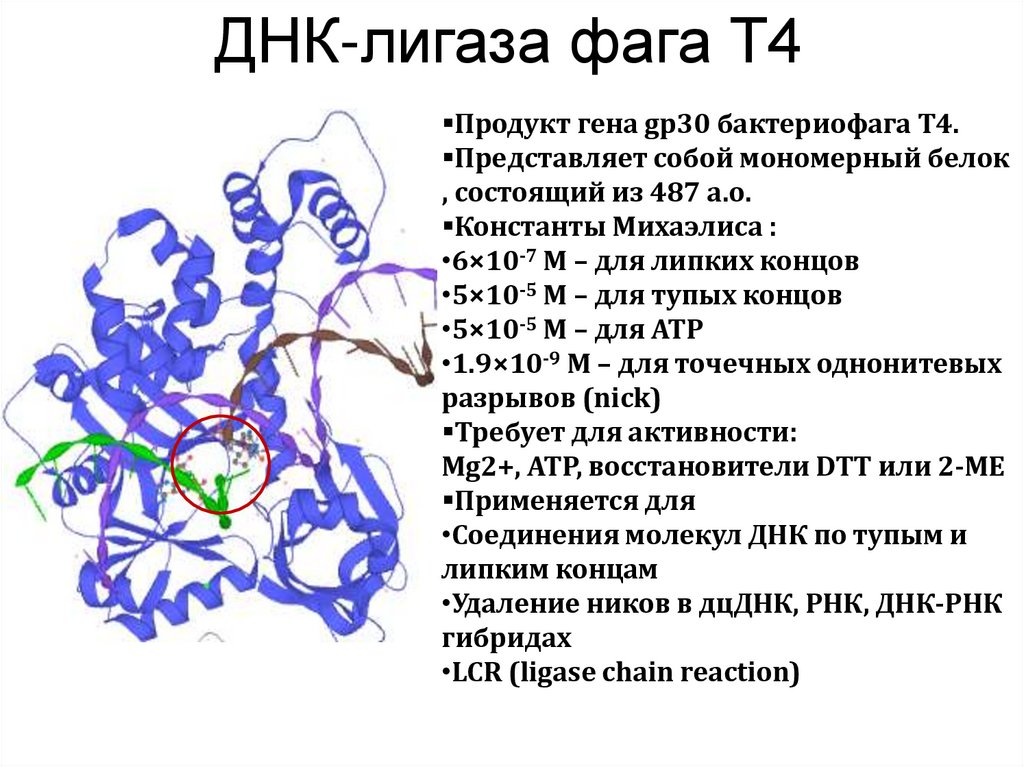
35. ДНК-лигаза фага Т4
Продукт гена gp30 бактериофага T4.Представляет собой мономерный белок
, состоящий из 487 а.о.
Константы Михаэлиса :
•6×10-7 М – для липких концов
•5×10-5 М – для тупых концов
•5×10-5 М – для ATP
•1.9×10-9 М – для точечных однонитевых
разрывов (nick)
Требует для активности:
Mg2+, ATP, восстановители DTT или 2-ME
Применяется для
•Соединения молекул ДНК по тупым и
липким концам
•Удаление ников в дцДНК, РНК, ДНК-РНК
гибридах
•LCR (ligase chain reaction)
36. Механизм действия
1. Образование ковалентного комплекса «Лигаза-АМФ»2. Перенос АМФ на 5’-фосфатную группу молекулы ДНК
3. Нуклеофильная атака 3’-гидроксигруппы связи ДНК с АМФ
37. Активность ДНК-лигазы фага Т4
Единица активности лигазы соответствуетколичеству фермента , которое необходимо
для переноса 1 нмоль 32P от неорганического
пирофосфата PPi до ATP за 20 минут при
370С(Weiss, 1968)
Единица активности лигазы соответствует
количеству фермента, необходимого для
50%-ного лигирования 5 мкг фрагментов
ДНК фага λ (300 мкг/мл), полученных
рестрикцией ферментом HindIII, за 30 минут
при температуре 160С, в реакционном
объеме 20 мкл («cohesive unit», NEB)
1CU ~ 0.015 WU 1WU ~ 60 WU
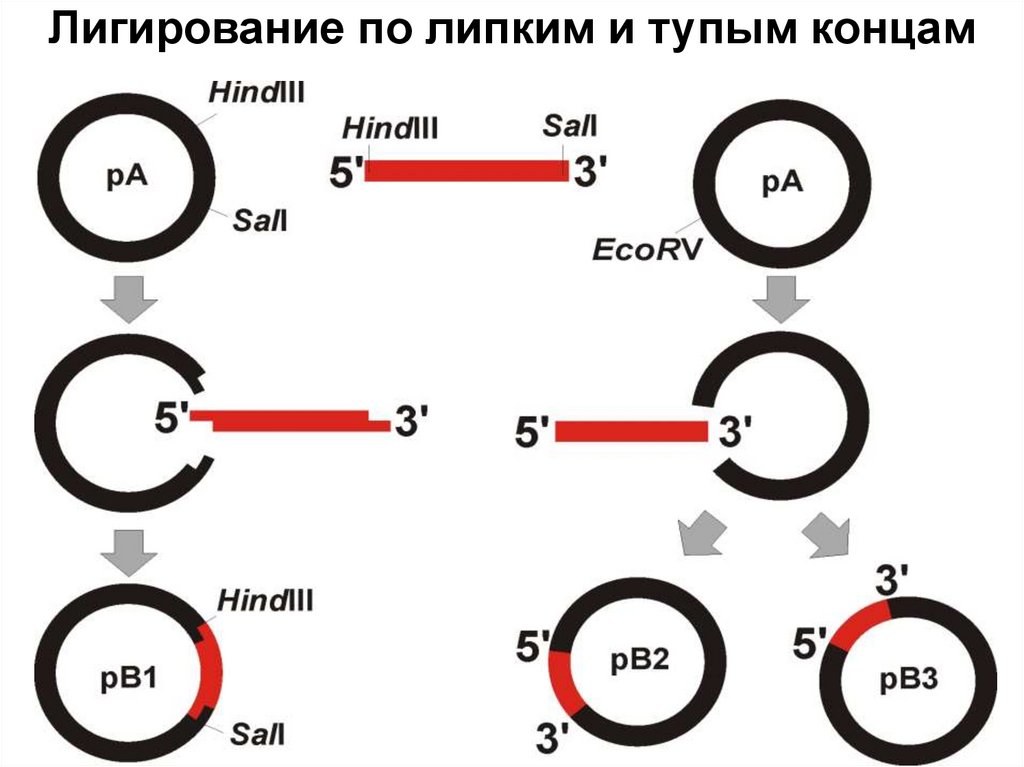
38. Лигирование по липким и тупым концам
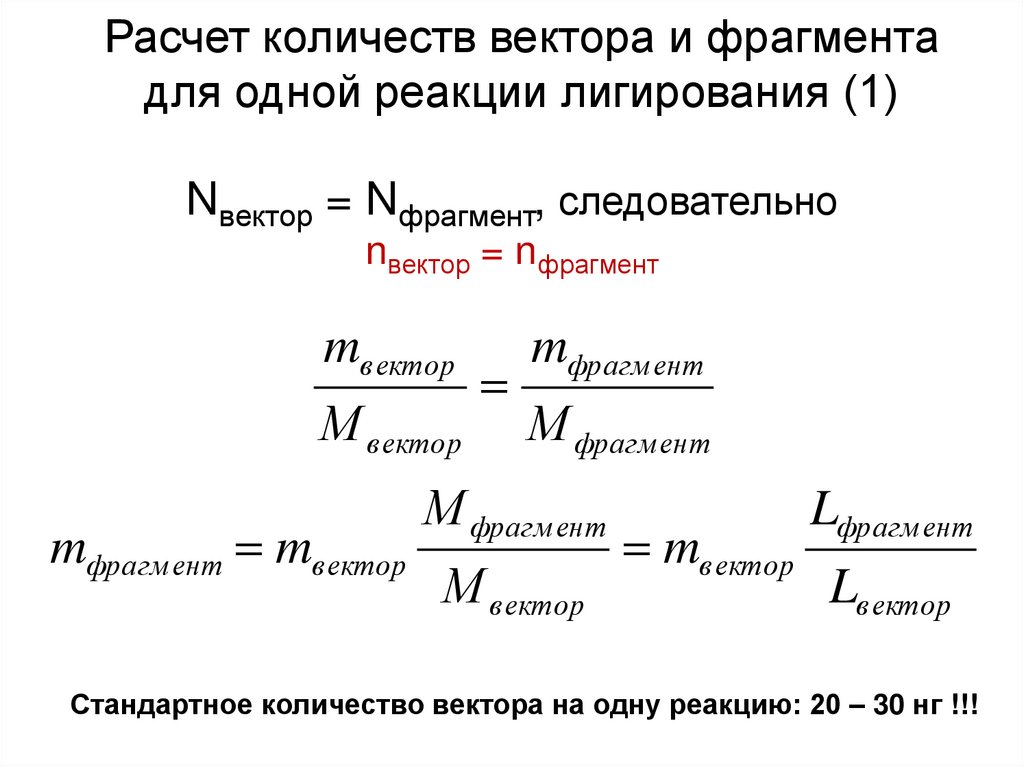
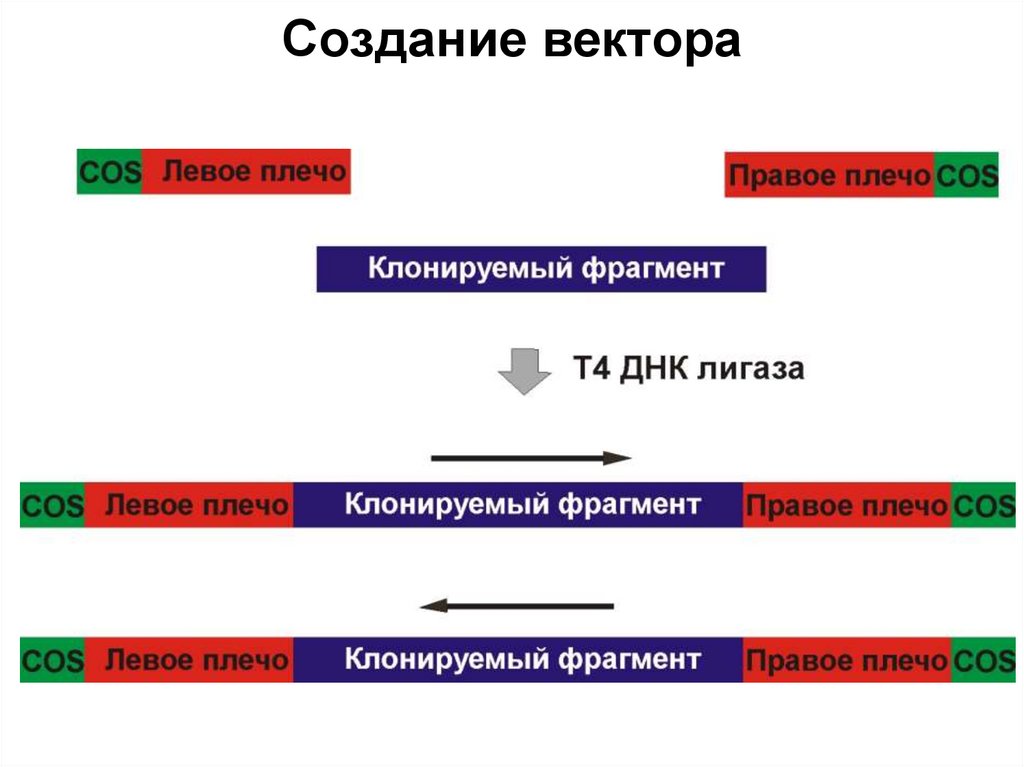
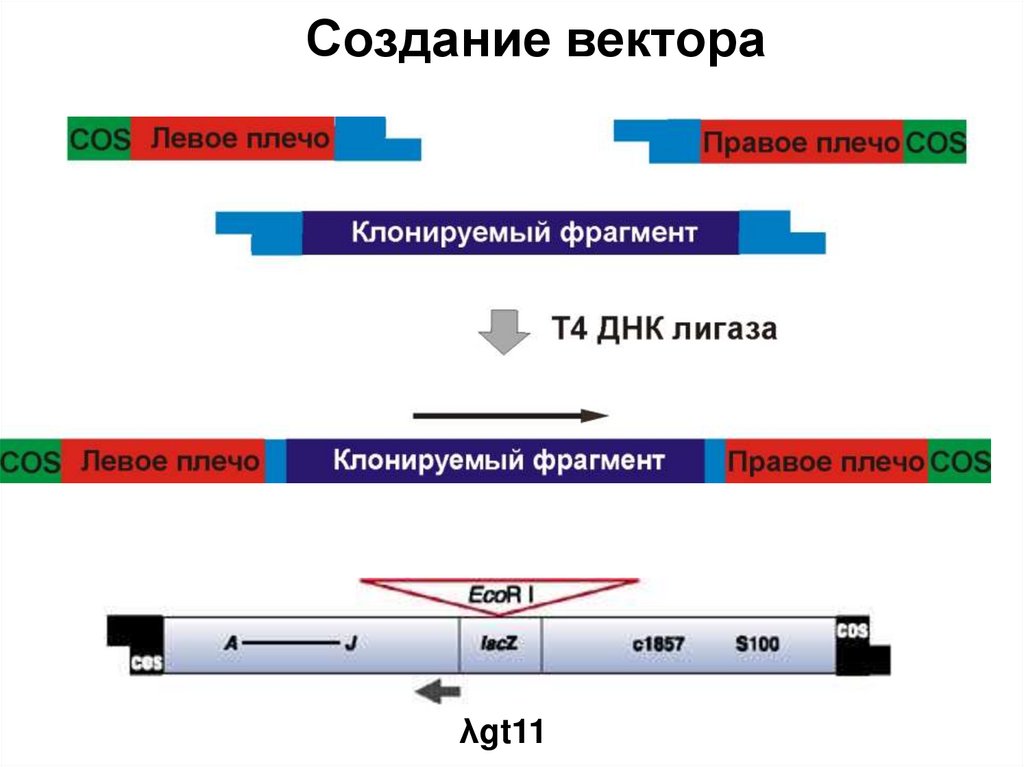
39. Расчет количеств вектора и фрагмента для одной реакции лигирования (1)
Nвектор = Nфрагмент, следовательноnвектор = nфрагмент
mв ектор
М в ектор
mфрагм ент mв ектор
mфрагм ент
М фрагм ент
М фрагм ент
М в ектор
mв ектор
Lфрагм ент
Lв ектор
Стандартное количество вектора на одну реакцию: 20 – 30 нг !!!
40.
Расчет количеств вектора и фрагментадля одной реакции лигирования (2)
Формула для лигирования по липким концам:
mфрагм ент ( 2 5) mв ектор
Lфрагм ент
Lв ектор
Условия лигирования: 0.5 – 2 часа при комнатной температуре
Формула для лигирования по тупым концам:
mфрагм ент (5 10) mв ектор
Lфрагм ент
Lв ектор
Условия лигирования: 0.5 – 2 часа при комнатной температуре или на
ночь при +100С!!!
41.
Лигирование ДНК1.Смешать:
20 нг фрагмента вектора,
N нг клонируемого фрагмента,
1 мкл лигазного буфера (рибоАТФ),
1 мкл лигазы
очищенной воды до 10 мкл.
2. Инкубировать при комнатной температуре в течение от 1 часа до
ночи.
Если в реакции лигирования используется дефосфорилированный векторный
фрагмент, то молярное соотношение вектор/фрагмент должно составлять 2:1.
Во всех остальных случаях – 1:2 – 1:10.
Особенности:
1. Т4 ДНК лигаза сильно ингибируется NaCl или KCl, если их
концентрация превышает 200 мМ.
2. Полиэтиленгликоль (PEG) значительно ускоряет скорость
лигирования ДНК с тупыми концами. Конечная концентрация PEG4000 в
реакционной смеси должна составлять 5% (w/v) (B.H.Pheiffer,
S.B.Zimmerman,1983, Nucleic Acids Res., 11, 7853-7871).
3.Следует проводить инактивацию Т4 ДНК лигазы перед
трансформацией. Прогрев в течение 10 мин при 65°С может увеличить
количество трансформантов, в некоторых случаях, на два порядка
(B.K.Michelsen,1995, Anal. Biochem., 225, 172-174)
42.
Проверка активности лигазы1.Смешать:
5 мкл воды
2 мкл лигазного буфера (х 10)
12 мкл ДНК фага , расщепленной HindIII (0.5 мкг/мкл)
1 мкл лигазы
Компоненты следует добавлять в той последовательности, как они
даны в прописи. Лигазу следует разбавлять в 1-кратном
лигазном буфере непосредственно перед опытом и хранить в ледяной
бане.
2. Инкубировать при 16°С и отбирать пробы по 5 мкл через 0, 15, 30 и 60
мин.
3. Смешать образец с краской для электрофореза и прогреть 15 мин при
65°С.
4.Проанализировать при помощи электрофореза в 1% агарозном геле.
За 30 мин ДНК должна полностью сшиться!
43.
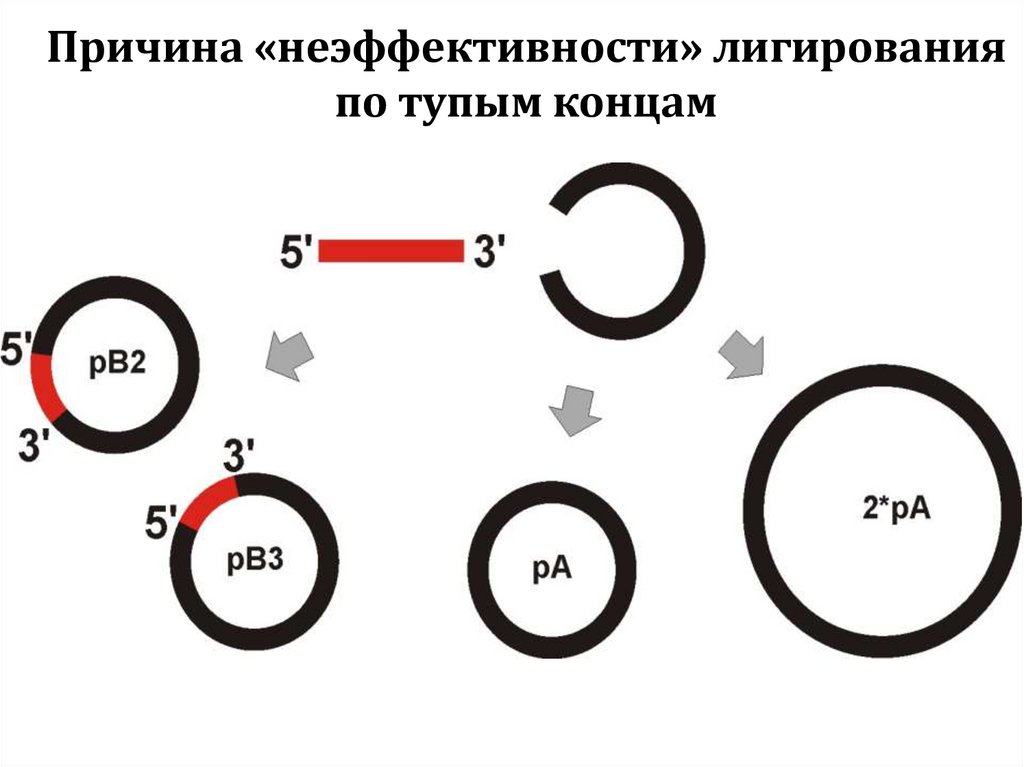
Причина «неэффективности» лигированияпо тупым концам
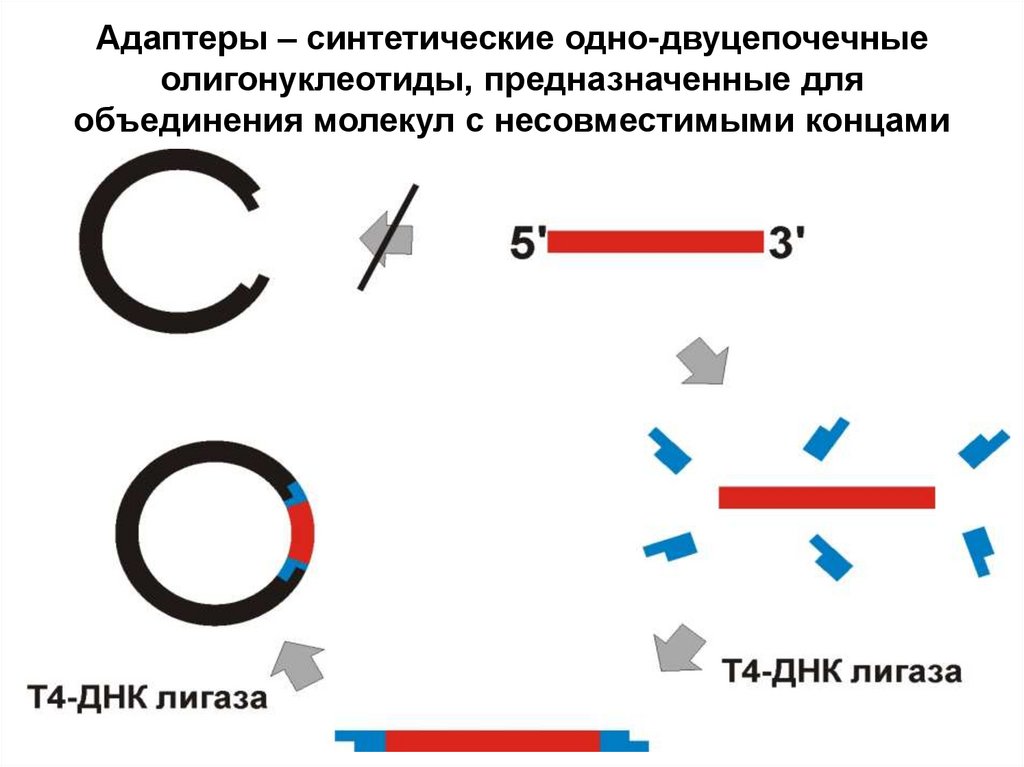
44. Адаптеры – синтетические одно-двуцепочечные олигонуклеотиды, предназначенные для объединения молекул с несовместимыми концами
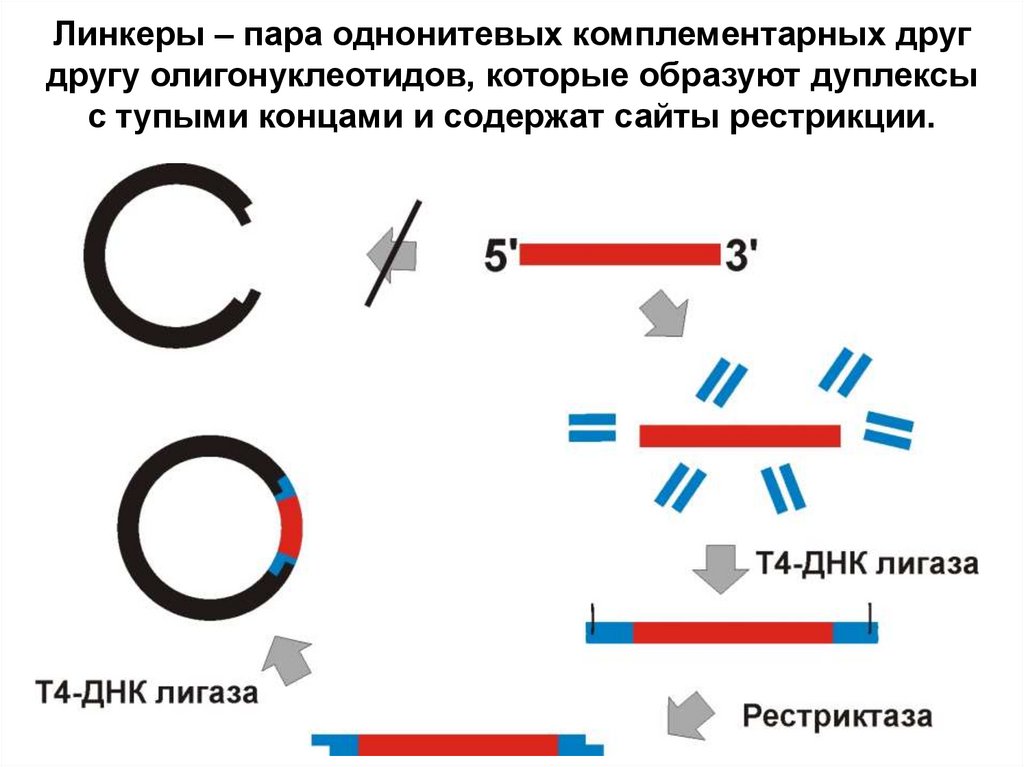
45. Линкеры – пара однонитевых комплементарных друг другу олигонуклеотидов, которые образуют дуплексы с тупыми концами и содержат
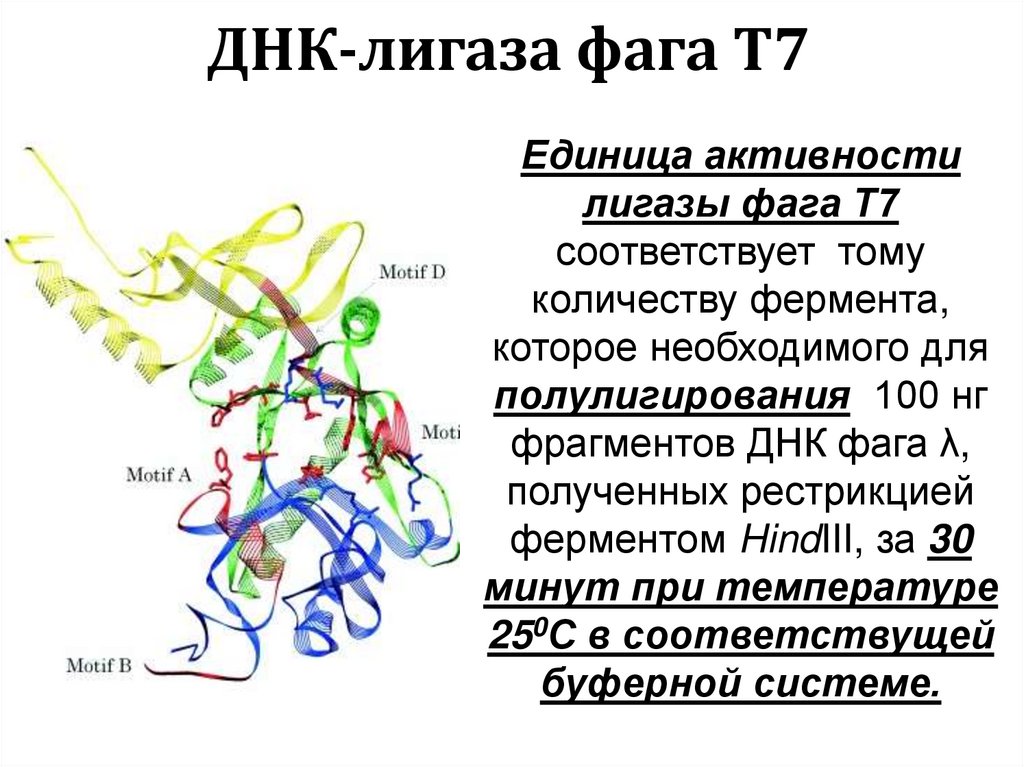
сайты рестрикции.46. ДНК-лигаза фага Т7
Единица активностилигазы фага Т7
соответствует тому
количеству фермента,
которое необходимого для
полулигирования 100 нг
фрагментов ДНК фага λ,
полученных рестрикцией
ферментом HindIII, за 30
минут при температуре
250С в соответствущей
буферной системе.
47.
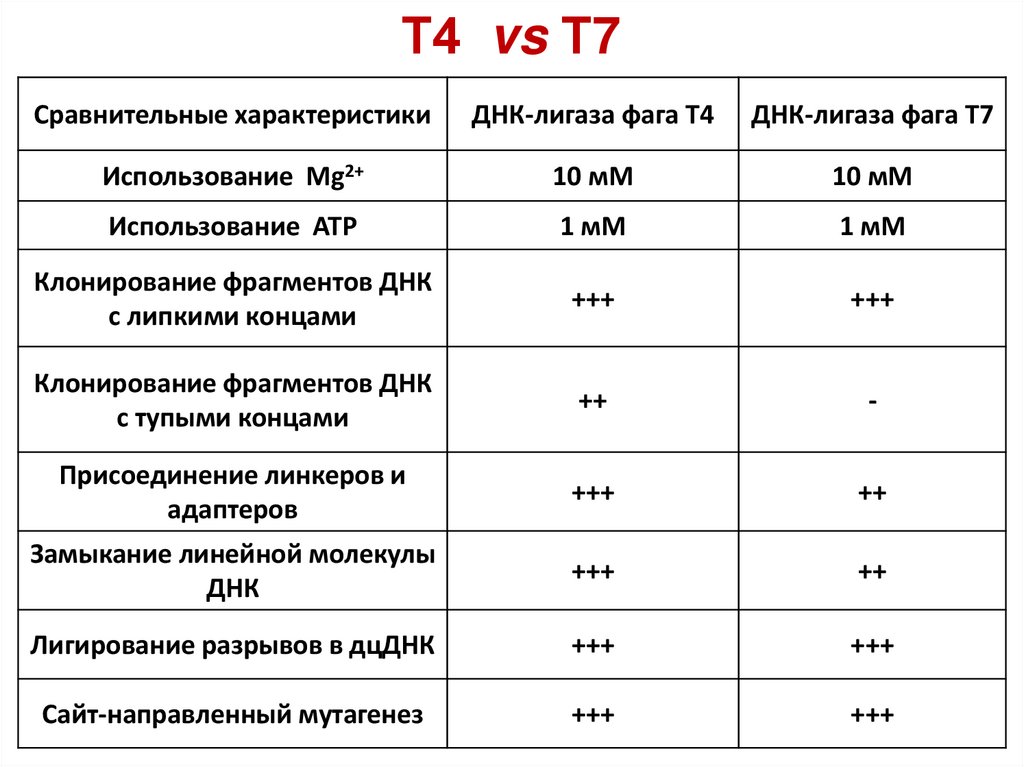
Т4 vs Т7Сравнительные характеристики
ДНК-лигаза фага T4
ДНК-лигаза фага T7
Использование Mg2+
10 мМ
10 мМ
Использование ATP
1 мМ
1 мМ
Клонирование фрагментов ДНК
с липкими концами
+++
+++
Клонирование фрагментов ДНК
с тупыми концами
++
-
Присоединение линкеров и
адаптеров
+++
++
Замыкание линейной молекулы
ДНК
+++
++
Лигирование разрывов в дцДНК
+++
+++
Сайт-направленный мутагенез
+++
+++
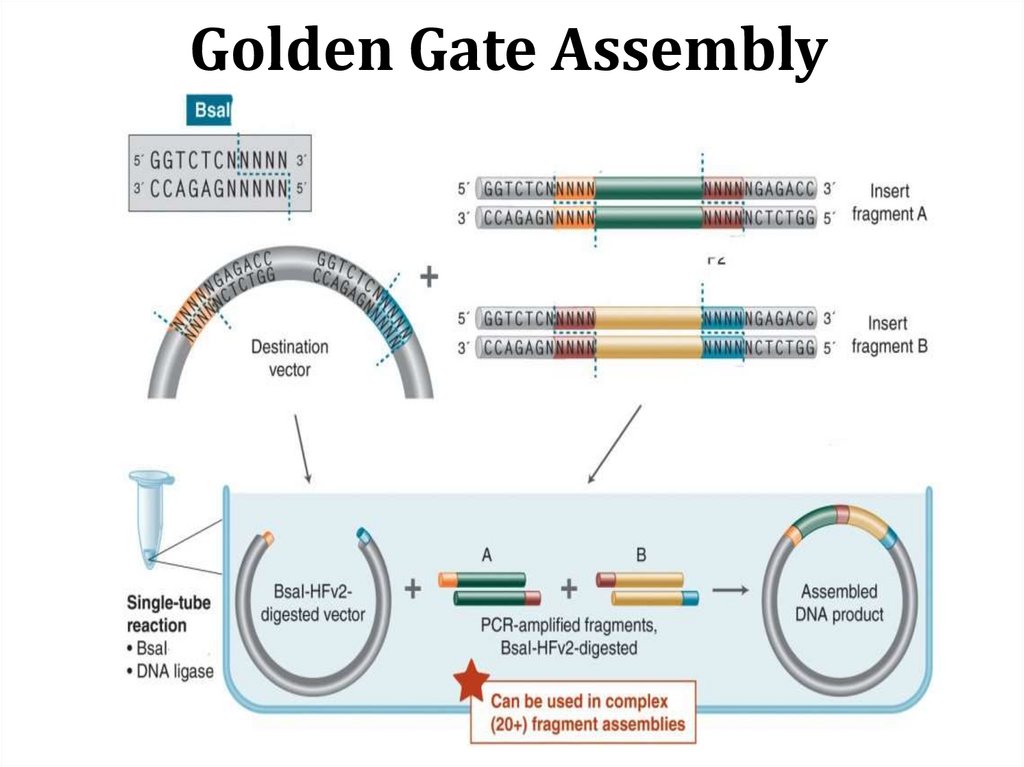
48. Golden Gate Assembly
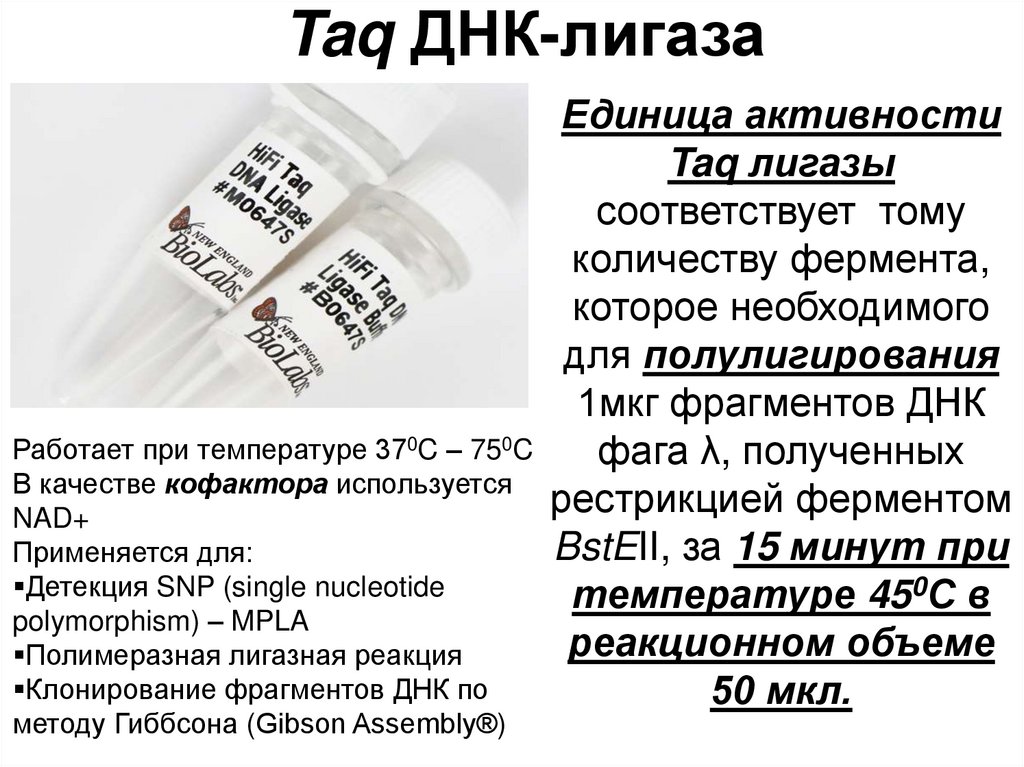
49. Taq ДНК-лигаза
Единица активностиTaq лигазы
соответствует тому
количеству фермента,
которое необходимого
для полулигирования
1мкг фрагментов ДНК
Работает при температуре 370C – 750C
фага λ, полученных
В качестве кофактора используется
рестрикцией ферментом
NAD+
BstEII, за 15 минут при
Применяется для:
Детекция SNP (single nucleotide
температуре 450С в
polymorphism) – MPLA
реакционном объеме
Полимеразная лигазная реакция
Клонирование фрагментов ДНК по
50 мкл.
методу Гиббсона (Gibson Assembly®)
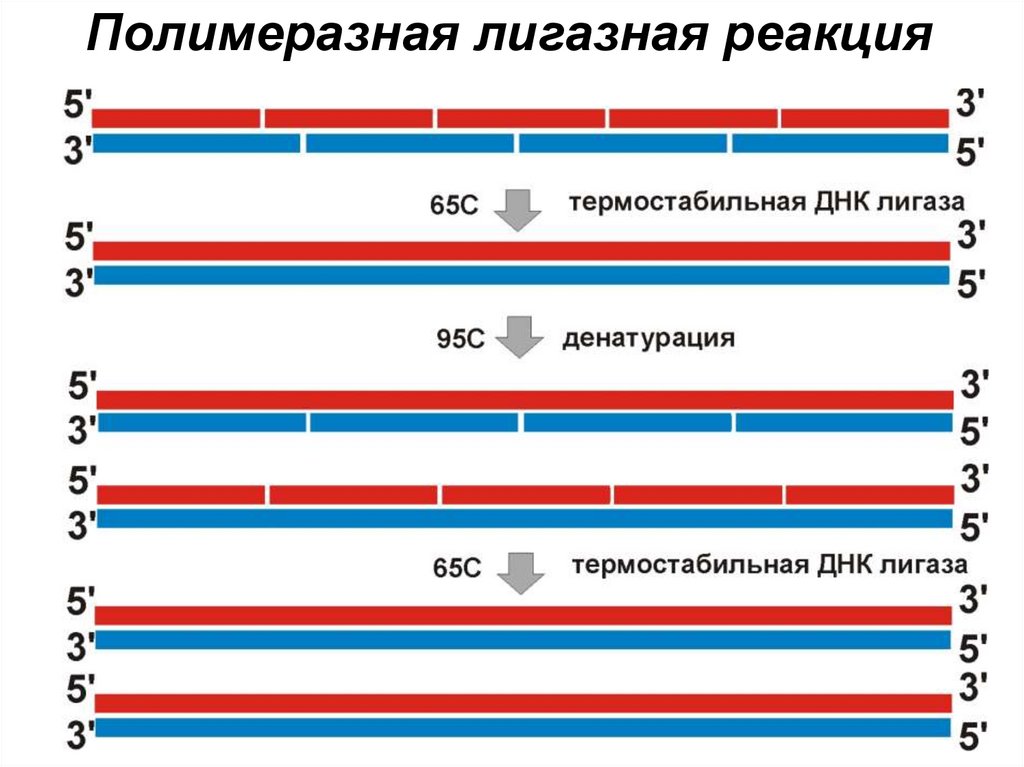
50.
Полимеразная лигазная реакция51.
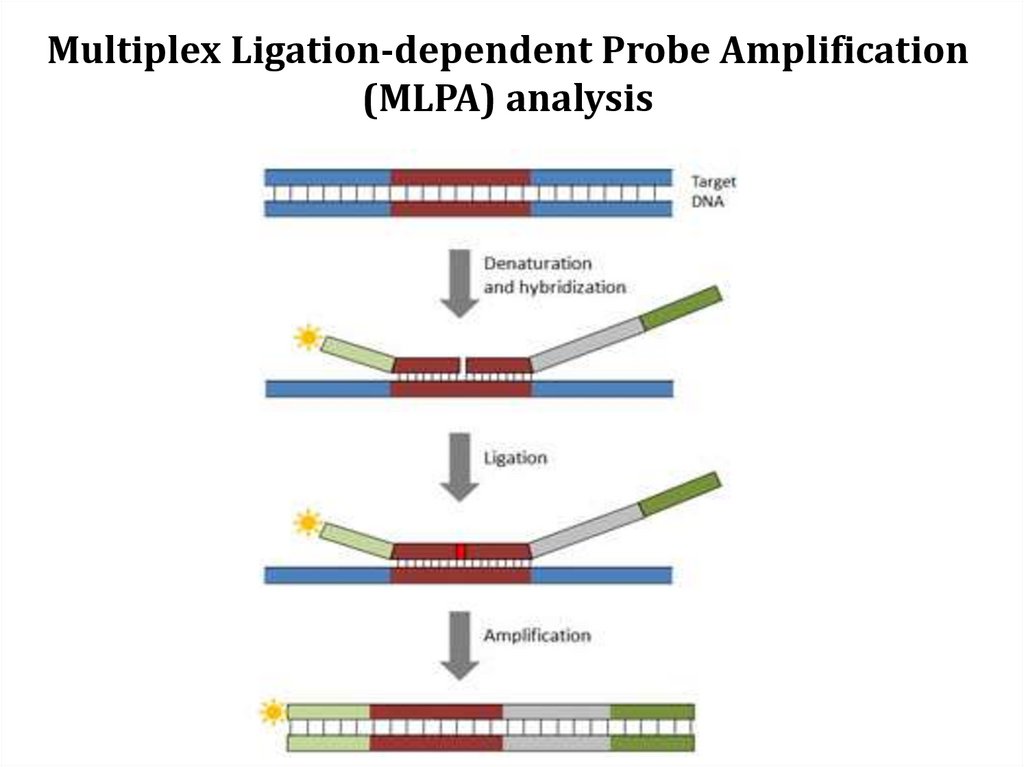
Multiplex Ligation-dependent Probe Amplification(MLPA) analysis
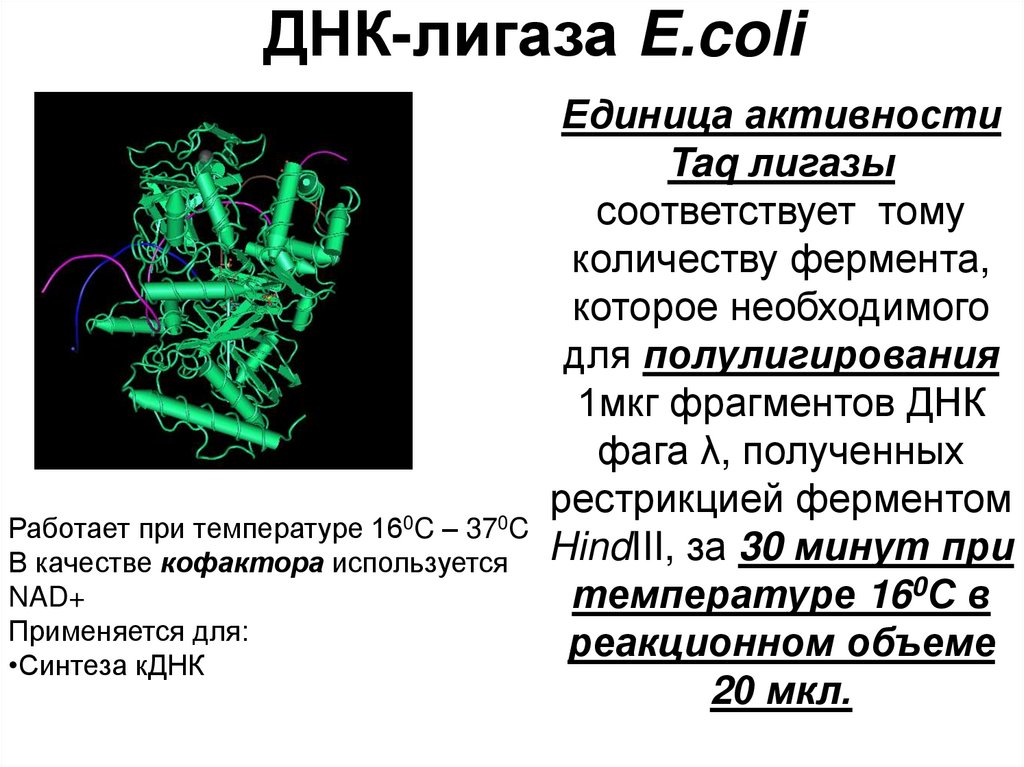
52. ДНК-лигаза E.coli
Единица активностиTaq лигазы
соответствует тому
количеству фермента,
которое необходимого
для полулигирования
1мкг фрагментов ДНК
фага λ, полученных
рестрикцией
ферментом
Работает при температуре 160C – 370C
В качестве кофактора используется HindIII, за 30 минут при
NAD+
температуре 160С в
Применяется для:
реакционном объеме
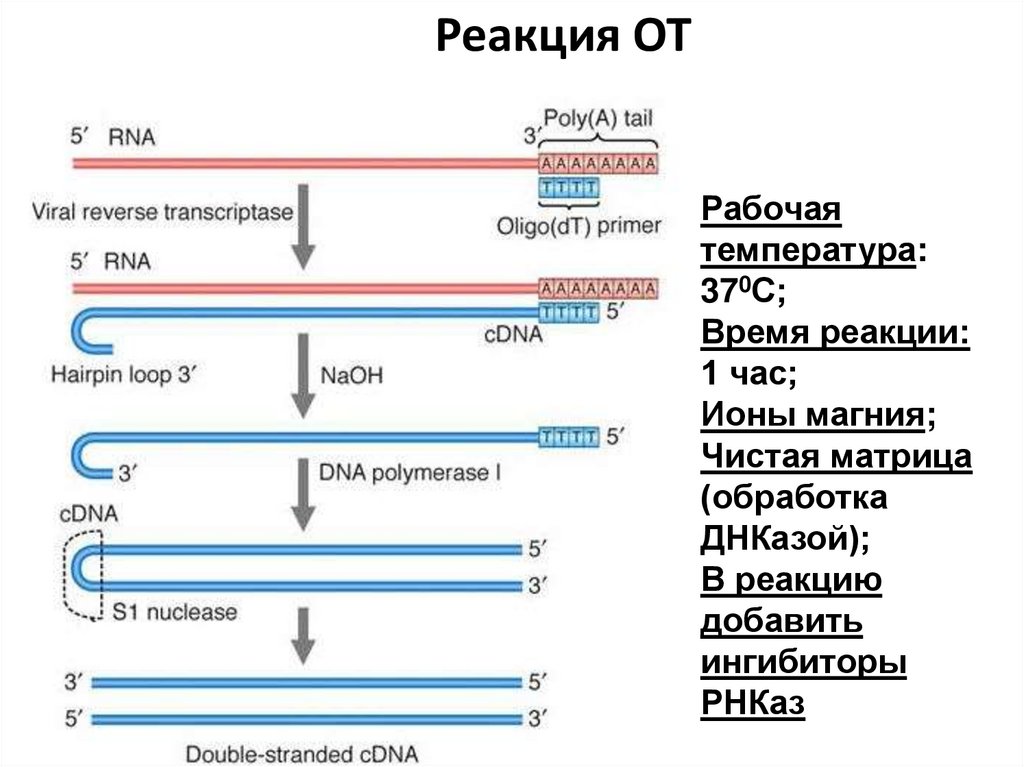
•Синтеза кДНК
20 мкл.
53.
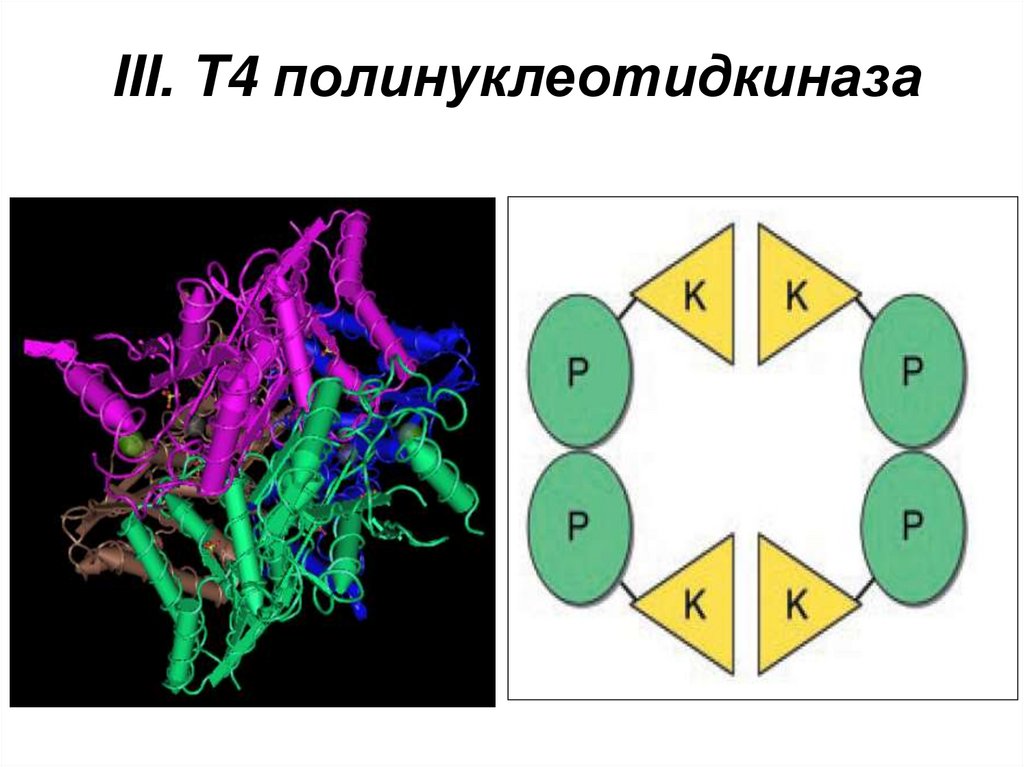
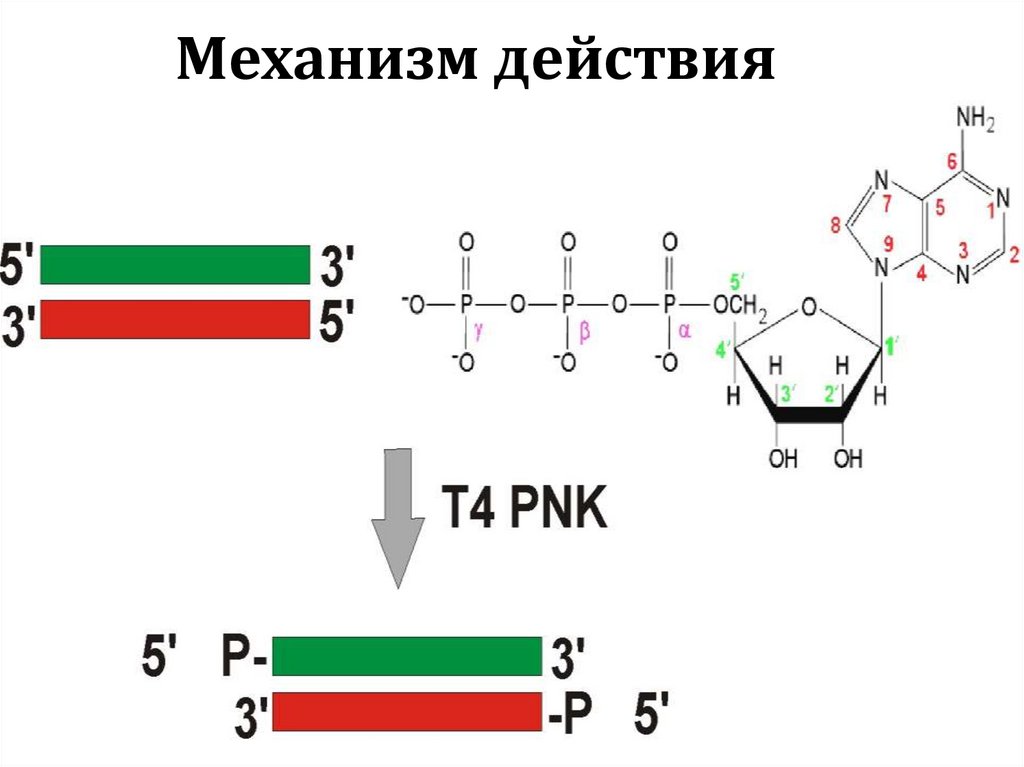
III. T4 полинуклеотидкиназа54. Механизм действия
55.
Активность полинуклеотидкиназыЕдиница активности
полинуклеотидкиназы
(единица Ричардсона)
соответствует количеству
фермента,
катализируемого
присоединение 1 нмоль
[32P] в реакционном
объеме 50 мкл за 30 минут
при 37С к 5’гидроксильным группам
ДНК спермы лосося.
56.
Препаративное фосфорилирование олигонуклеотидов.1. Смешать:
20-40 пмолей ДНК
1 мкл 10 х PNK-буфер
2 мкл 5 мМ rATP
1 мкл PNK (10 ед)
воды очищенной до 10 мкл.
2. Инкубировать при 37°С в течение 1 часа
3.Ингибировать реакцию при 70°С в течение 30 мин.
Для пересчета концентраций олигонуклеотидов используйте формулу:
C(мкМ, pmol/mkl) = 100 х OD / L, OD – количество оптических единиц в мл;
L – длина олигонуклеотида.
Пропись буфера 10 х PNK-буфер : 700 мМ Трис-НСl, 100 мМ MgCl2, 50 мМ DTT
(pH 7.6 при 25°С).
Эффективность фосфорилирования олигонуклеотида зависит
в немалой степени от основания, которое находится на его 5’-конце .
Эффективность фосфорилирования увеличивается в ряду:
G (60%)> A (45%) > T (45%) > C (10%).
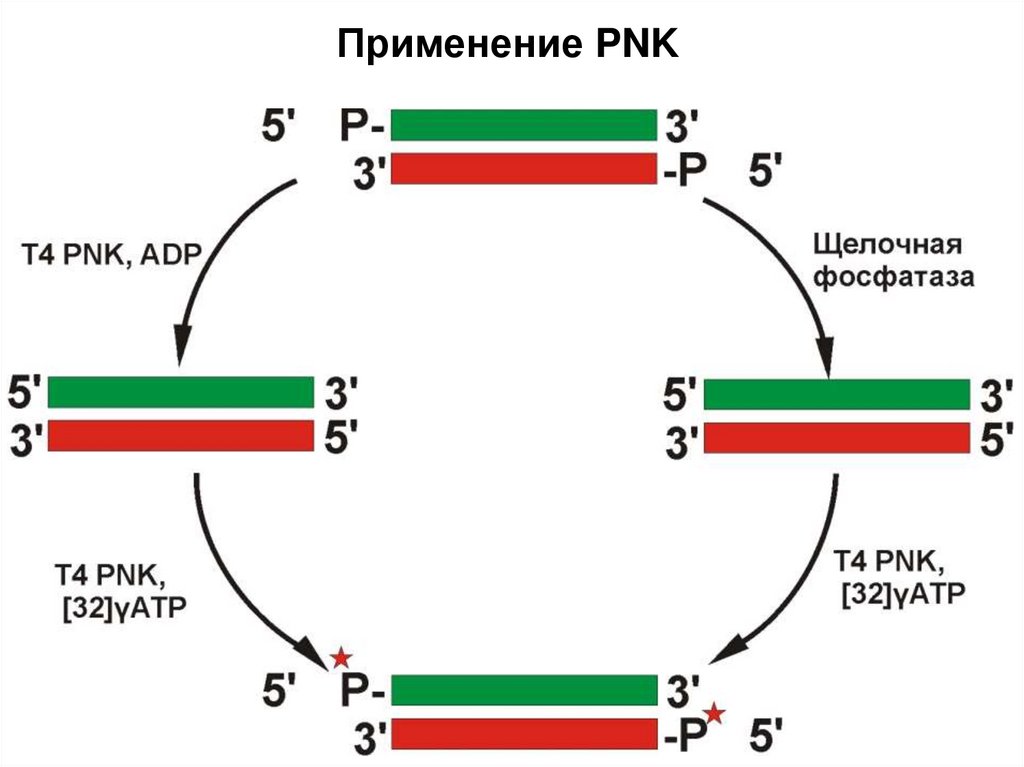
57. Применение PNK
58. Терминальная трансфераза (дезоксирибонуклеотидилтрансфераза)
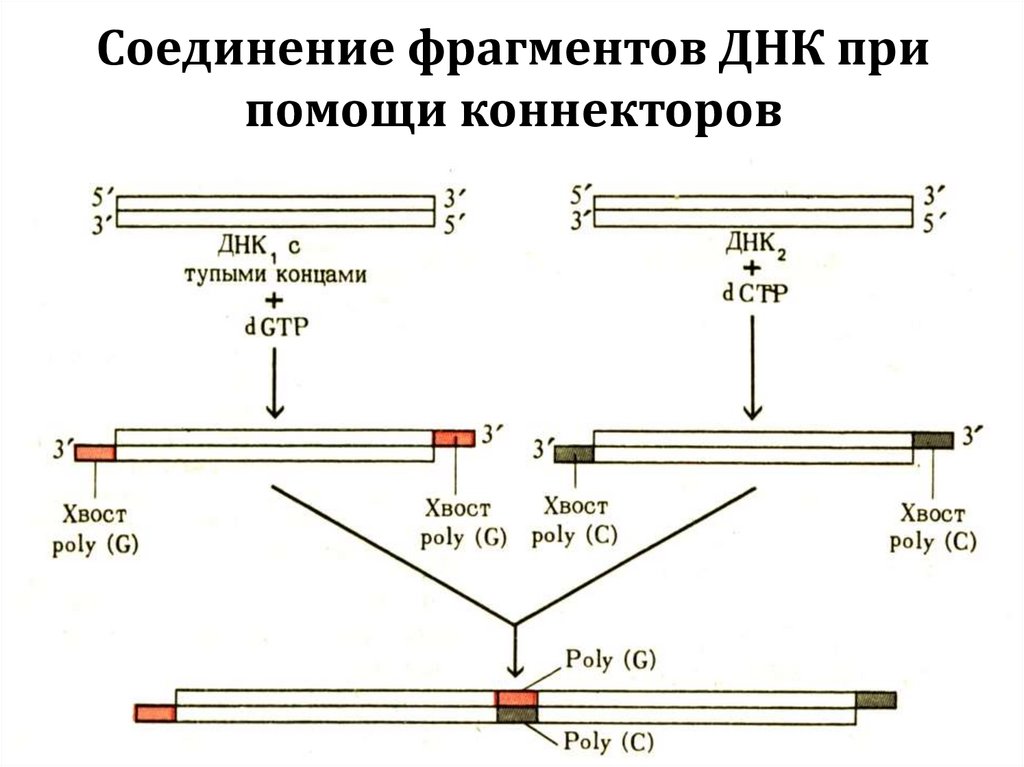
Осуществляет последовательное присоединениедезоксирибонуклеозидмонофосфатов к 3’-ОНгруппам молекул ДНК. Используется для введения
радиоактивной метки в составе меченых
нуклеотидов в 3’-концы ДНК, а также
присоединения к 3’-концам фрагментов ДНК
(кДНК) протяженных гомополимерных
последовательностей нуклеотидов (коннекторов)
для последующего их клонирования.
59. Соединение фрагментов ДНК при помощи коннекторов
60.
IV. Щелочные фосфатазы61.
Активность щелочной фосфатазыОдна единица активности
щелочной фосфатазы
соответствует количеству
фермента, необходимого для
полного дефосфолирирования
(удаления 5’-фосфатных групп)
1 мкг вектора pUC19,
расцепленного рестриктазами
HindIII (5´-выступающие), HincII
(липкие концы) или PstI (5’невыступающие концы) в
течение 30 минут при 370С.
62. Щелочные фосфатазы
CIAP (CAP)Calf Intestinal
SAP
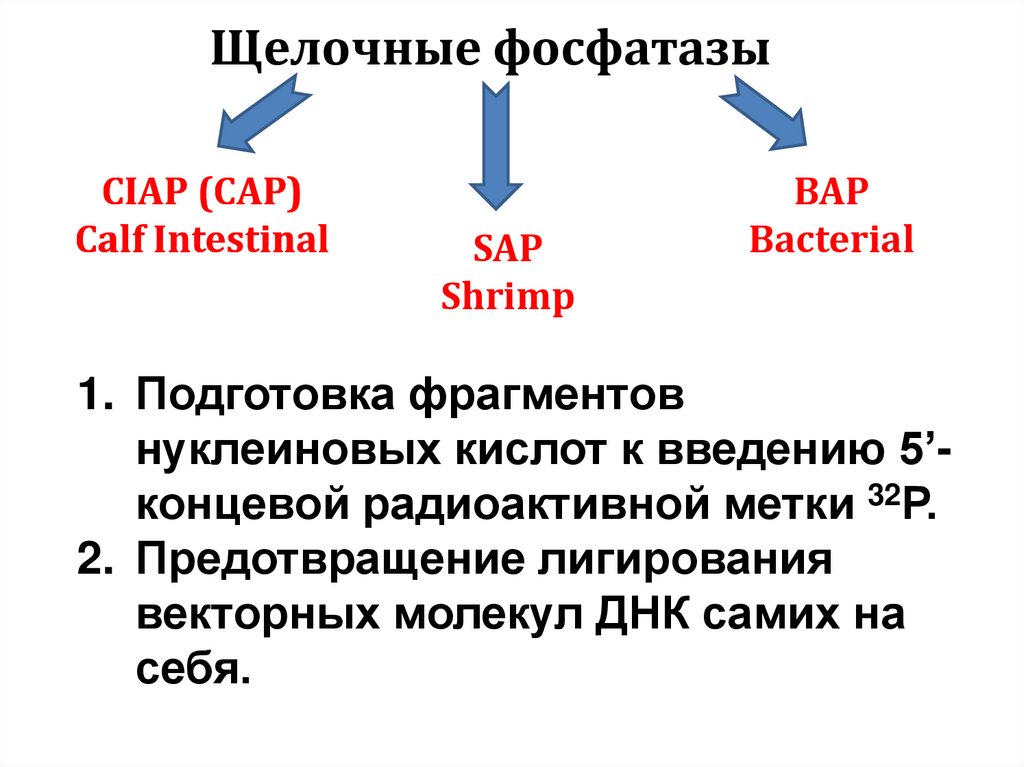
Shrimp
BAP
Bacterial
1. Подготовка фрагментов
нуклеиновых кислот к введению 5’концевой радиоактивной метки 32Р.
2. Предотвращение лигирования
векторных молекул ДНК самих на
себя.
63. Дефосфолирирование вектора
•Расщепить плазмидную ДНК подходящей рестриктазой. Экстрагировать реакционную смесьравным объемом фенол-хлороформ (1:1). Осадить ДНК этанолом. Промыть осадок 1 мл
70% этанола, высушить и растворить в 40 мкл 10 мМ Трис-НСl, рН 8.0.
•Добавить 5 мкл 10 х CIAP буфера.
•Добавить в реакционную смесь 0.5 мкл фосфатазы (1 ед/мкл). Инкубировать при 37°С в
течение 30 мин.
•Добавить 0.5 мкл фосфатазы и продолжить инкубирование еще 30 мин при 37°С.
•Добавить 300 мкл стоп-буфера: 10 мМ Трис-НСl, рН 7.5, 1 мМ ЕДТА, 200 мМ NaCl, 0.5%
SDS.
•Экстрагировать раствор равным объемом смеси фенол-хлороформ (1:1).
•Добавить 10 мкл объема 3 М ацетат Na, рН 6.0 и 300 мкл 96% - этанола. Оставить на столе
в течение 15 мин. Затем центрифугировать при 12000 g в течение 15 мин.
•Осадок промыть 1 мл 70%-этанола, высушить и растворить в 45 мкл воды.
•Добавить 5 мкл 10 х лигазного буфера и 2 ед. Т4 ДНК лигазы. Инкубировать при 16°С в
течение 16 ч.
•Фракционировать продукты реакции на 0.8% агарозном геле. Вырезать ту полосу, которая
соответствует линеаризованной плазмиде.
Если в качестве субстрата выступает ДНК с выступающими 3'-концами, то
рекомендуется проводить инкубацию при 37°С в течение 15 мин и при 56°С в течение 15
мин. Затем добавить вторую порцию фермента и обработку повторить в тех же
условиях.
64.
V. Нуклеазы – гидролитическиеферменты, расщепляющие
фосфодиэфирные связи в
нуклеиновых кислотах
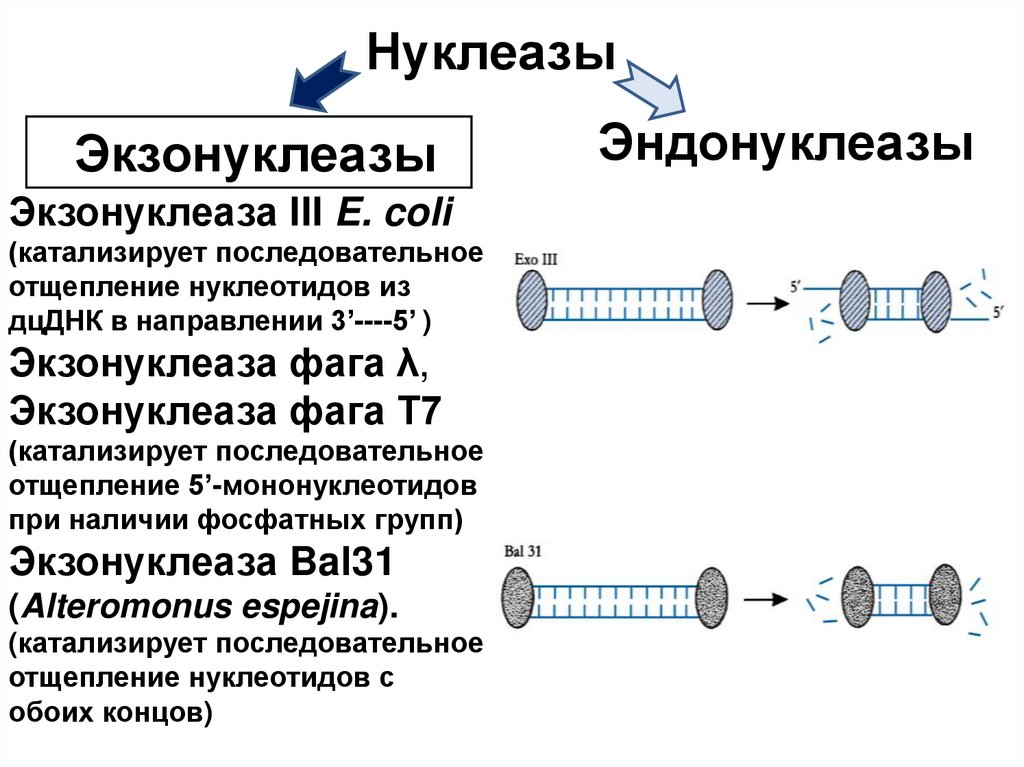
65. Нуклеазы
ЭкзонуклеазыЭкзонуклеаза III E. coli
(катализирует последовательное
отщепление нуклеотидов из
дцДНК в направлении 3’----5’ )
Экзонуклеаза фага λ,
Экзонуклеаза фага Т7
(катализирует последовательное
отщепление 5’-мононуклеотидов
при наличии фосфатных групп)
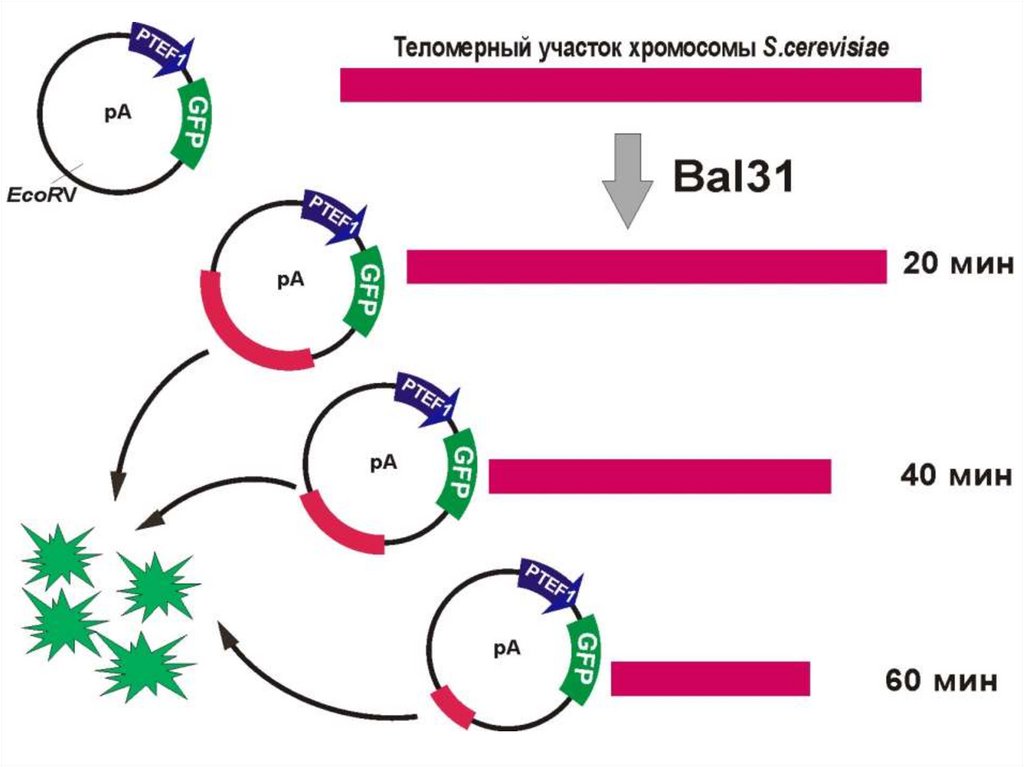
Экзонуклеаза Bal31
(Alteromonus espejina).
(катализирует последовательное
отщепление нуклеотидов с
обоих концов)
Эндонуклеазы
66. Нуклеазы
ЭкзонуклеазыЭндонуклеазы
Нуклеаза S1 (Aspergillus
orizae)
Нуклеаза Mung Bean
Нуклеаза P1 (Penicillium
citrinum )
(распознает и расщепляет
одноцепочечные участки в
одноцепочечных разрывах)
РНКазаА (расщепляет
фосфодиэфирные связи,
образованные пиримидиновыми
нуклеотидами)
ДНКазаI
67.
Удаление 3’- и 5’-выступающих концовпри помощи Mung Bean Nuclease
1. Суспендировать ДНК (0.1 мкг/мкл) в 1
Mung Bean Nuclease буфере.
2. Добавить 1 ед. Mung Bean Nuclease на
каждый мкг ДНК.
3. Инкубировать при 30°С в течение 30 мин.
4. Инактивировать фермент экстракцией
фенол/хлороформ или путем добавления SDS до
0.01%.
68.
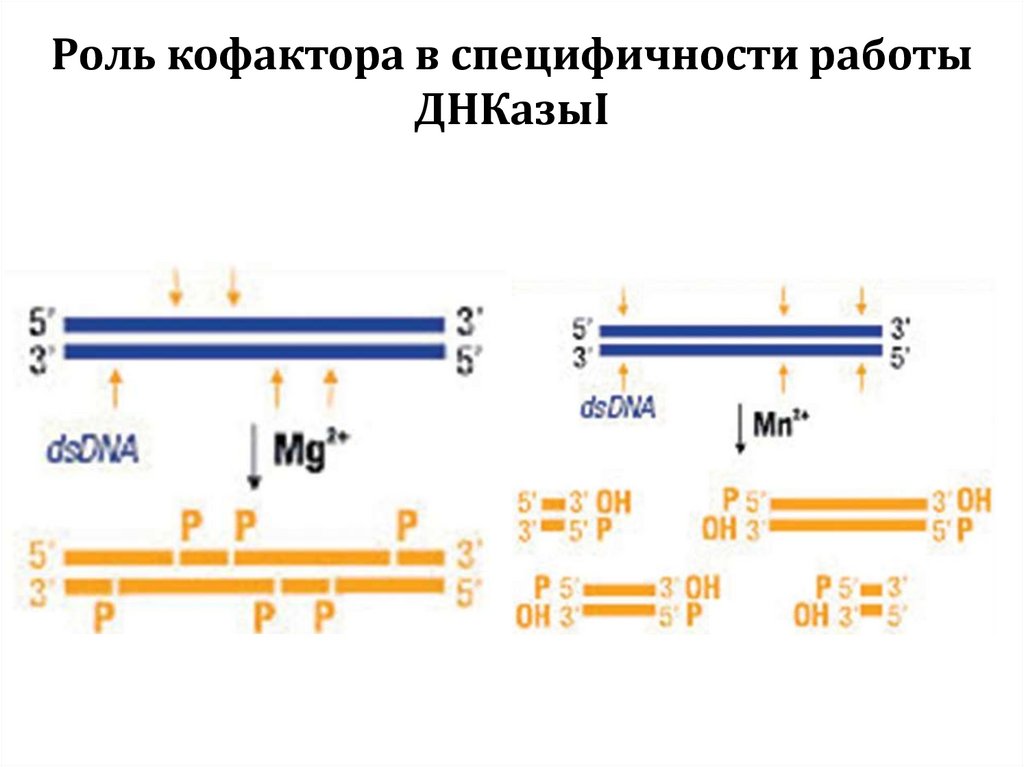
69. Роль кофактора в специфичности работы ДНКазыI
70.
Эндонуклеаза I фага T7Распознает и расщепляет не идеально спаренные участки ДНК,
крестообразные структуры ДНК, структуры Холлидея,
гетеродуплексную ДНК, ДНК-РНК гибриды, ники
Генотипирование
трансгенных организмов
Создание ников
Распознавание событий
рекомбинации
71. V. ДНК-полимеразы
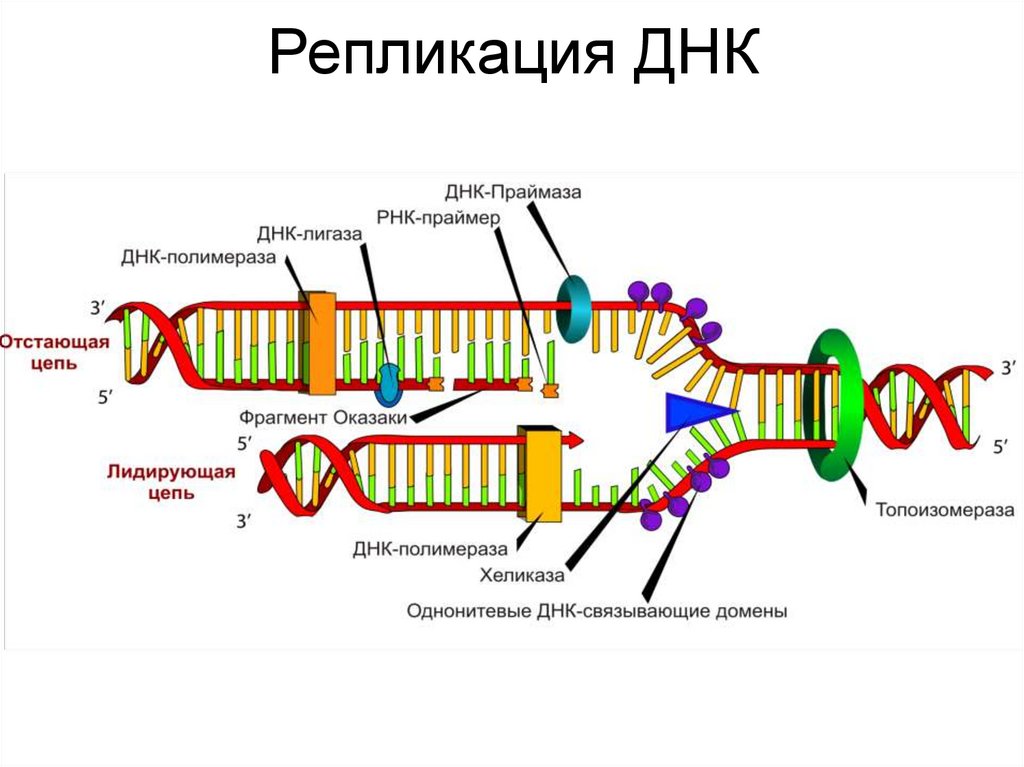
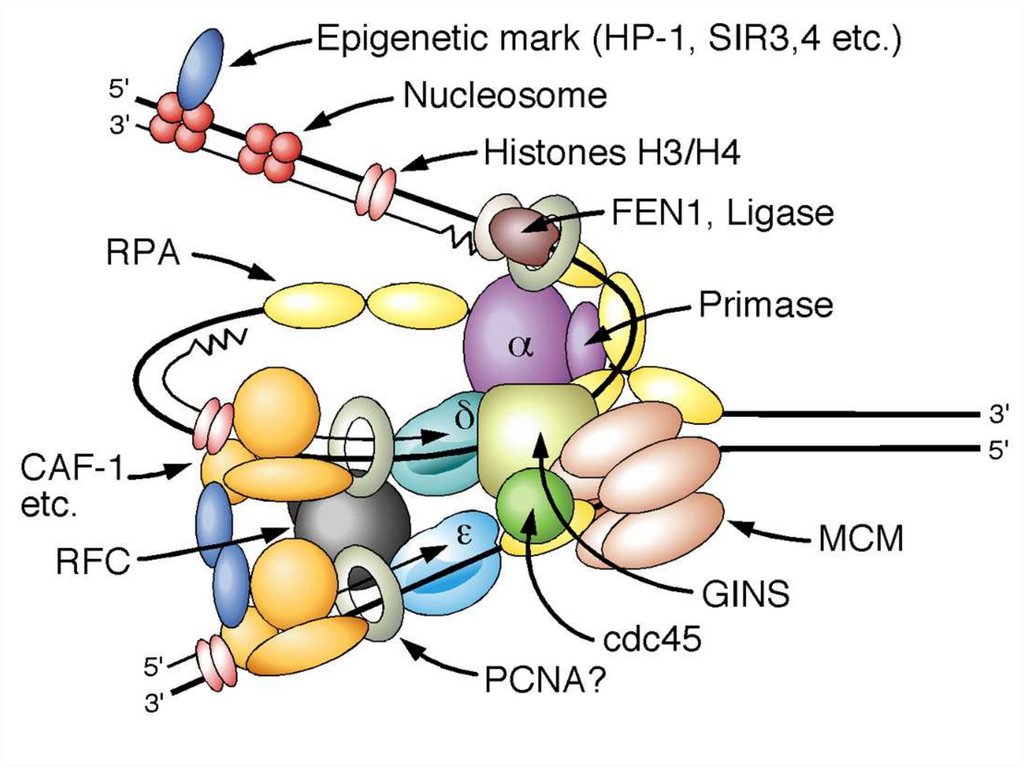
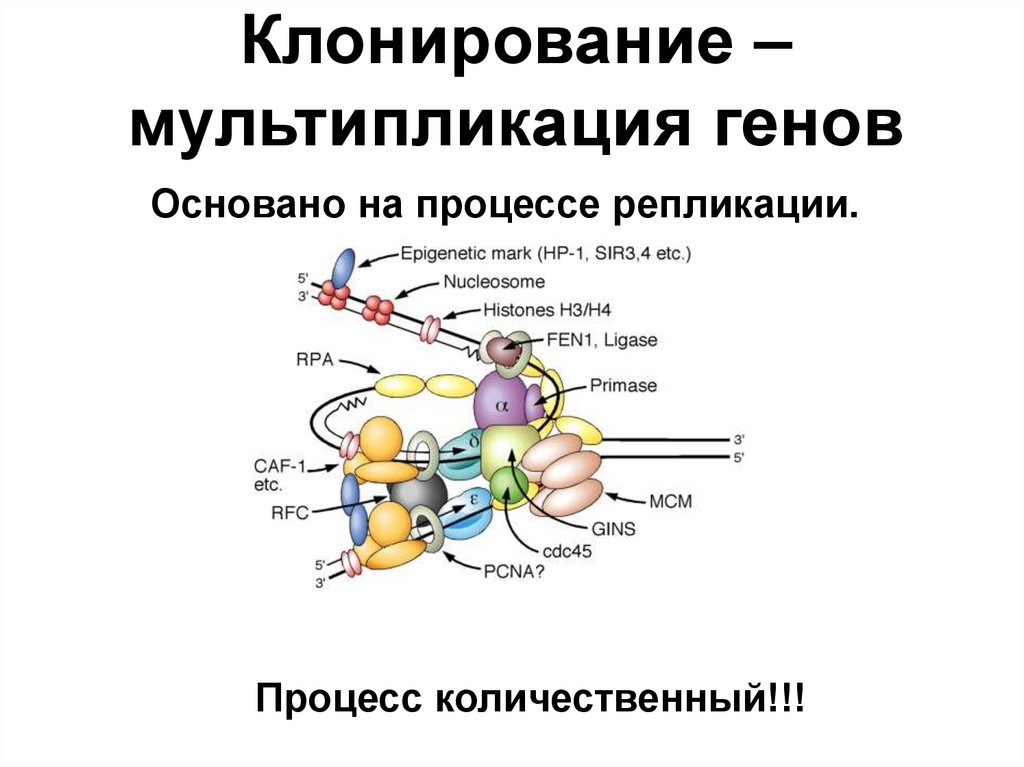
72. Репликация ДНК
73.
74.
Компоненты репликационной машиныE. сoli
dnaA
Primasome
dnaB
dnaC
dnaG
DNA gyrase
Rep protein
SSB
DNA pol III
DNA pol I
DNA ligase
Связывается с участком начала репликации
хеликаза (расплетает ДНК в ori)
связывается с dnaB
праймаза (синтезирует РНК праймер)
вносит отрицательные супервитки перед
репликативной вилкой
хеликаза (расплетает ДНК перед
репликативной вилкой)
связывается с однонитевой ДНК
главная репликативная полимераза
удаляет РНК-праймеры и заполняет бреши
«зашивает» однонитевые разрывы
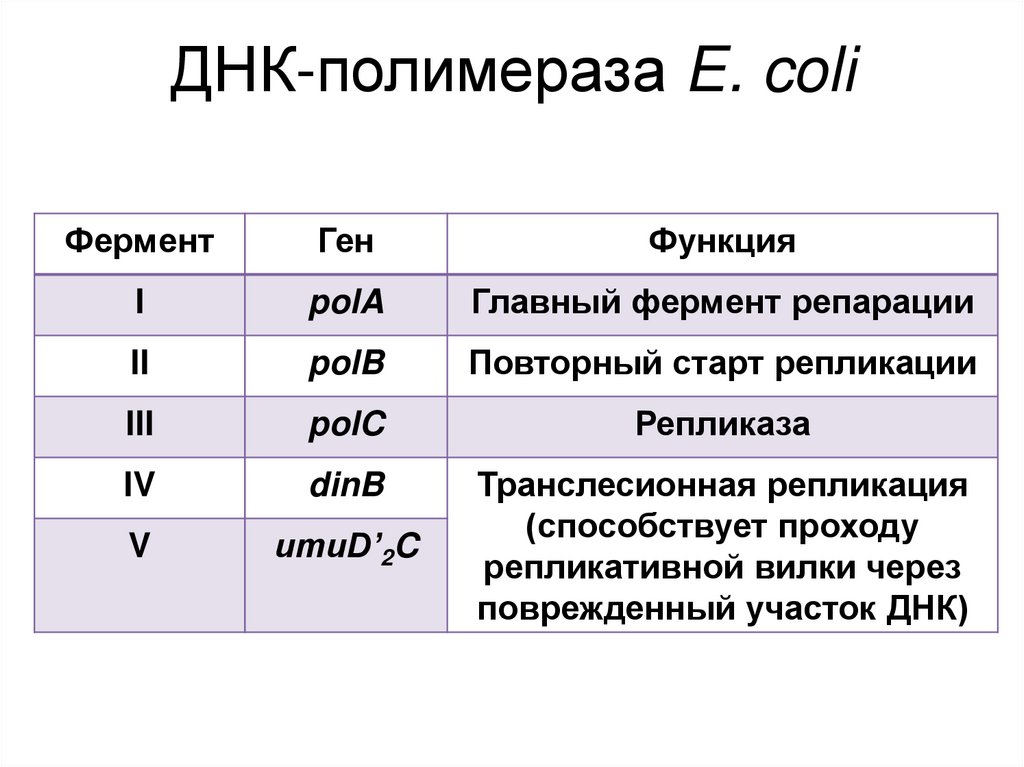
75.
ДНК-полимераза E. coliФермент
Ген
Функция
I
polA
Главный фермент репарации
II
polB
Повторный старт репликации
III
polC
Репликаза
IV
dinB
V
umuD’2C
Транслесионная репликация
(способствует проходу
репликативной вилки через
поврежденный участок ДНК)
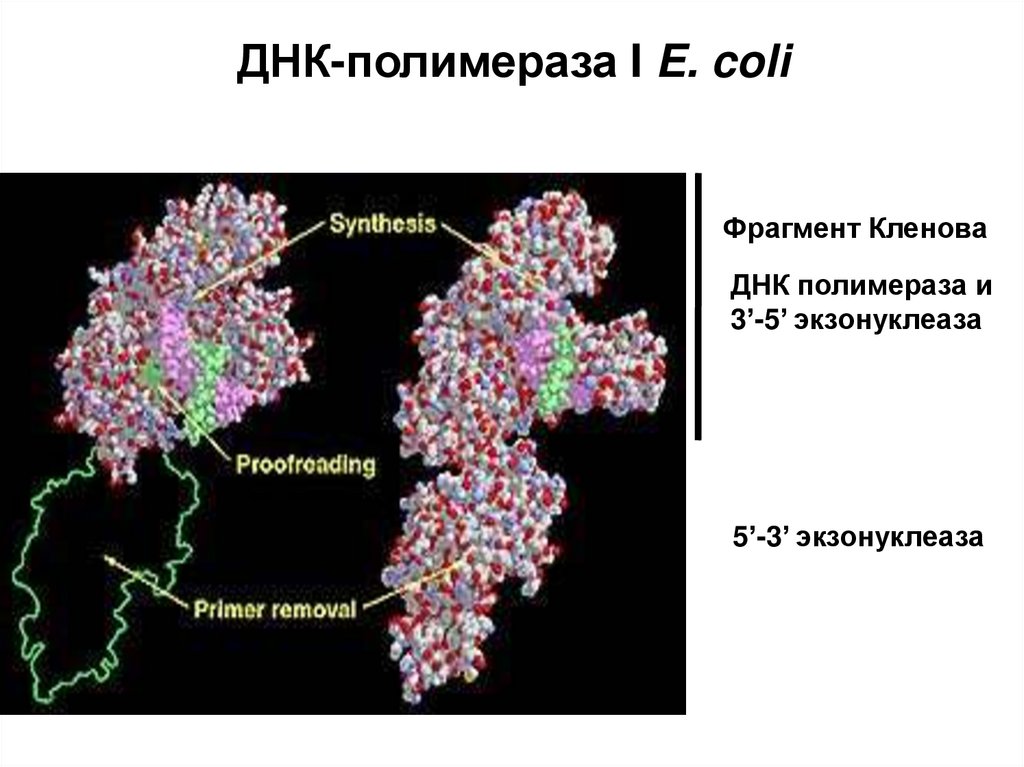
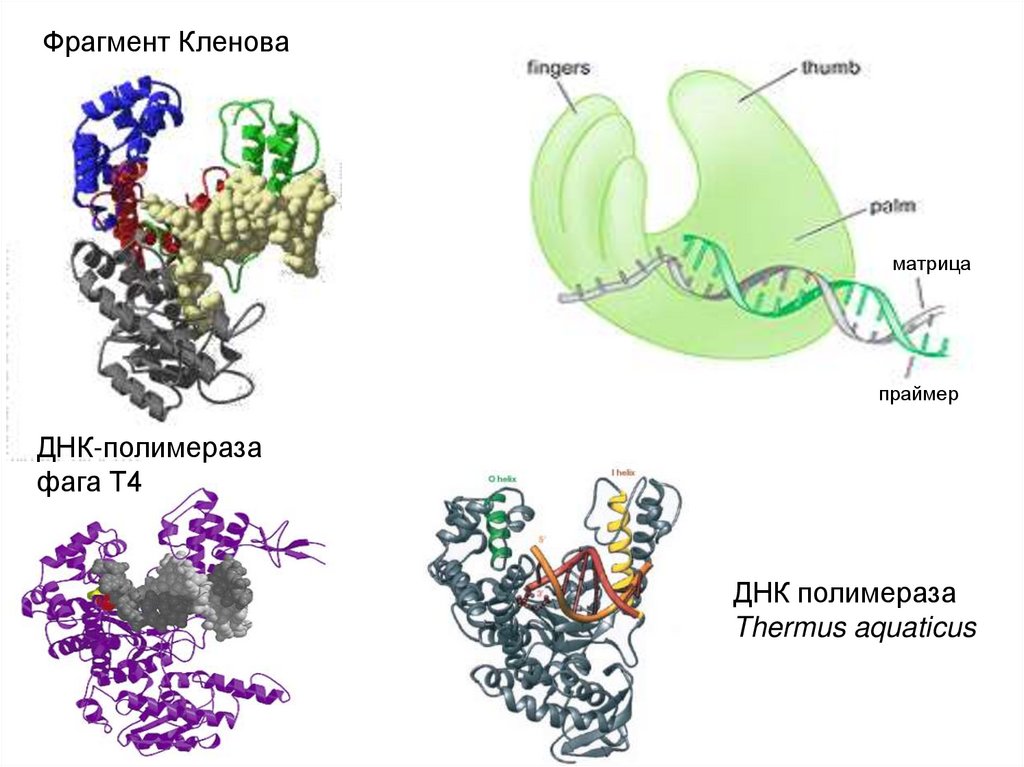
76. ДНК-полимераза I E. coli
Фрагмент КленоваДНК полимераза и
3’-5’ экзонуклеаза
5’-3’ экзонуклеаза
77.
Фрагмент Кленоваматрица
праймер
ДНК-полимераза
фага Т4
ДНК полимераза
Thermus aquaticus
78. Функции доменов фрагмента Кленова
Домен пальцы:Связывает dNTP, и помещает правильно спаренные dNTP в
каталитический центр
Связывает матрицу и обеспечивает экспонирование основания, с
которым должен спариваться очередной dNTP
Домен большой палец:
Прямо не участвует в катализе
Взаимодействует с матрицей и синтезирующейся ДНК с тем, чтобы
обеспечить правильную позицию праймера относительно активного
центра, одновременно препятствуя «соскакиванию» полимеразы с
ДНК
Домен ладонь:
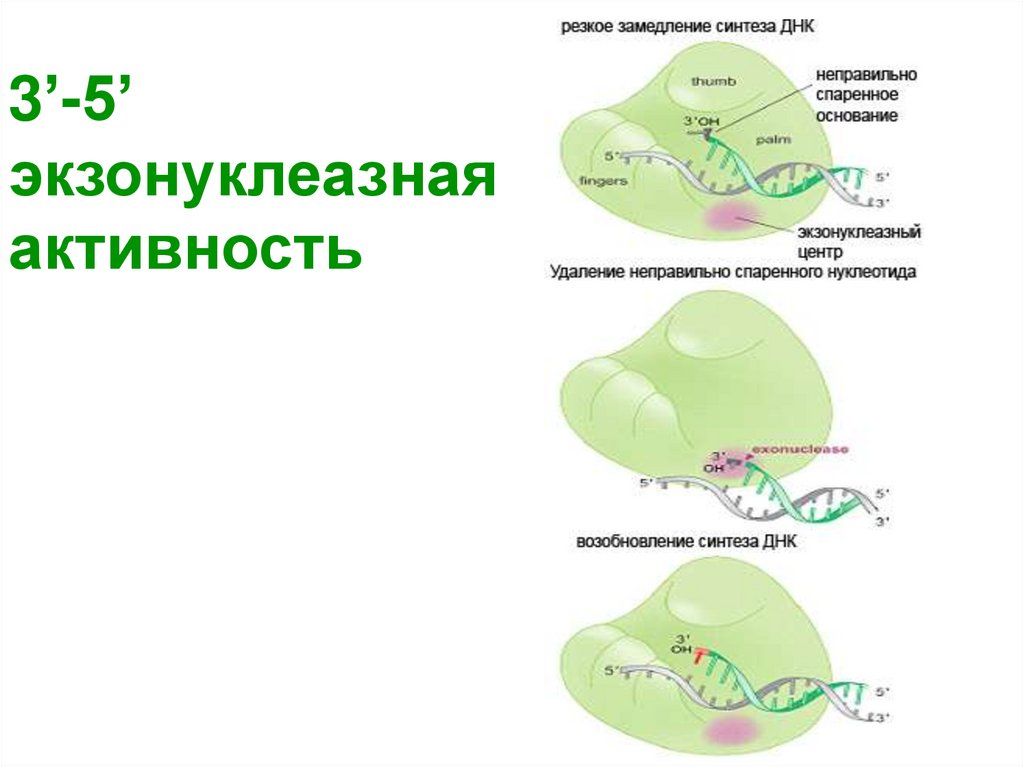
3’ ---- 5’ экзонуклеазная активность
79. 3’-5’ экзонуклеазная активность
80. Активность фрагмента Кленова
Одна единица большогофрагмента ДНКполимеразы I E. coli
(фрагмента Кленова)
соответствует количеству
фермента, необходимого
для полного
присоединения 10 нмоль
dNTP в течение 30 минут
при 37°C
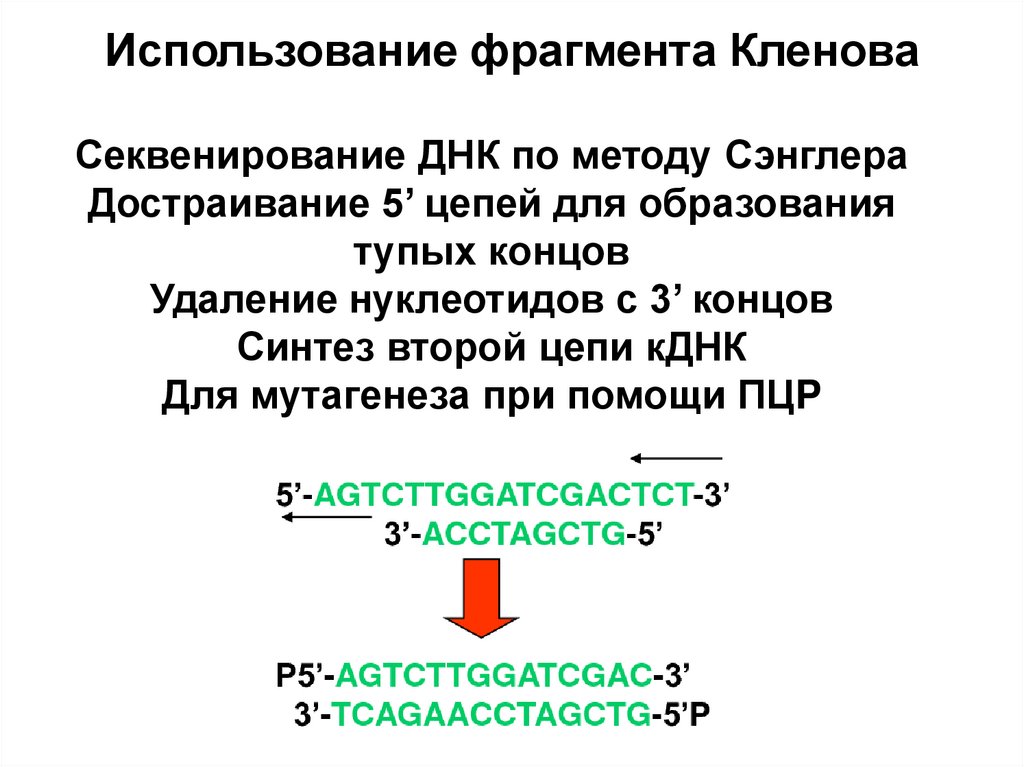
81. Использование фрагмента Кленова
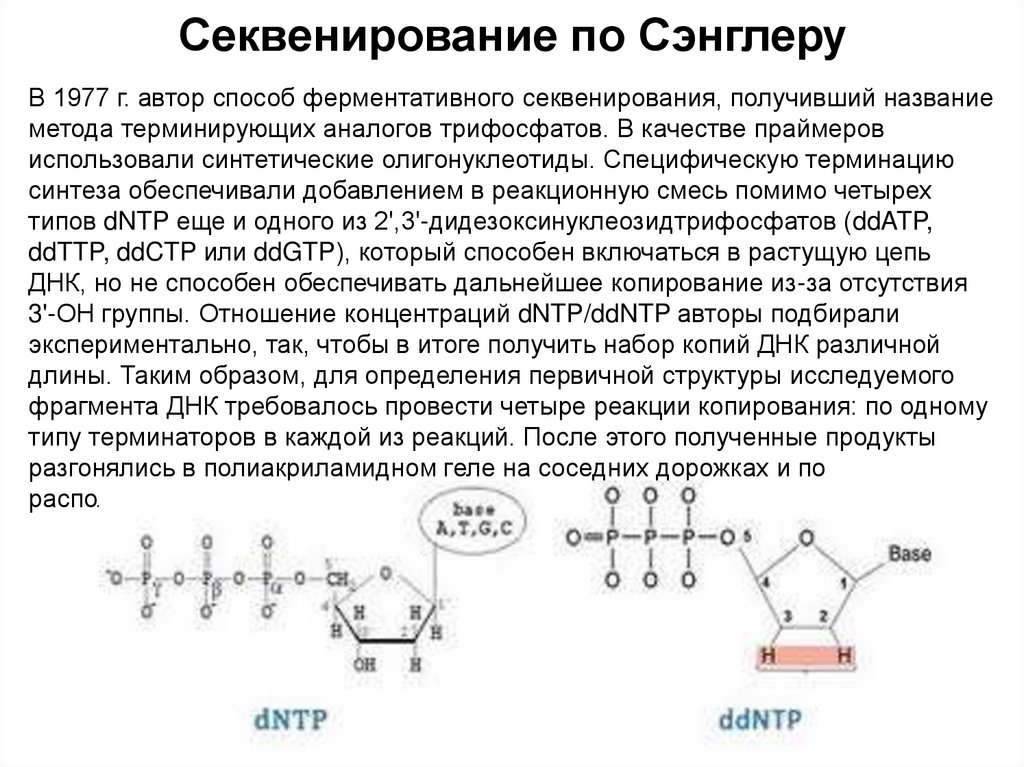
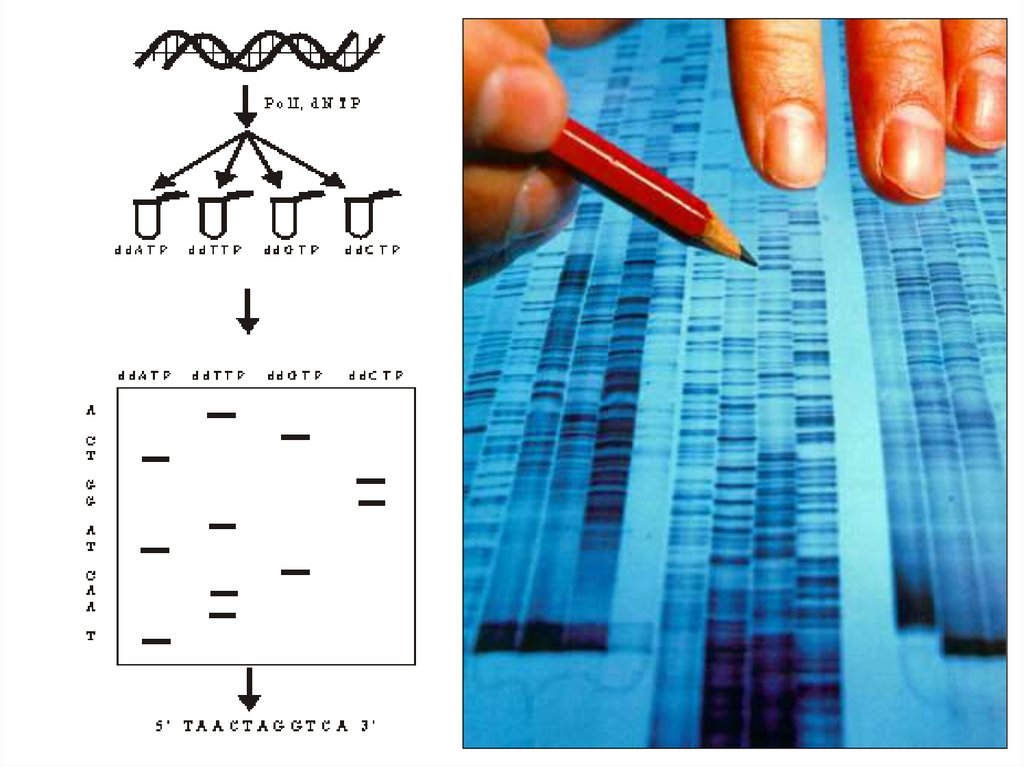
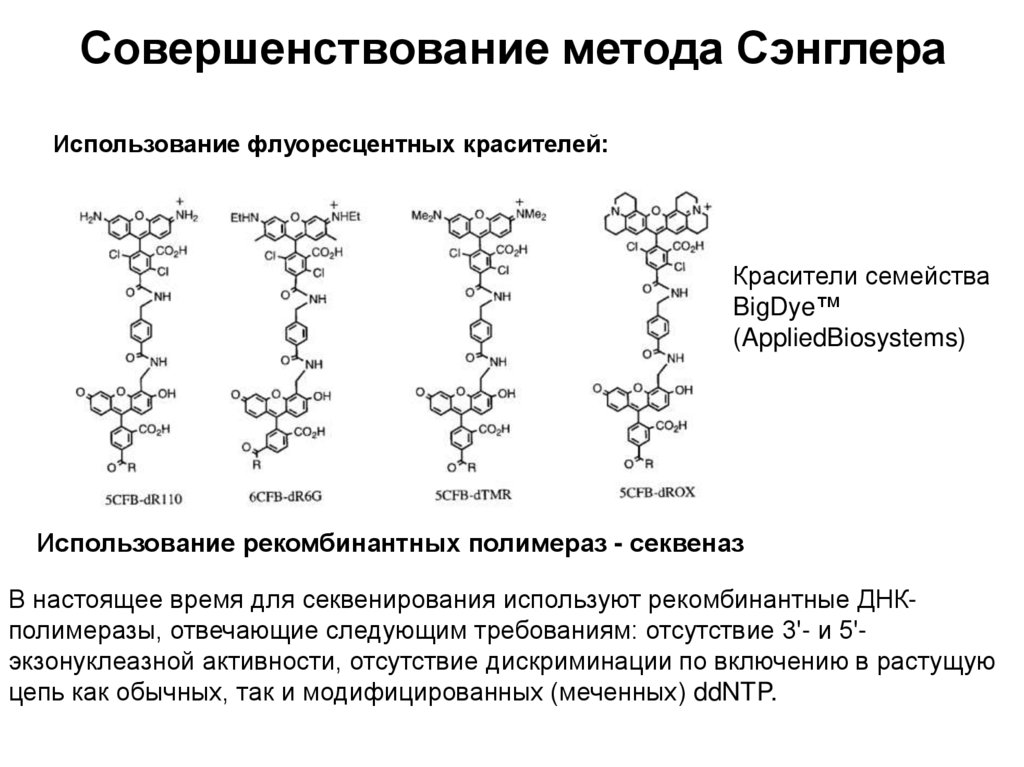
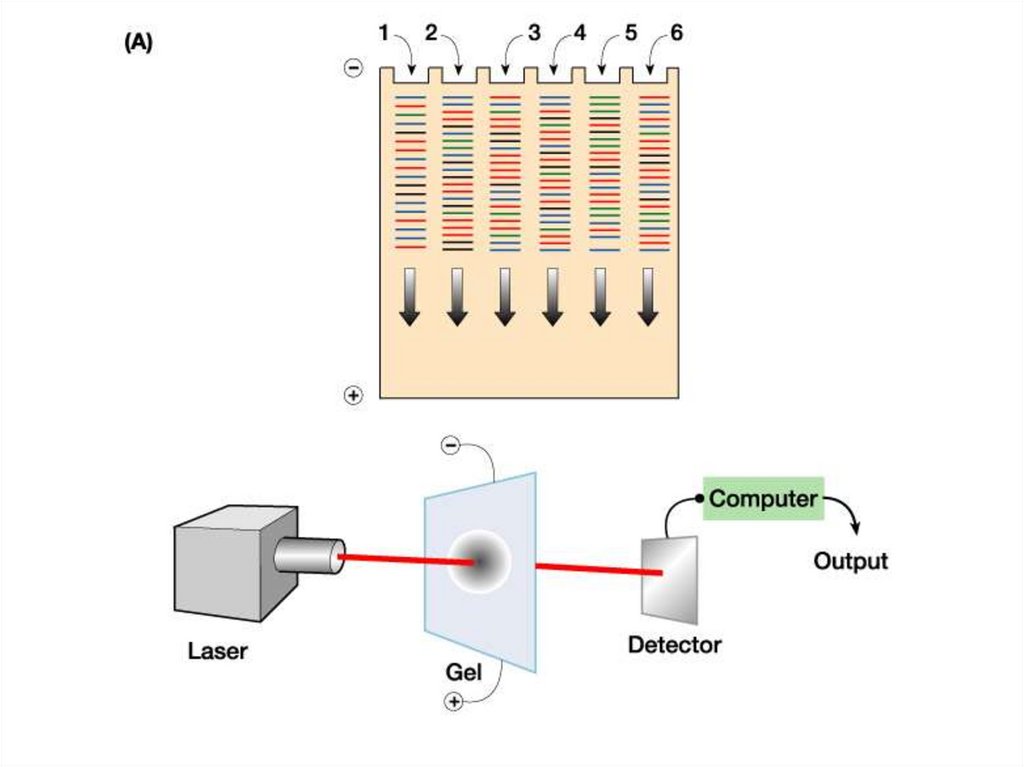
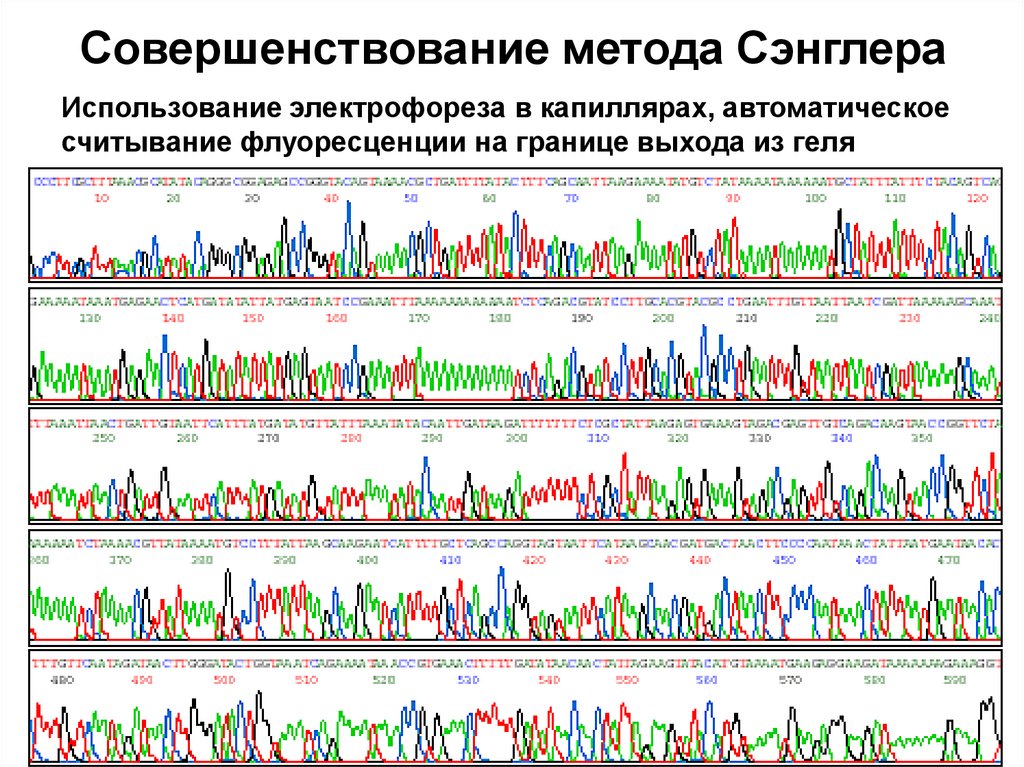
Секвенирование ДНК по методу СэнглераДостраивание 5’ цепей для образования
тупых концов
Удаление нуклеотидов с 3’ концов
Синтез второй цепи кДНК
Для мутагенеза при помощи ПЦP
82.
Затупление 5’-выступающих концов при помощифрагмента Кленова
По окончании рестрикции добавить в реакционный раствор:
dNTPs до конечной концентрации 0.5 мМ,
2 ед. фрагмента Кленова.
Инкубировать при комнатной температуре в течение 30 мин.
Добавить ТЕ-буфера до 100 мкл и 100 мкл нейтрализованного
фенол/хлороформа. Перемешать тщательно смесь на вортексе ( 30 сек
на больших оборотах). Центрифугировать при 12 000 g в течение 3 мин.
Отобрать осторожно верхнюю (водную) фазу в новую пробирку.
Добавить к органической фазе 100 мкл ТЕ-буфера. Перемешать
тщательно смесь на вортексе. Центрифугировать при 12 000 g в течение
3 мин.
Отобрать осторожно водную фазу и объединить ее с той, которая была
отобрана раньше.
Добавить 20 мкл 3 М ацетата Na, рН 6.0 и 600 мкл 96%-го этанола.
Далее провести осаждение ДНК
83. ДНК-полимеразы термофильных архебактерий
Полимераза ИсточникTaq
Thermus
aquaticus
Vent
Thermococcus
litoralis
Deep
Vent
Pyrococcus
sp. GB-D
Pfu
Pyrococcus
furiosus
3’-5’ эндо
нуклеазная
активность
Ошибка
Особенности
-
10-4
Добавляет полиА
последовательнос
ти на концы
фрагмента
+
4×10-5
+
4×10-5
+
2×10-6
Высокоточная
84. ДНК-зависимая ДНК-полимераза Thermophilus aquaticus (Taq)
Впервые изолирована в 1976 году из штамма YT-1 T. aquaticus, описанногомикробиологом Томасом Броком (Chien et al, 1976)
85.
Ген Taq-полимеразы кодирует полипептид (Mr = 93.9 kDa), состоящий из двухдоменов (832 а.о.) и обладающий 3 различными ферментативными
активностями (Lawyer, 1989):
1. N-концевой (1 – 290 а.о.) схож по структуре и функциям с
5’→ 3’ экзонуклеазным доменом ДНК-полимеразы I E. coli.
Некоторые коммерческие ферментные препараты
содержат полимеразу без N-концевого фрагмента
(KlenTaq, фрагмент Штоффеля (Stoffel fragment)), однако
такой фермент менее эффективен и процессивен
2. Основной полимеразный домен (424 – 831 а.о.) очень
похож на фрагмент Кленова. (Joyce and Steitz 1994, 1995;
Pelletier, 1994; Perler et al, 1996)
3. Обладает независимой, но низкой трансферазной
активностью, который способен нуклеотиды на 3’-конец
(обычно 3’-А)
4. Остатки 294 – 422 включают в себя неактивный 3’→5’
экзонуклеазный домен. Таким образом, более 50%
ампликонов, произведенных ферментом за 25 циклов
будут иметь ошибки!
5. Термостабильность обусловлена повышением
гидрофобности кора фермента, усиленное стабилизацией
электростатических взаимодействий и улучшенным
контактом с молекулами растворителя. Последнее
обеспечивается увеличением содержания остатков
пролина на поверхности молекулы, по сравнению с ДНКполимеразой I E. coli
86. Биохимические свойства Taq ДНК-полимеразы
Биохимические свойства Taq ДНКполимеразыОптимальная температура реакции: 75°С – 80°С;
Оптимальная концентрация ионов Mg2+: 1.5 мМ
Оптимальная ионная сила: 50 – 55 мМ КСl
Оптимальный рН раствора: 7.8 – 9.0
Константы Михаэлиса фермента для
ДНК: 1.5 нМ
dNTP: 10 – 15 мкМ
Скорость амплификации (молекул dNTP за 1 секунду на одну молекулу
фермента):
при 97°С – 900
при 75°С – 150
при 70°С – <60
при 55°С – 24
при 37°С – 1.25
при 22°С – 0.25
Процессивность (молекул dNTP за 1 секунду на одну молекулу фермента):
42
Время полужизни фермента:
97.5°С – 9 минут
95°С – 40 минут
92.5°С – 130 минут
87.
1 единица Taq-полимеразы определяет такое количествофермента, которое необходимо для присоединения 10
нмоль dNTP за 30 минут при 74°С
Среднее количество единиц в 1 мг коммерческой Taq-полимеразы составляет
80000 единиц, т.о. 1 единица активности содержит примерно 8×1010 молекул
фермента
88.
89.
Генно-инженерные ДНКполимеразы90. Инженерия ДНК-полимераз: ДНК-полимераза Phusion высокой точности
Инженерия ДНК-полимераз: ДНКполимераза Phusion высокой точности91.
Pyrococcusfuriosus
Thermococcus
kodakaraensis
Thermus
aquaticus
92.
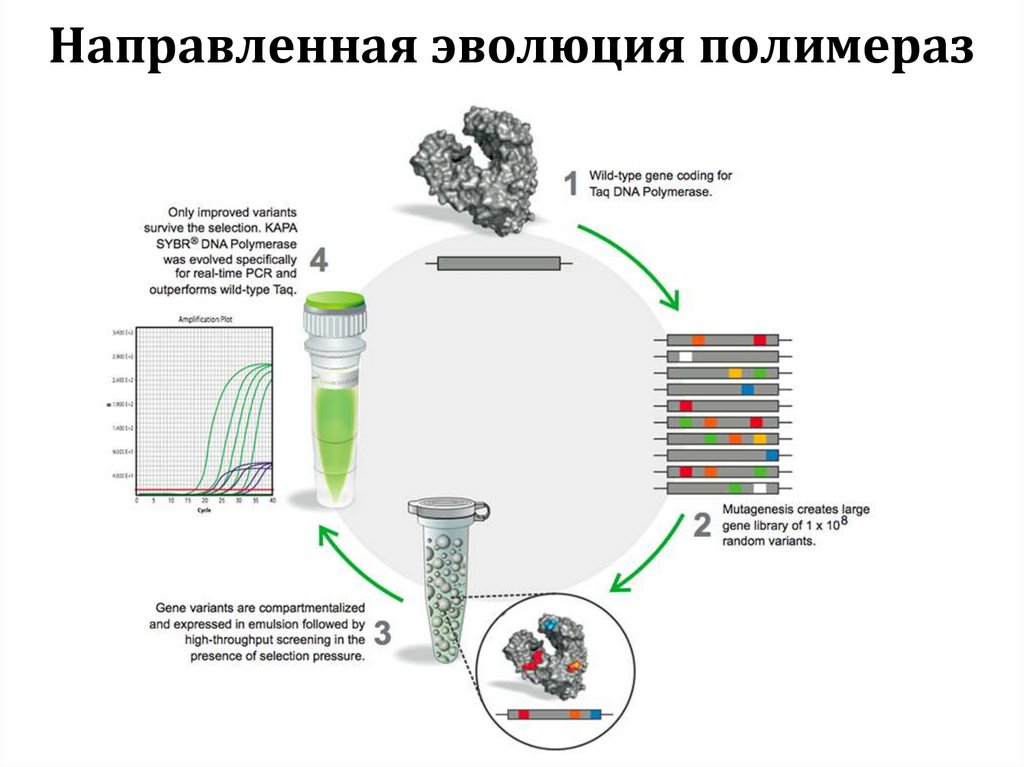
Направленная эволюция полимераз93. Полимеразная цепная реакция (ПЦР)
94. История открытия
Апрель 1983 г – идея ПЦРДекабрь 1983 г – осуществление ПЦР
1993 г – Нобелевская премия по химии
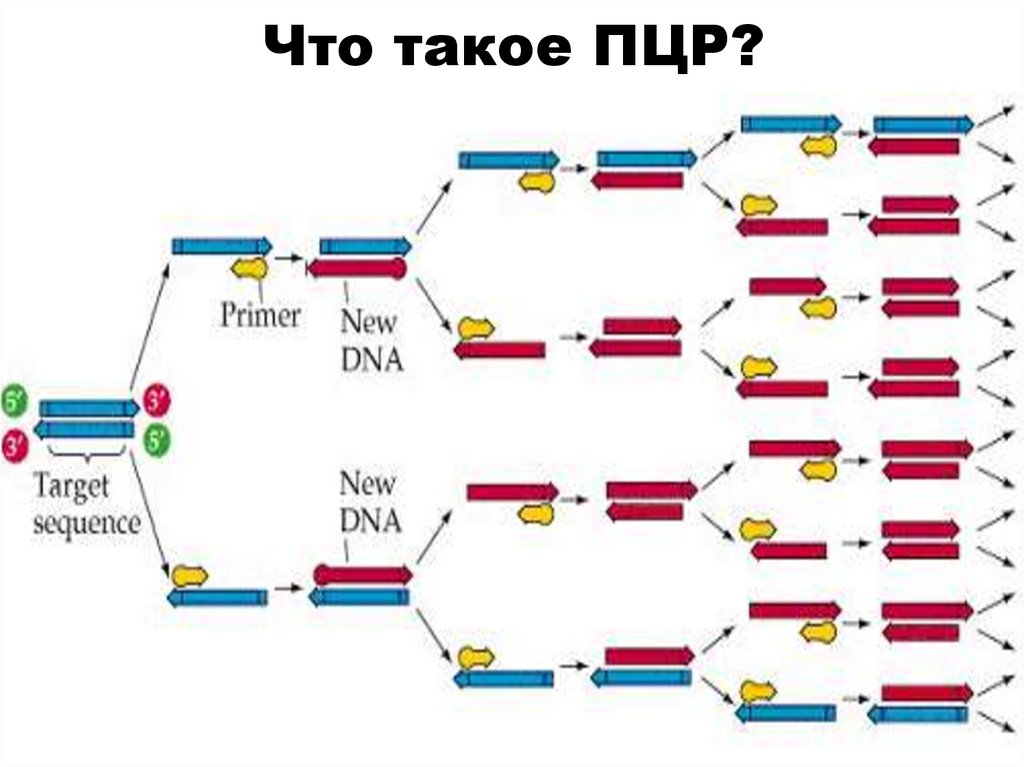
95. Что такое ПЦР?
По сути, это упрощенная версиярепликации бактерий, при
которой возрастает количество
копий специфической
последовательности ДНК. Их
называют ПЦР-продуктами или
ампликонами.
96. Что такое ПЦР?
97. Рабочий цикл ПЦР
Temperature, 0C>90 0C
денатурация
50 – 65 0C
отжиг
72 - 75 0C
амплификация
>90 0C
денатурация
98. Сравнение ПЦР и репликации in vivo (1)
Общие процессыПЦР
Репликация
Назначение
Для получения миллионов
копий ДНК размером до 15
– 20 kb
Для получения одной копии ДНК
размером несколько Mb. Геном E. coli
– 4.6 Mb
Денатурация:
разделение
цепей дцДНК
Для разрыва водородных
связей изпользуется
нагревание матрицы ДНК
до Т > 94 градусов
Фактор начала репликации разделяет
цепи ДНК в ориджине и вовлекает
другие необходимые факторы в
репликацию. Цепи ДНК остаются
связанными перед репликативной
вилкой.
Топоизомераза постепенно
расплетает спираль ДНК перед
репликативной вилкой. Геликаза
разрушает водородные связи между
цепями ДНК
SSB-белки предотвращают
спаривание ДНК
Отжиг праймеров
на оцДНК
ДНК праймеры
сконструированы
комплементарно матрице.
Праймеры химически
синтезированы
РНК-праймеры (затравки)
производятся при помощи РНКпраймазы, которая считывает
матрицу ДНК
99. Сравнение ПЦР и репликации in vivo (2)
Общие процессыПЦР
Репликация
Построение
второй цепи ДНК
при помощи ДНКполимеразы,
которая
добавляет
нуклеотиды в 5’3’ направлении
Термостабильная ДНКполимераза «удлиняет»
ДНК-праймер
Нуклеотиды,
использующиеся в
построении цепи,
добавляются в реакцию
исследователем
ДНК-полимераза III строит
лидирующую цепь непрерывно, а
отстающую цепь ДНК между РНКпраймерами в 5’-3’ направлении.
Образуются фрагменты Оказаки.
РНК-нуклеотиды праймеров
заменяются на ДНК при помощи ДНКполимеразы I .
Фосфодиэфирная связь сахарофосфатного остова соединяется при
помощи лигазы
Нуклеотиды, используемые в
репликации, синтезируются в
результате специальных
метаболических реакций в клетке.
Завершение
репликации
В момент, когда
полимераза достраивает
участок ДНК,
ограниченный
праймерами
Заканчивается в момент
изготовления полной копии ДНК,
которая переходит в другую клетку
при делении.
100. Компоненты реакции ПЦР
Компоненты реакцииКонцентрации
стоковых растворов
ДНК-матрица (DNA template)
----
dNTPs (dATP, dCTP, dTTP,
dGTP)
MgCl2
Буфер для ПЦР (PCR buffer)
Прямой праймер (Forward
primer)
Обратный праймер (Reverse
primer)
ДНК-полимераза
Вода MQ
Минеральное масло
Конечная концентрация на
одну реакцию
50 – 500 нг геномной ДНК
50 пк – 50 нг плазмидной ДНК
10 мМ
0.2 мМ
50 мМ
10х
1 – 6 мМ
1х
100 мкМ
0.1 – 1 мкМ
100 мкМ
0.1 – 1 мкМ
В зависимости от
типа полимеразы и ее
производителя
Не более 1 – 2 мкл (до 2.5
ед.)
До конечного объема
(обычно от 10 – 100 мкл)
В случае использования амплификатора без
нагрева крышки
-----
Master Mix (MM)
101. Компоненты реакции ПЦР
Термостабильные ДНК-полимеразы:Для рутинного проведения ПЦР обычно применяется 0.5 – 2.5 единицы фермента на 20 – 50
мкл реакционной смеси. Таким образом, стандартная реакция ПЦР содержит от 4×1010 до
2×1011 молекул фермента. Учитывая, что средняя эффективность трансформации
составляет 0.7, а количество молекул ампликонов может превышать 100 раз, количество
молекул ПЦР-продукта может составить от 1.4×1012 до 2×1013 молекул ДНК в пробирке
(Gelfand and White, 1990). Выбираются под определенные задачи!
Праймеры:
Стандартная реакция ПЦР содержит 0.1 – 0.5 мкМ каждого праймера, что соответствует от
6×1012 до 3×1013 молекул на реакцию. Данного количества обычно хватает на 30 циклов
для амплификации фрагмента ДНК длиной 1-kb. Высокая концентрация способствует
неправильному спариванию праймеров и амплификации неспецифического продукта.
Дизайн праймеров осуществляется под конкретную задачу. Праймеры синтезируются
химическим методом, и затем очищаются от побочных продуктов путем детанурирующего
ПААГ-гель электрофореза и/или ВЭЖХ (NENSORB, NEN Life Science Products).
Матрица:
Может представлять собой молекулы оцДНК, дцДНК, РНК. Кольцевая ДНК
амплифициреутся сложнее, чем линейная ДНК. Хотя размер ДНК для проведения реакции
не критичен, многими специалистами рекомендуется линеаризовать кольцевые молекулы
матрицы при длине более 10-kb. Стандартная реакция должна содержать несколько тысяч
копий целевой ДНК. В случае gDNA млекопитающих до 1 мкг ДНК, что соответствует ~
3×105 молекул матрицы. Типичное количество дрожжевой, бактериальной, плазмидной
ДНК составляет 10 нг, 1 нг, 1 пг соответственно. Важно качество ДНК!
102. Компоненты реакции ПЦР
Дезоксинуклеотидтрифосфаты (dNTPs):Стандартная реакция ПЦР содержит эквимолярное количество dATP, dTTP, dCTP, dGTP. Для
Taq-полимеразы обычно 1 реакция содержит 200 – 250 мкМ dNTP при содержании ионов
Mg2+ 1,5 mM. Это позволяет наработать ~ 6 – 6.5 мкг целевого фрагмента ДНК в
реакционном объеме 50 мкл, даже для мультиплексной ПЦР. Пороговые концентрации dNTP
– 20 мкМ и 4 мМ. Многие производители (Roche, QIAQEN. Boehringer-Indelheim, GE
Healthcare) используют ВЭЖХ для ультра очистки реагента от свободных тетра- и
пирофосфатов, которые ингибируют ПЦР. Стоковый раствор dNTP чувствителен к
«замораживанию – оттаиванию»
Катионы двухвалентных металлов:
Все термостабильные ДНК полимеразы для своей активности требуют присутствия
свободных ионов Mg2+ . Однако некоторые полимеразы также способны работать, но с
меньшей эффективностью и точностью, на ионах Mn2+ и Co2+. Ионы кальция неэффективны.
Когда ионов магния слишком мало, праймер отжигается на матрице неэффективно. Когда
ионов магния слишком много, то образуются прочные комплексы ДНК с праймерами, а
также праймеров друг с другом. Рутинная концентрация Mg2+ составляет 1,5 mM, однако,
она может быть неоптимальной. Обычно проводится подбор оптимальной концентрации
ионов магния эмпирически. Насколько это возможно, при пробоподготовке используйте
минимальное количество хелатирующих соединений, таких как ЭДТА, PO43 pH и моновалентные катионы: Для проведения реакции используется буферный
раствор Трис-HCl с концентрацией 10 – 66 мМ и рН 8.3-8.8. Стандартная реакция ПЦР
содержит 50 – 60 мМ KCl. Иногда, повышение концентрации KCl до 70 – 100 мМ
приводит к повышению выхода фрагментов ДНК менее 500 bp
103.
104. Подбор концентрации ионов Mg2+
105. Эффективность ПЦР
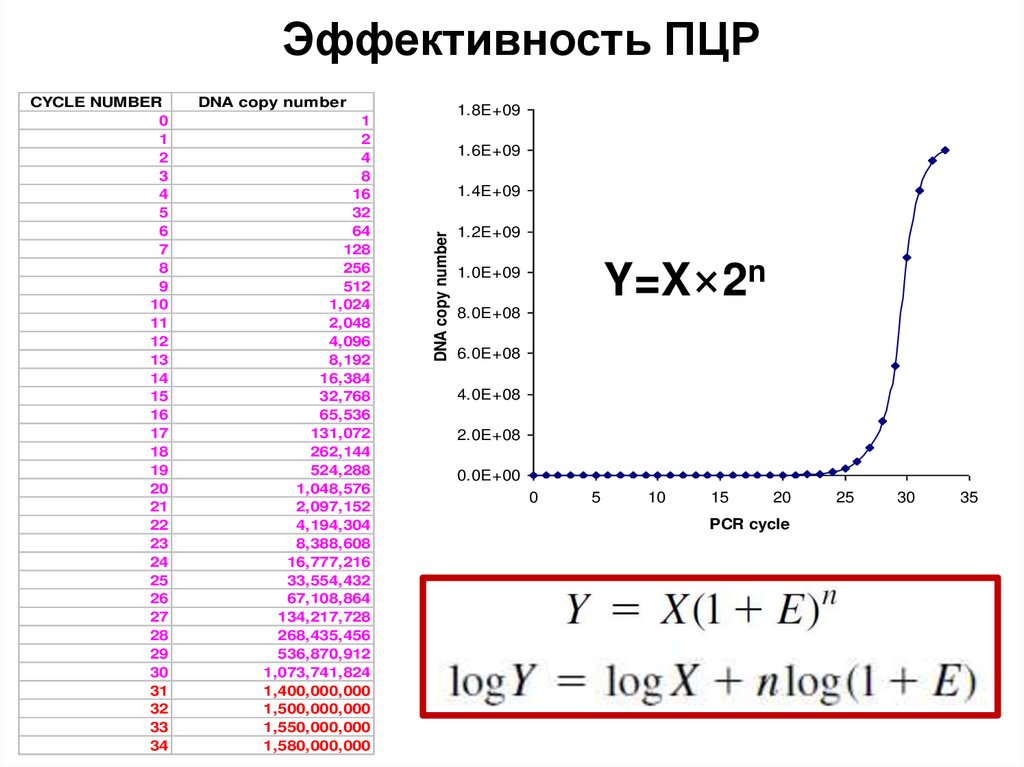
DNA copy number1
2
4
8
16
32
64
128
256
512
1,024
2,048
4,096
8,192
16,384
32,768
65,536
131,072
262,144
524,288
1,048,576
2,097,152
4,194,304
8,388,608
16,777,216
33,554,432
67,108,864
134,217,728
268,435,456
536,870,912
1,073,741,824
1,400,000,000
1,500,000,000
1,550,000,000
1,580,000,000
1.8E+09
1.6E+09
1.4E+09
DNA copy number
CYCLE NUMBER
0
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
1.2E+09
Y=X×2n
1.0E+09
8.0E+08
6.0E+08
4.0E+08
2.0E+08
0.0E+00
0
5
10
15
20
PCR cycle
25
30
35
106.
107. Компоненты реакции ПЦР
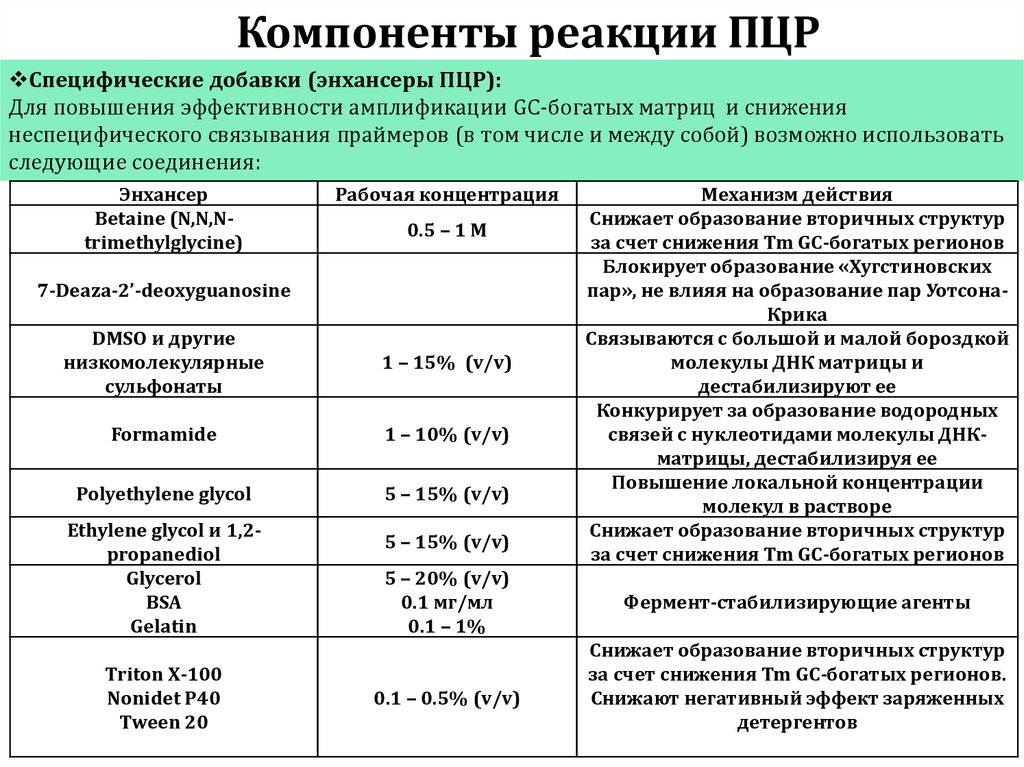
Специфические добавки (энхансеры ПЦР):Для повышения эффективности амплификации GC-богатых матриц и снижения
неспецифического связывания праймеров (в том числе и между собой) возможно использовать
следующие соединения:
Энхансер
Betaine (N,N,Ntrimethylglycine)
Рабочая концентрация
0.5 – 1 M
7-Deaza-2’-deoxyguanosine
DMSO и другие
низкомолекулярные
сульфонаты
1 – 15% (v/v)
Formamide
1 – 10% (v/v)
Polyethylene glycol
5 – 15% (v/v)
Ethylene glycol и 1,2propanediol
Glycerol
BSA
Gelatin
Triton X-100
Nonidet P40
Tween 20
5 – 15% (v/v)
5 – 20% (v/v)
0.1 мг/мл
0.1 – 1%
0.1 – 0.5% (v/v)
Механизм действия
Снижает образование вторичных структур
за счет снижения Tm GC-богатых регионов
Блокирует образование «Хугстиновских
пар», не влияя на образование пар УотсонаКрика
Связываются с большой и малой бороздкой
молекулы ДНК матрицы и
дестабилизируют ее
Конкурирует за образование водородных
связей с нуклеотидами молекулы ДНКматрицы, дестабилизируя ее
Повышение локальной концентрации
молекул в растворе
Снижает образование вторичных структур
за счет снижения Tm GC-богатых регионов
Фермент-стабилизирующие агенты
Снижает образование вторичных структур
за счет снижения Tm GC-богатых регионов.
Снижают негативный эффект заряженных
детергентов
108. Компоненты реакции ПЦР
Ингибиторы ПЦР:Практически любое соединение, присутствующее в реакции ПЦР является
ингибитором, если оно в избытке!!! Самые частые контаминанты ПЦР-реакции это:
протеиназа К (деградация ДНК-полимеразы), фенол, ЭДТА. Не приветствуется
концентрация ЭДТА выше 0.1 мМ в растворе!!! Также серьезные проблемы могут доставить
анионные детергенты, гепарин, полианионы (такие как спермидин), гемоглобин,
красители, используемые для электрофореза, такие как бромфеноловый синий и ксилен
цианол. Часто, большинство ингибиторов, поступают вместе с матрицей ДНК.
Следовательно, матрица должна быть хорошо очищена от различных примесей.
Контаминация.
В идеале, все стадии пробоподготовки и проведения ПЦР должны проводиться в
отдельных кабинетах. Наилучшие условия приготовления ПЦР-смеси – в условиях
ламинарного потока с UV-обработкой. На каждой этапе используется отдельный
комплект оборудования и реагентов. Они ни в коем случае не переносятся «с одного
стола на другой». В лаборатории, где проводят ПЦР передвижения должны быть
ограничены. Персонал работает в перчатках, масках, шапочках для снижения риска
контаминации. ПЦР-реагенты никогда не используются для других целей. В ПЦР
лабораториях используется одноразовый пластик соответствующей квалификации
(PCR grade). Для постановки реакции ПЦР также используется вода соответствующей
квалификации.
Контаминированные поверхности требуют соответствующей обработки (гипохлорид
натрия и т.д.), а пространство лаборатории подвергается UV-обработке. Для
предотвращения контаминации одного ПЦР-продукта другим можно использовать
урацилглигозилазу (Ung)
109. Рабочая программа ПЦР
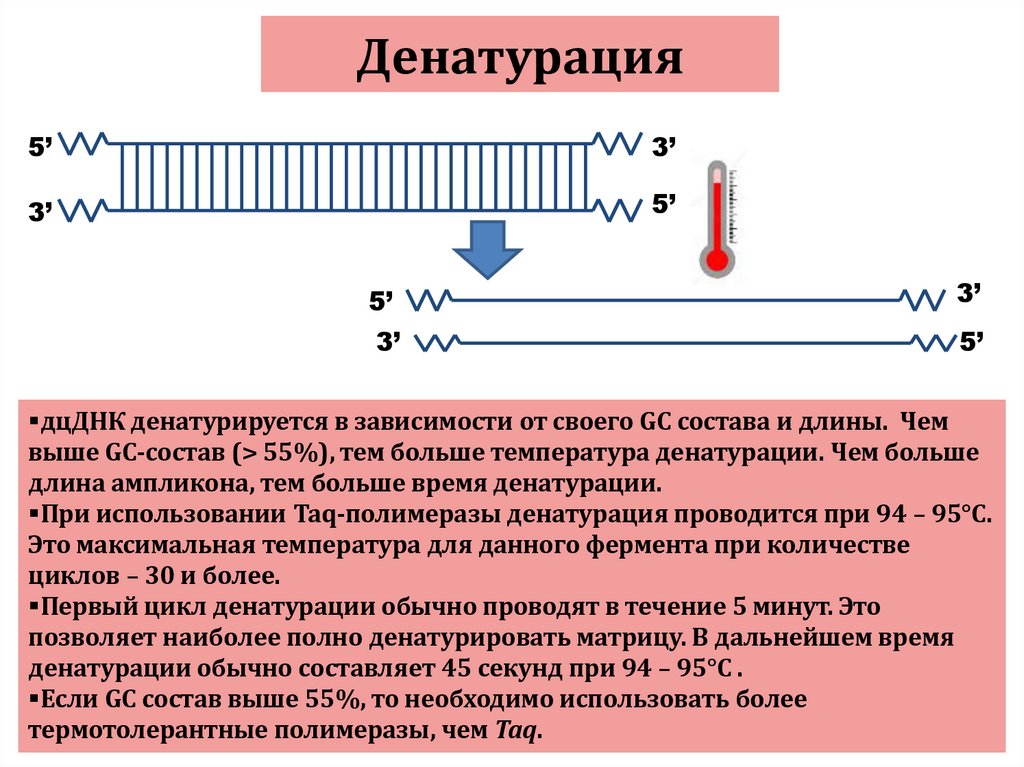
110. Денатурация
5’3’
3’
5’
5’
3’
3’
5’
дцДНК денатурируется в зависимости от своего GC состава и длины. Чем
выше GC-состав (> 55%), тем больше температура денатурации. Чем больше
длина ампликона, тем больше время денатурации.
При использовании Taq-полимеразы денатурация проводится при 94 – 95°С.
Это максимальная температура для данного фермента при количестве
циклов – 30 и более.
Первый цикл денатурации обычно проводят в течение 5 минут. Это
позволяет наиболее полно денатурировать матрицу. В дальнейшем время
денатурации обычно составляет 45 секунд при 94 – 95°С .
Если GC состав выше 55%, то необходимо использовать более
термотолерантные полимеразы, чем Taq.
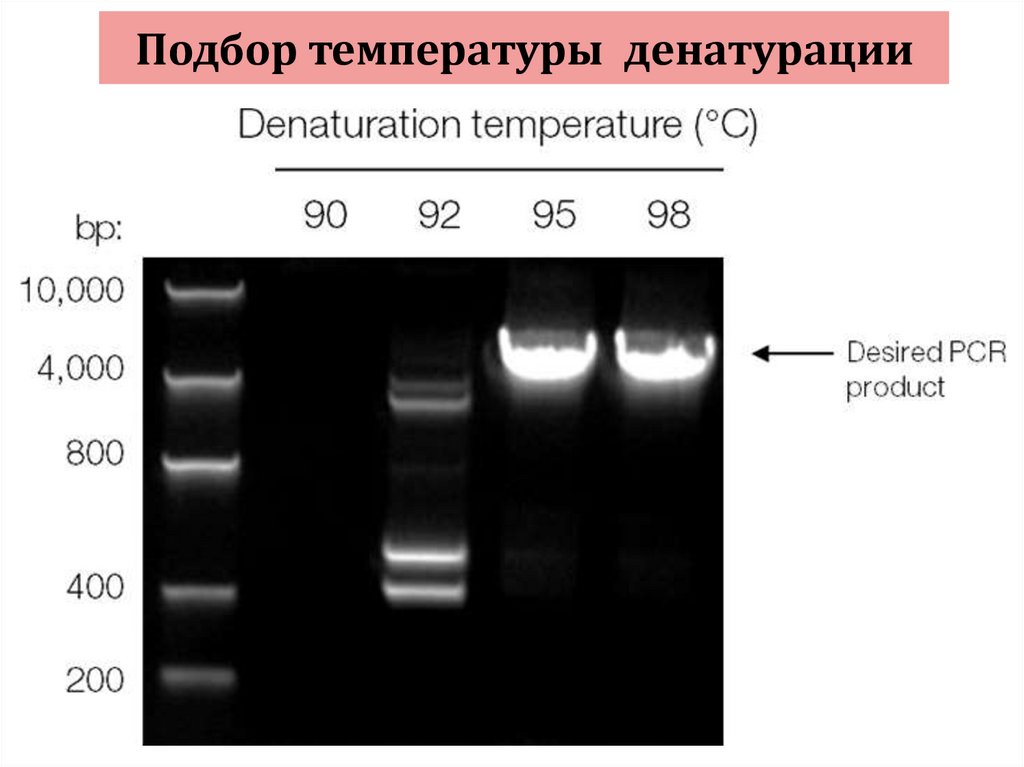
111. Расчет оптимальной температуры денатурации ампликона
Td=Tm+3-40С112. Подбор температуры денатурации
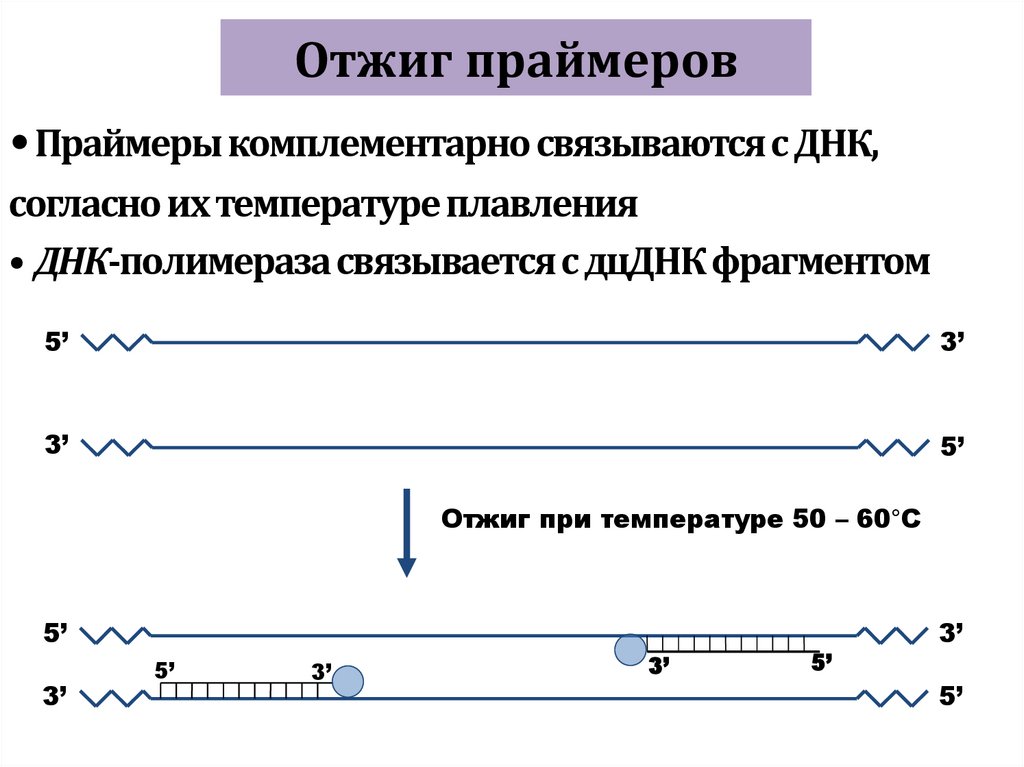
113. Отжиг праймеров
• Праймеры комплементарно связываются с ДНК,согласно их температуре плавления
• ДНК-полимеразасвязывается с дцДНК фрагментом
5’
3’
3’
5’
Отжиг при температуре 50 – 60°C
5’
3’
5’
3’
3’
5’
3’
5’
114. Температура плавления (T melting, Tm)
Под температурой плавления понимают температуру, прикоторой половина молекул гибридизована (находится в
двуцепочечном состоянии), а половина находится в растворе.
Данный параметр является характеристикой олигонуклеотида
для конкретных условий среды.
Tm (0С)= 2 (А+Т) + 4 (Г+Ц)
При длине праймера < 25 нуклеотидов
Tm (0С)=81.5+16,6lgI +0.41(%Г+%Ц)-500/N
Tm (0С)= 81.5+16.6(lg[K+]) +0.41(%Г+%Ц)-675/N
При длине праймера >25 нуклеотидов
Oligocalculator
115.
Температура отжига (T annealing, Ta)Под температурой плавления понимают температуру, соответствующего
режима в программе амплификации, оптимальную с точки зрения
эффективности, специфичности или других параметров ПЦР.
Еслитемпература будет ниже оптимальной, то праймербудет отжигаться
неспецифически, что приведет к получению побочных продуктов реакции.
Еслитемпература будет выше оптимальной, то продукта будет мало (зато
высокая специфичность), либо не будет вообще.
Зависит от температуры плавления:
Ta=Tm – 50С (по Ребрикову)
Ta=Tm – 100С (по Патрушеву)
Ta=Tm – 3-50С (Green and Sambrook)
Зависит от вида полимеразы:
Ta=Tm ± 2-100С
116.
Задача №1Определите температуры плавления и отжига
следующего олигонуклеотида:
5’ GGCATTTAGCTTAGGC 3’
Решение:
N=16: А=3, Т=5, Г=5, Ц=3
Tm= 2 (А+Т) + 4 (Г+Ц) = 2(3+5) + 4(5+3) = 480С
Ta = 48 – 5 = 430С
117.
Задача №2Определите температуры плавления и отжига следующего
олигонуклеотида:
5’ GGCATTTAGCTTAGGCTTAAGGCGGGAG 3’
Решение:
N=28: А=6, Т=7, Г=11, Ц=4, %Г=11*100/28 =
39.3, %Ц = 4*100/28 = 14.3
Tm (0С)= 81.5+0.41(%Г+%Ц)-675/N = 81.5 +
0.41(39.3+14.3) – 675/28 = 79.4
Ta = 79.4 – 10 = 69.40С
Tm= 2(6+7) + 4(11+4) = 860С
118.
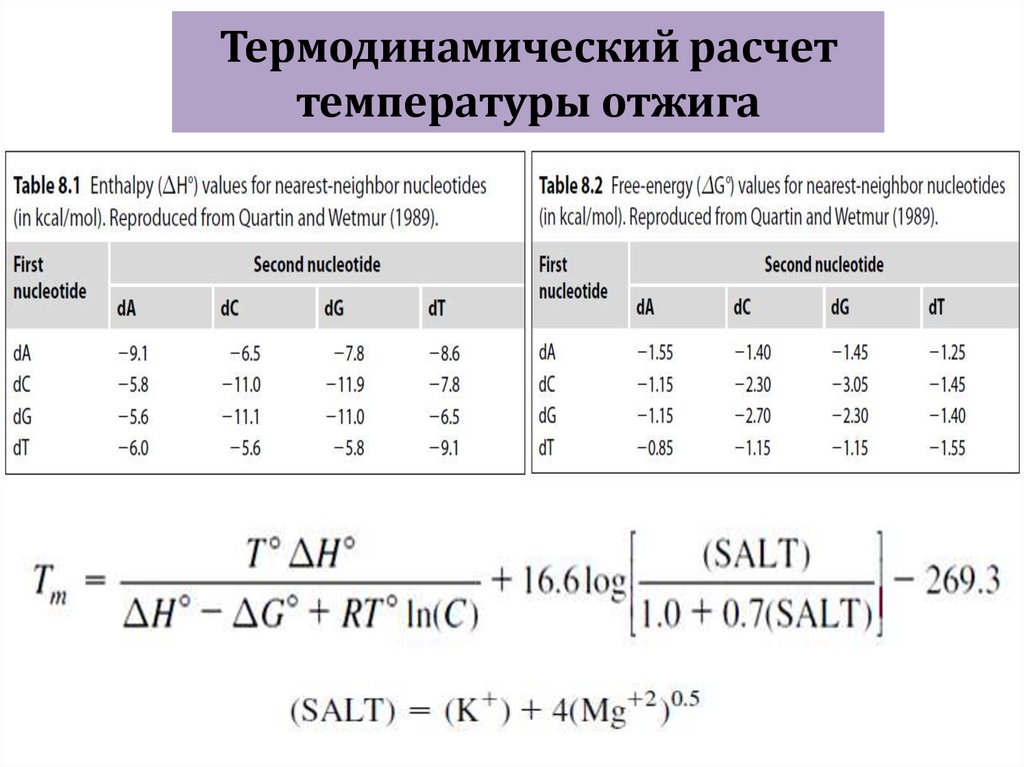
Термодинамический расчеттемпературы отжига
119.
Амплификация ПЦР-продуктаАмплификация (наработка) ПЦР-продукта или удлинение (экстензия) праймера
проводится при температурах, близких к оптимальной для ДНК-полимеразы. В случае
Taq-полимеразы – 72 – 78 °С. В первых двух циклах амплификация превышает
пределы, заданные парой праймеров. Начиная с 3 цикла, количество целевого
фрагмента ДНК растет с геометрической прогрессией, а количество «удлиненного»
фрагмента – с арифметической.
Стандартная скорость амплификации 1000 нуклеотидов/сек. Как правило, после
последнего цикла многие исследователи включают стадию финальной
амплификации, которая по продолжительности составляет 3 – 10 циклов
амплификации для полной достройки фрагмента ДНК.
Количество циклов ПЦР (денатурация+отжиг+амплификация) зависти от количества
копий матрицы в растворе, эффективности отжига праймера и эффективности
амплификации. По крайней мере 25 циклов необходимо для достижения
приемлемого количества ПЦР-продукта, имея исходно 1 копию целевой
последовательности в составе gДНК млекопитающих
120.
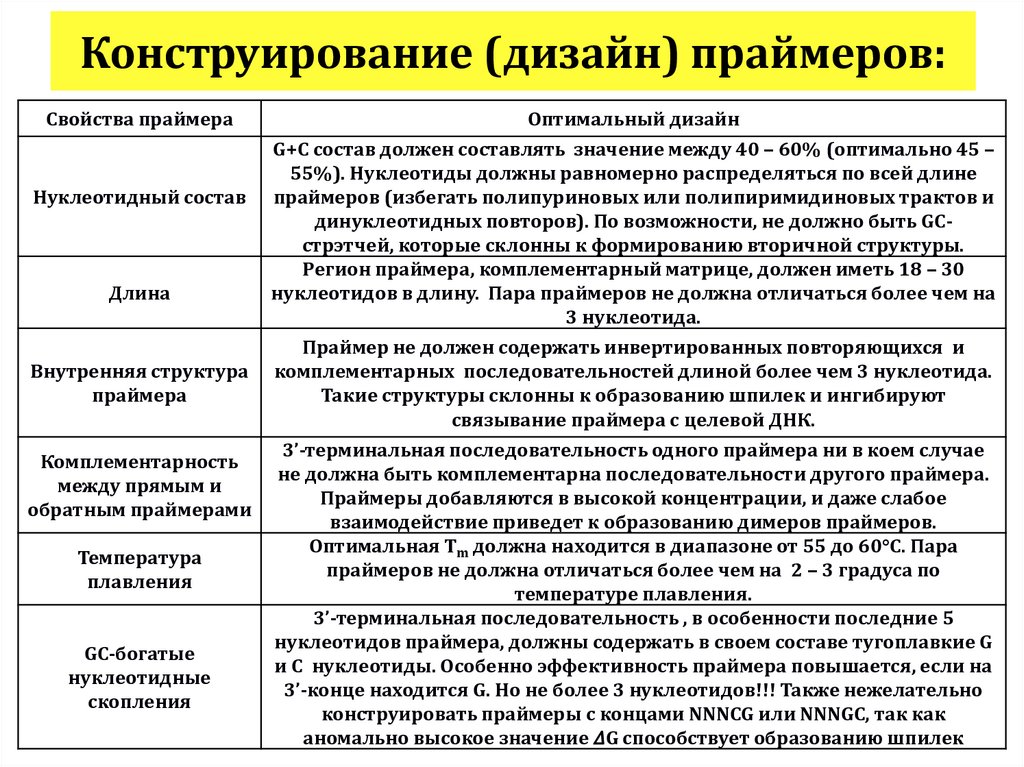
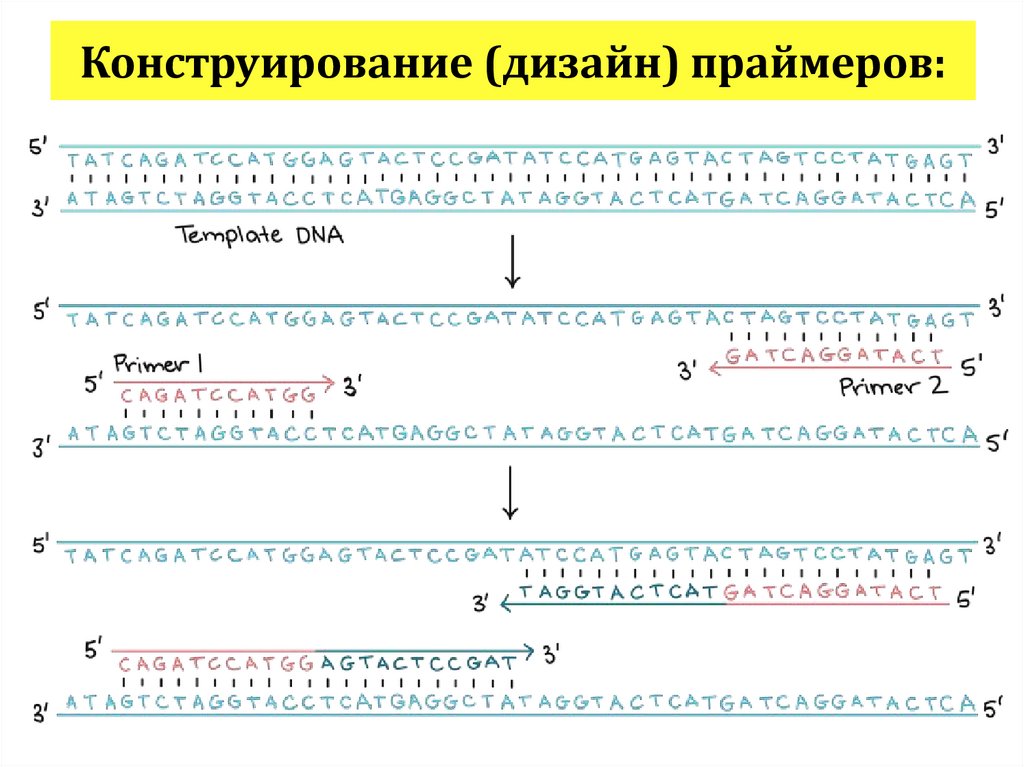
121. Конструирование (дизайн) праймеров:
Свойства праймераНуклеотидный состав
Длина
Внутренняя структура
праймера
Комплементарность
между прямым и
обратным праймерами
Температура
плавления
GC-богатые
нуклеотидные
скопления
Оптимальный дизайн
G+C состав должен составлять значение между 40 – 60% (оптимально 45 –
55%). Нуклеотиды должны равномерно распределяться по всей длине
праймеров (избегать полипуриновых или полипиримидиновых трактов и
динуклеотидных повторов). По возможности, не должно быть GCстрэтчей, которые склонны к формированию вторичной структуры.
Регион праймера, комплементарный матрице, должен иметь 18 – 30
нуклеотидов в длину. Пара праймеров не должна отличаться более чем на
3 нуклеотида.
Праймер не должен содержать инвертированных повторяющихся и
комплементарных последовательностей длиной более чем 3 нуклеотида.
Такие структуры склонны к образованию шпилек и ингибируют
связывание праймера с целевой ДНК.
3’-терминальная последовательность одного праймера ни в коем случае
не должна быть комплементарна последовательности другого праймера.
Праймеры добавляются в высокой концентрации, и даже слабое
взаимодействие приведет к образованию димеров праймеров.
Оптимальная Tm должна находится в диапазоне от 55 до 60°С. Пара
праймеров не должна отличаться более чем на 2 – 3 градуса по
температуре плавления.
3’-терминальная последовательность , в особенности последние 5
нуклеотидов праймера, должны содержать в своем составе тугоплавкие G
и C нуклеотиды. Особенно эффективность праймера повышается, если на
3’-конце находится G. Но не более 3 нуклеотидов!!! Также нежелательно
конструировать праймеры с концами NNNCG или NNNGC, так как
аномально высокое значение





































































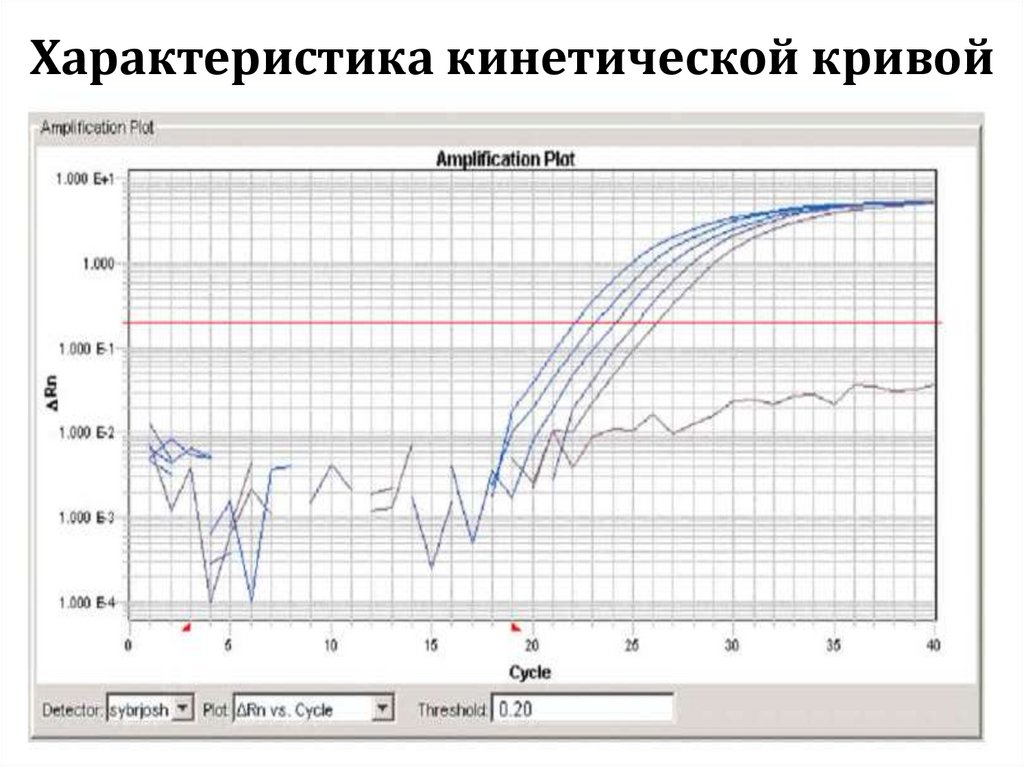
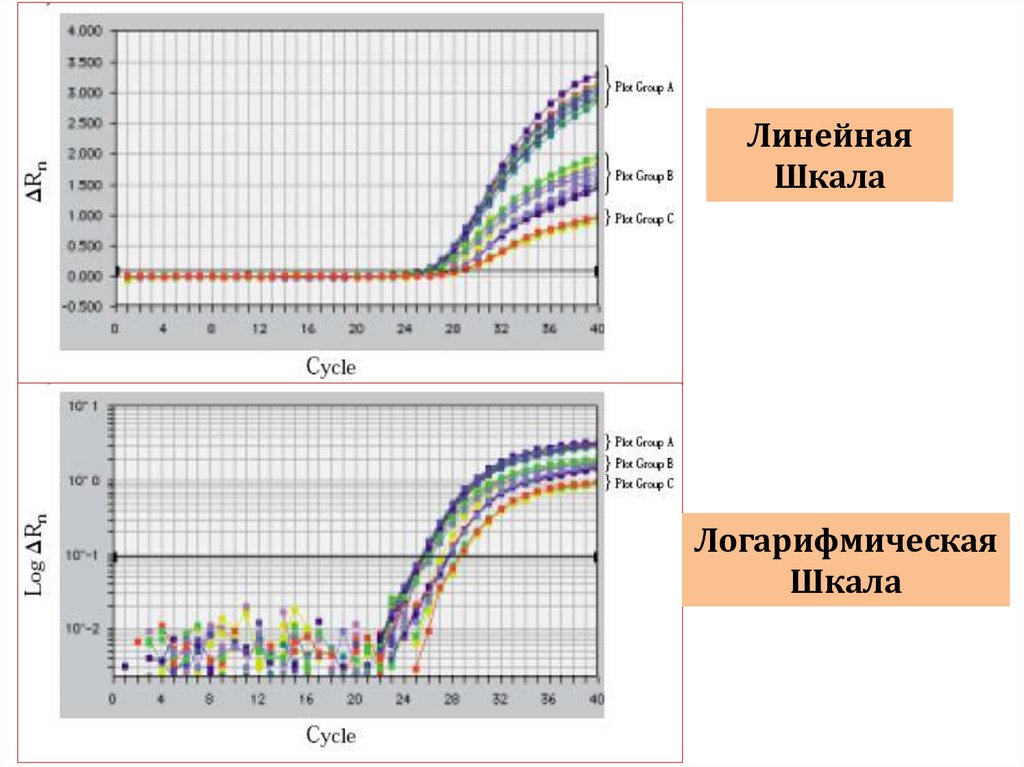
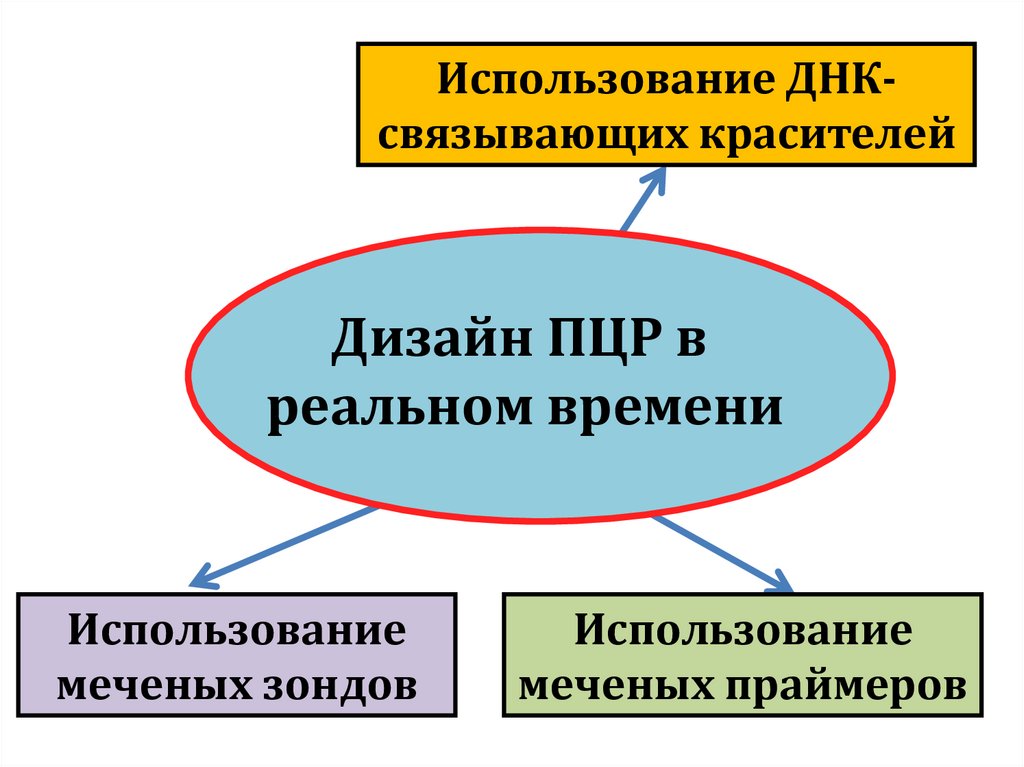


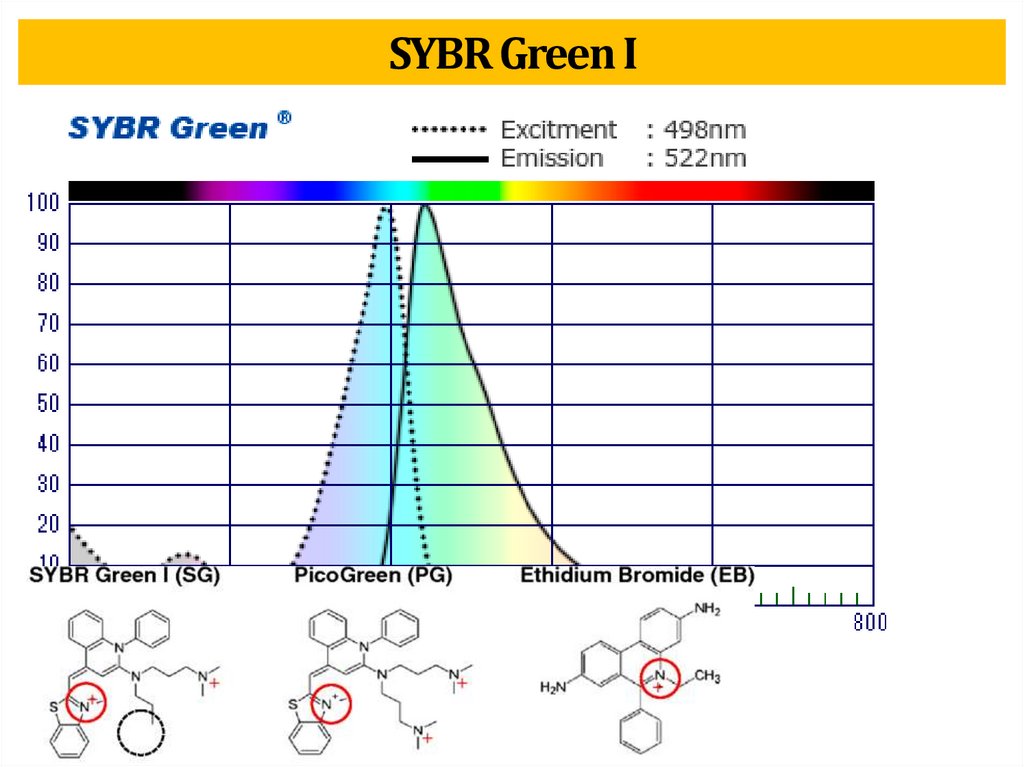
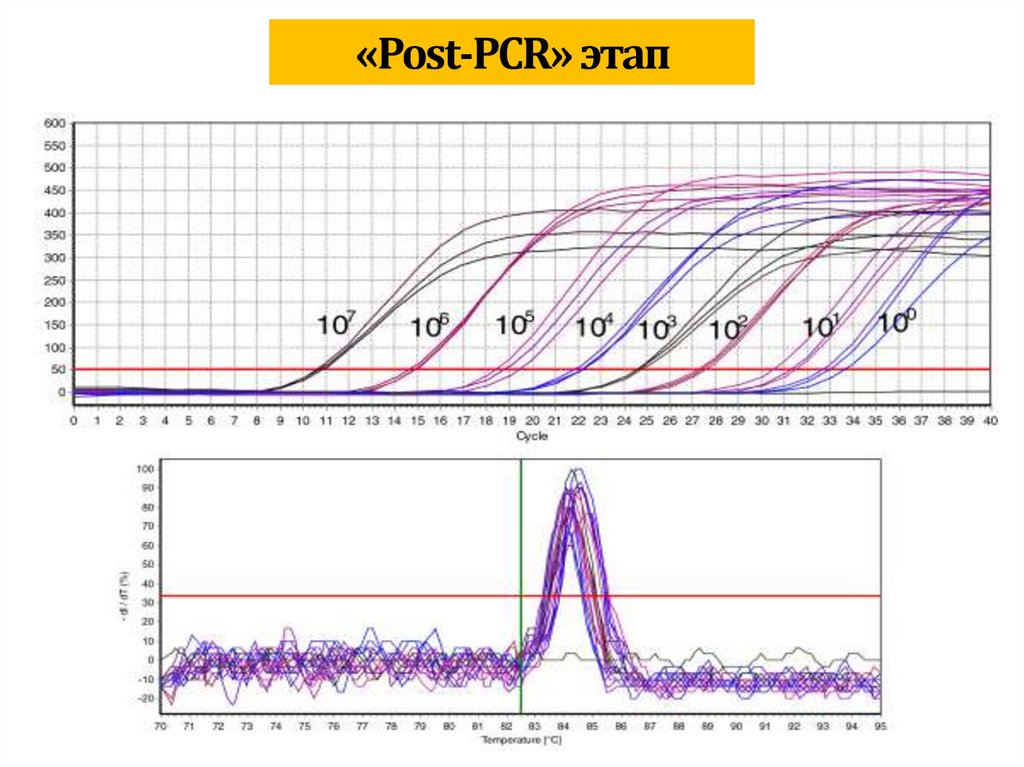

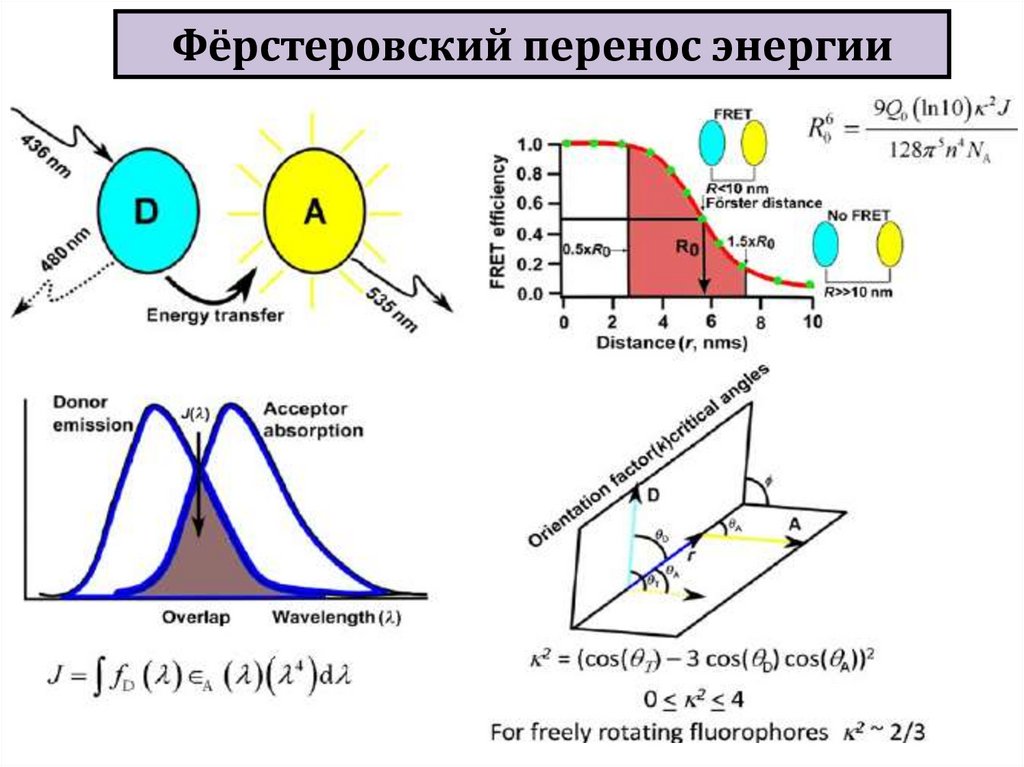
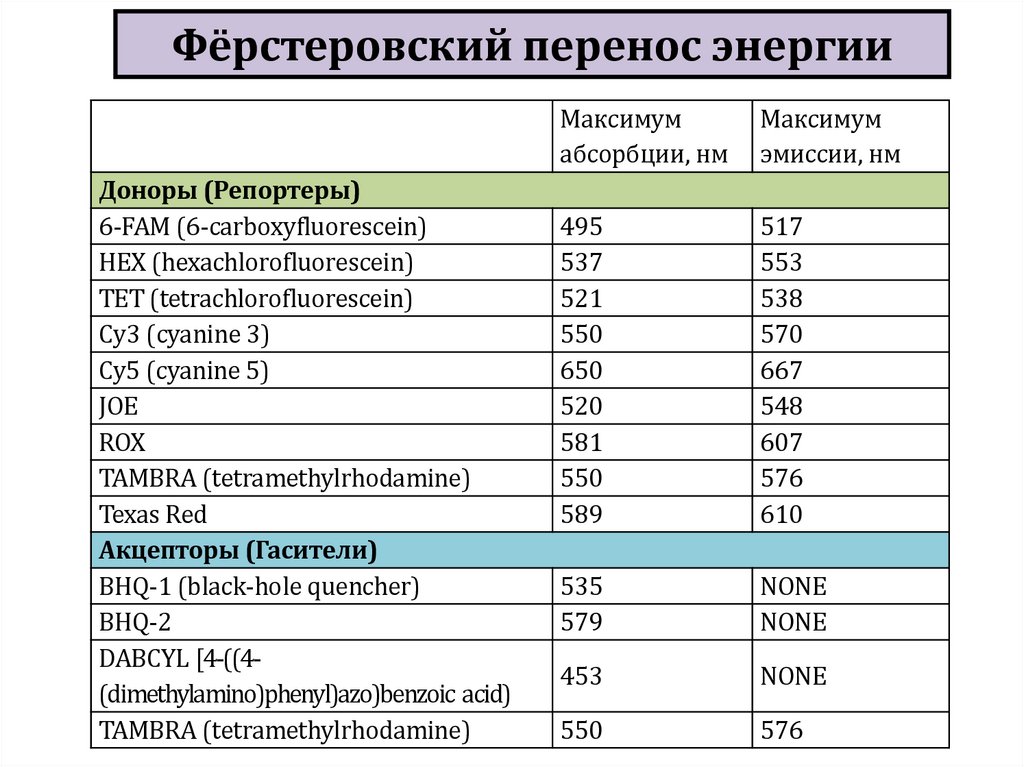
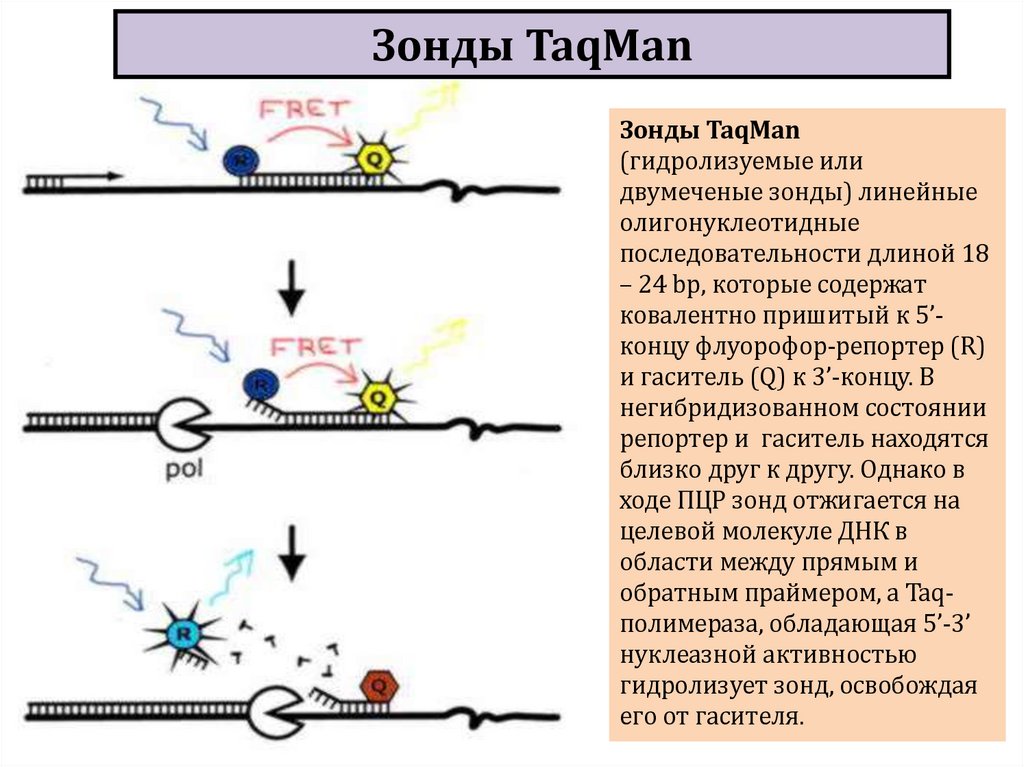

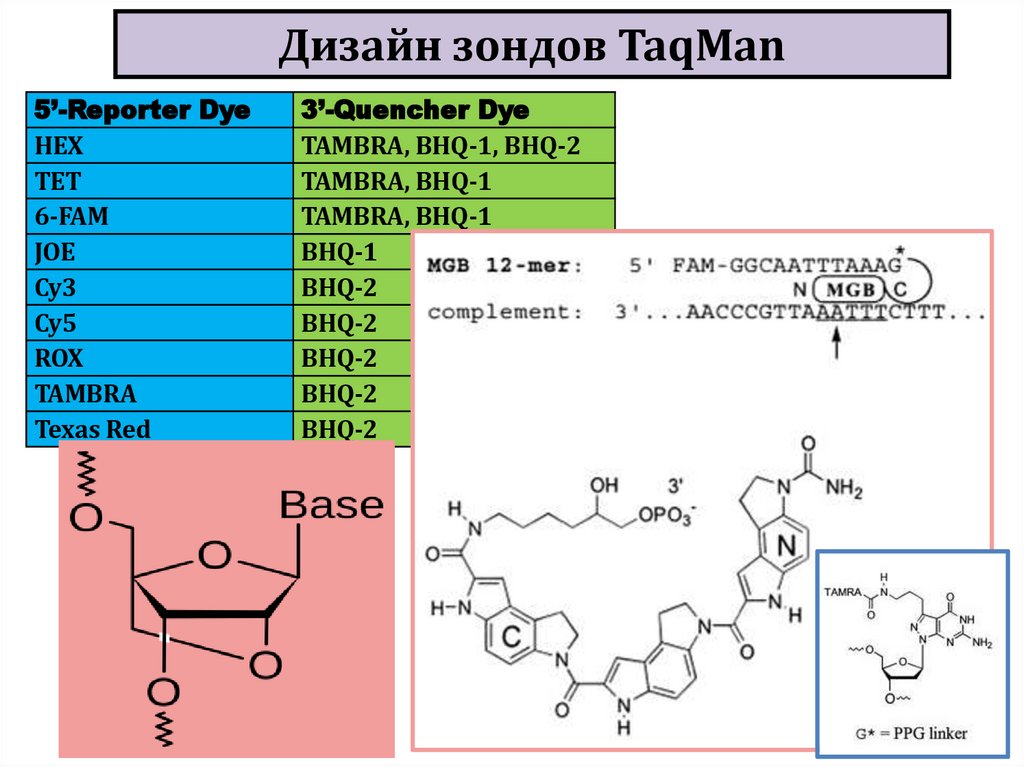
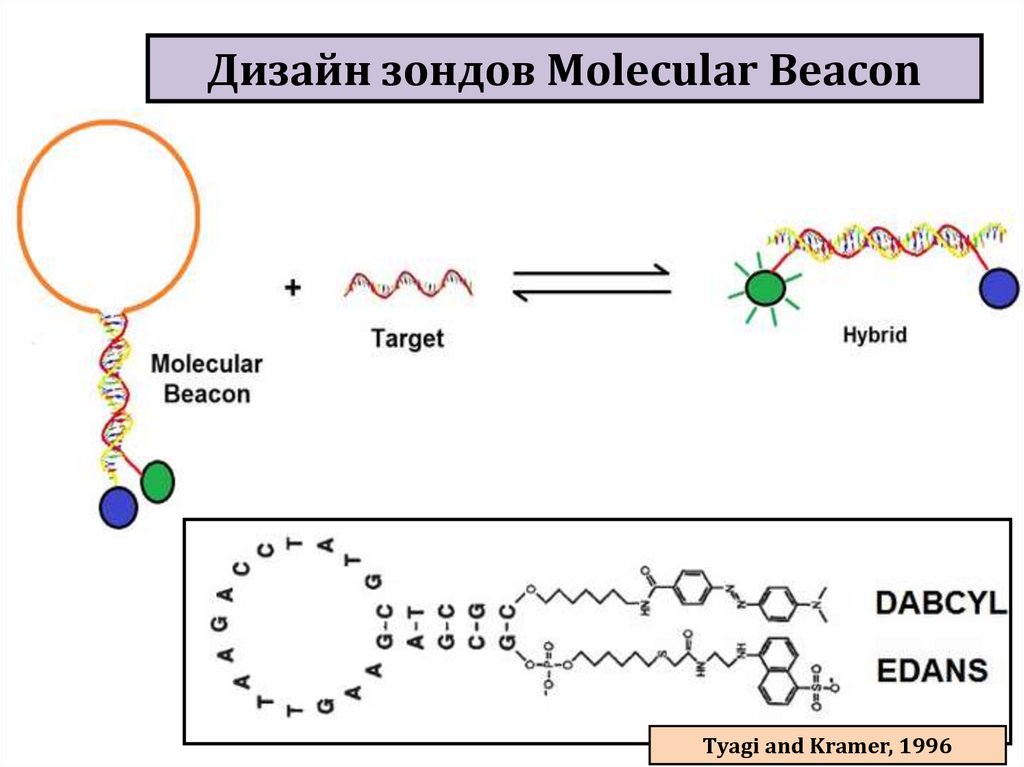

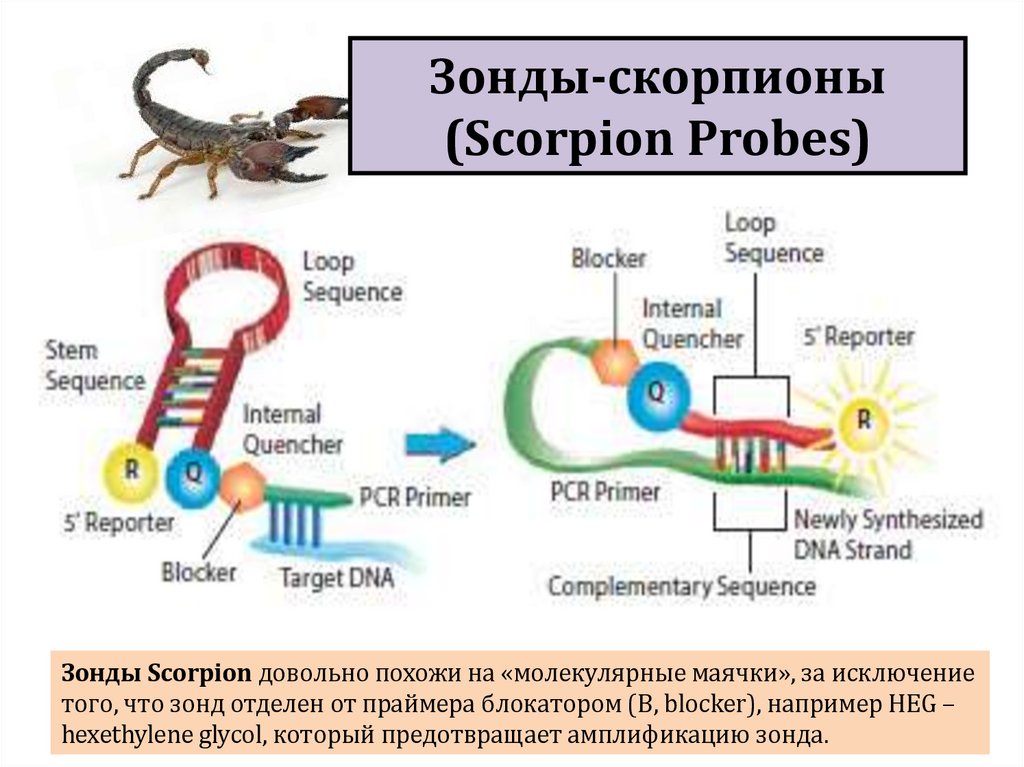
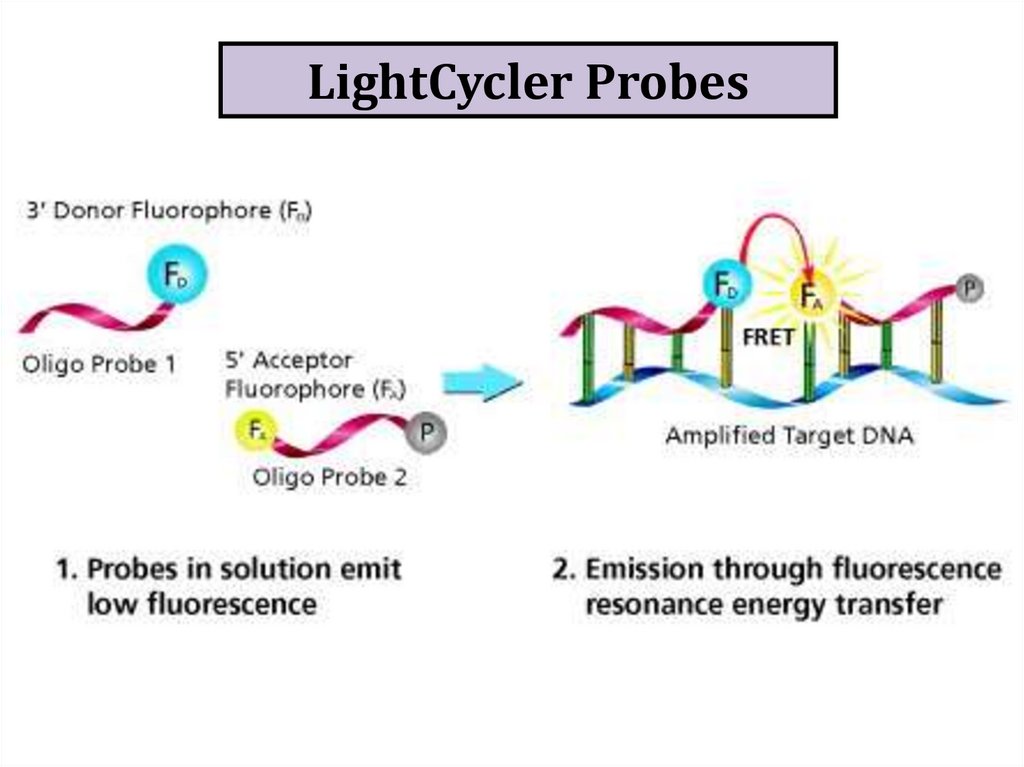
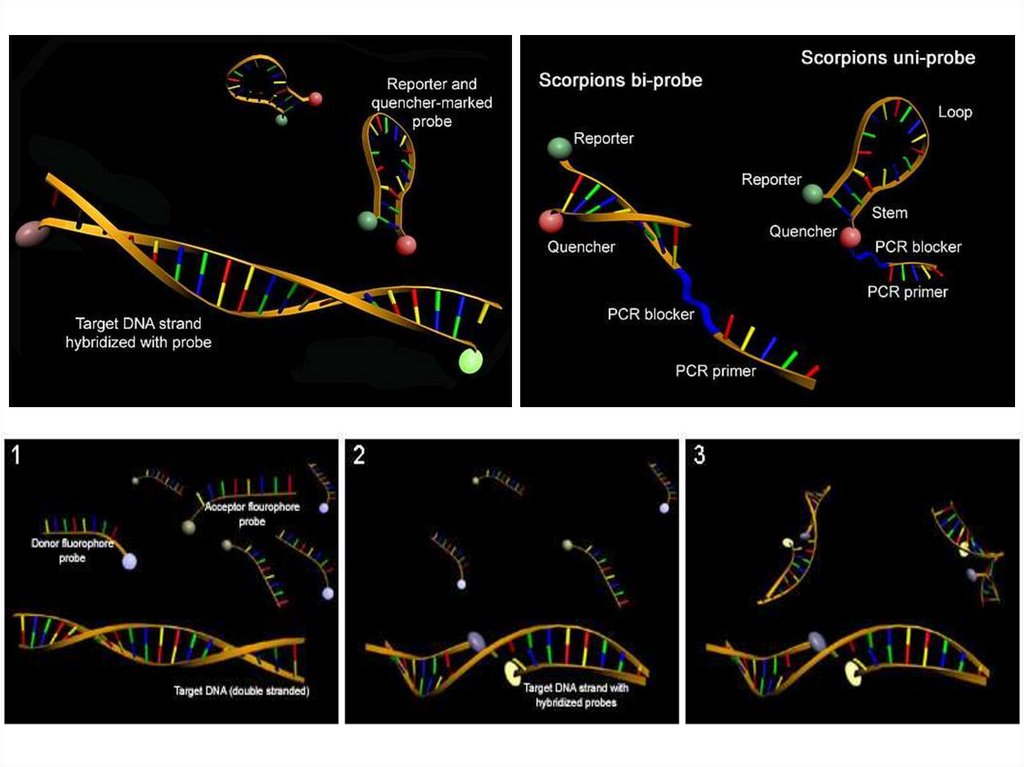

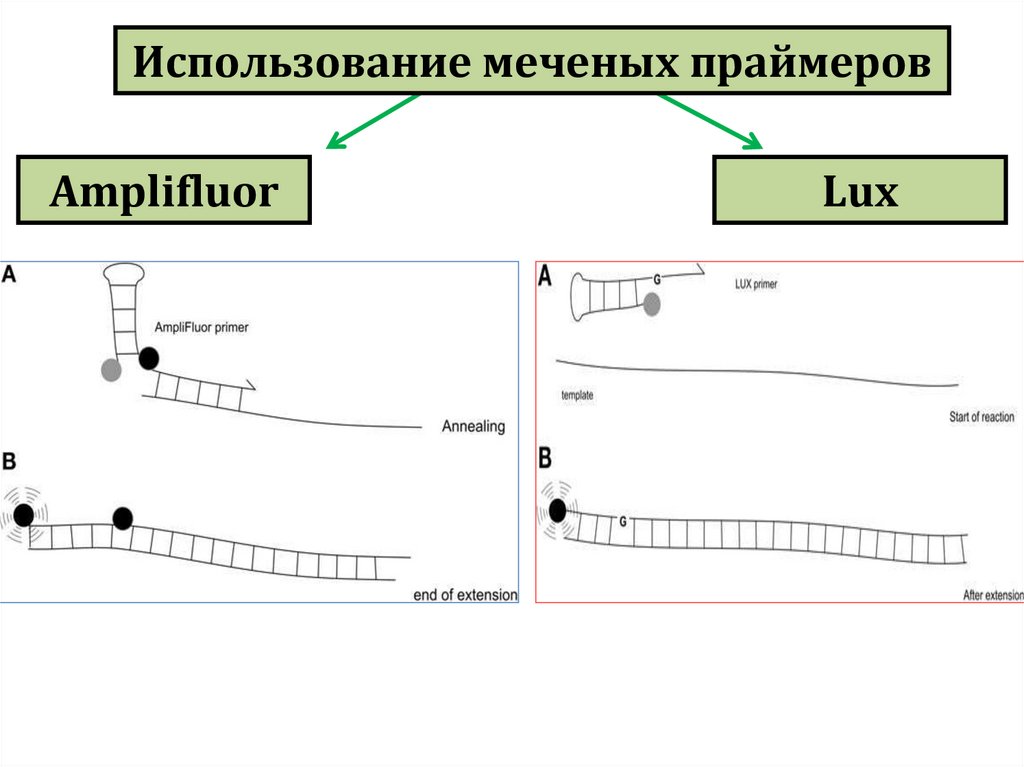

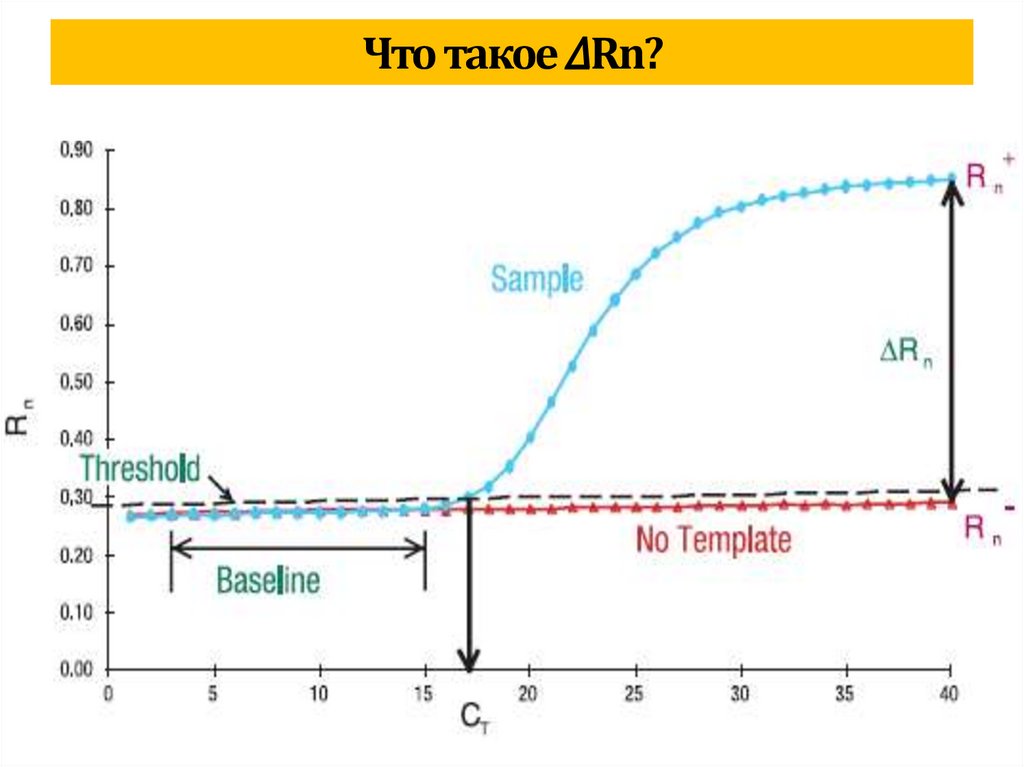
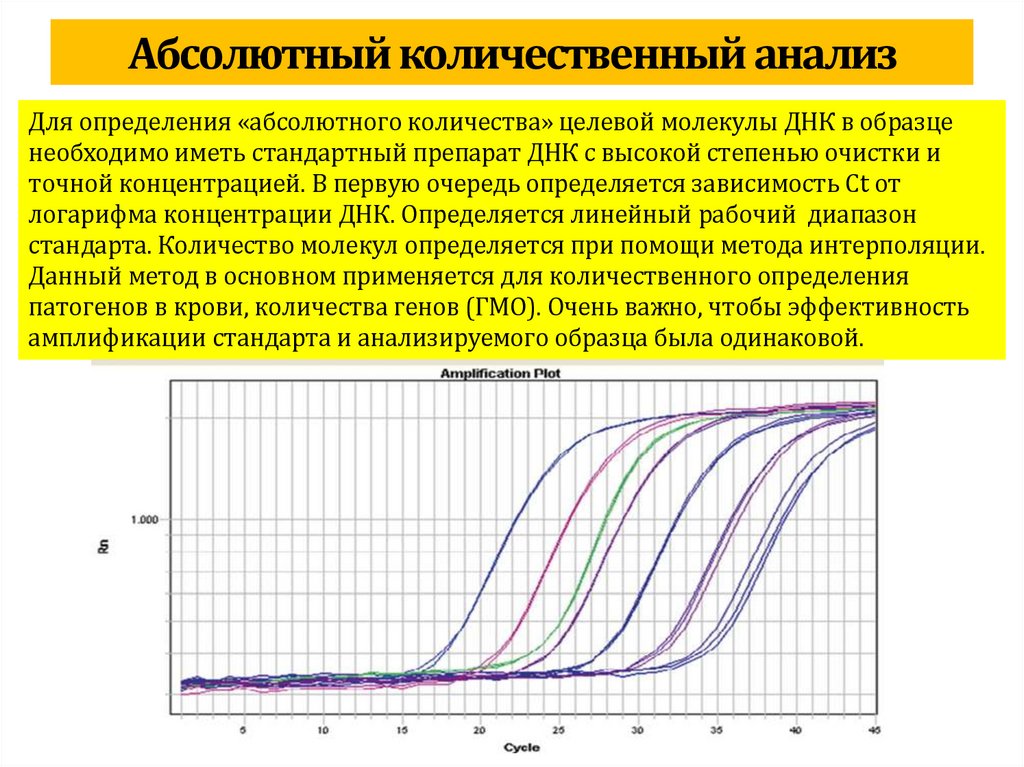
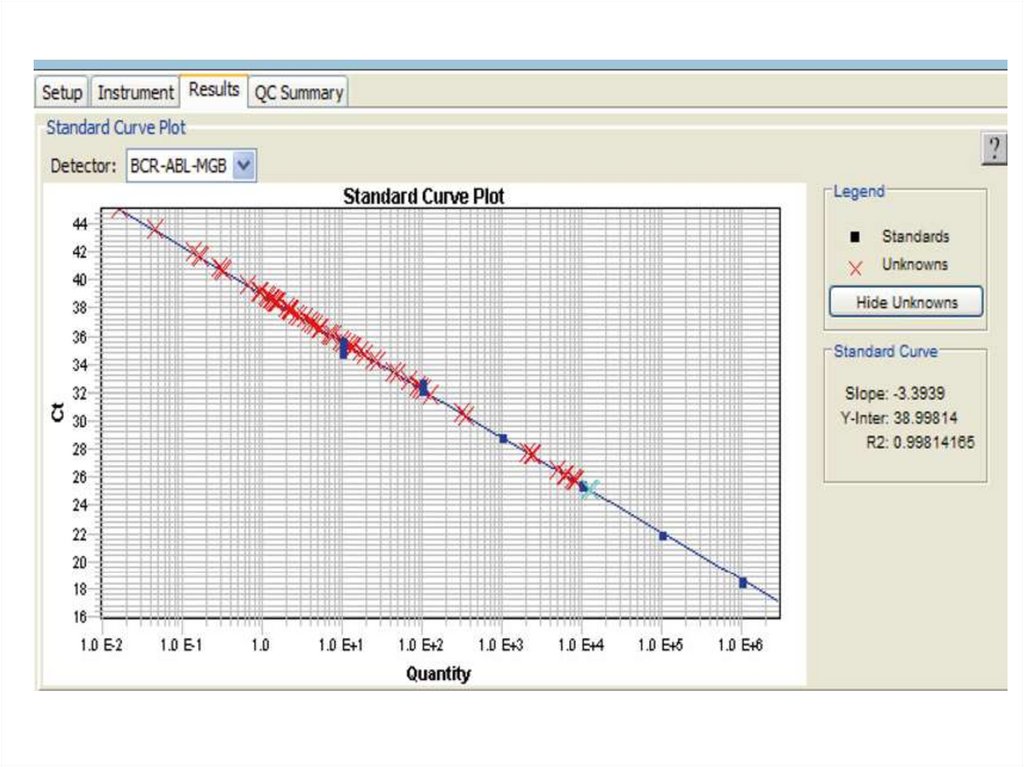
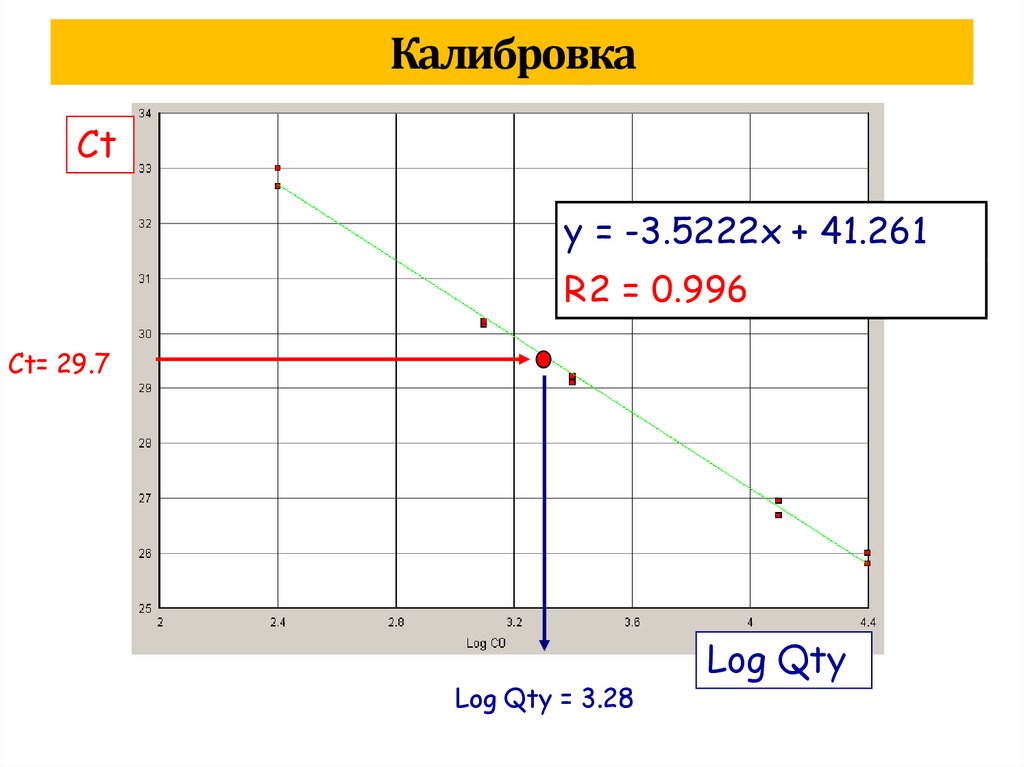



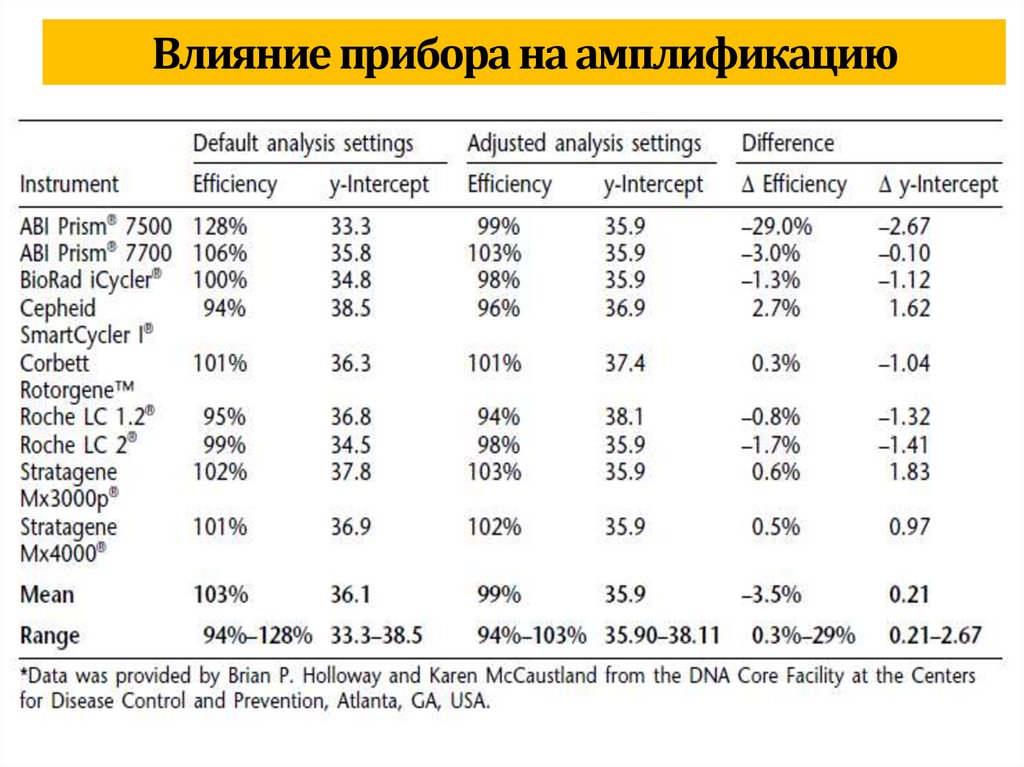

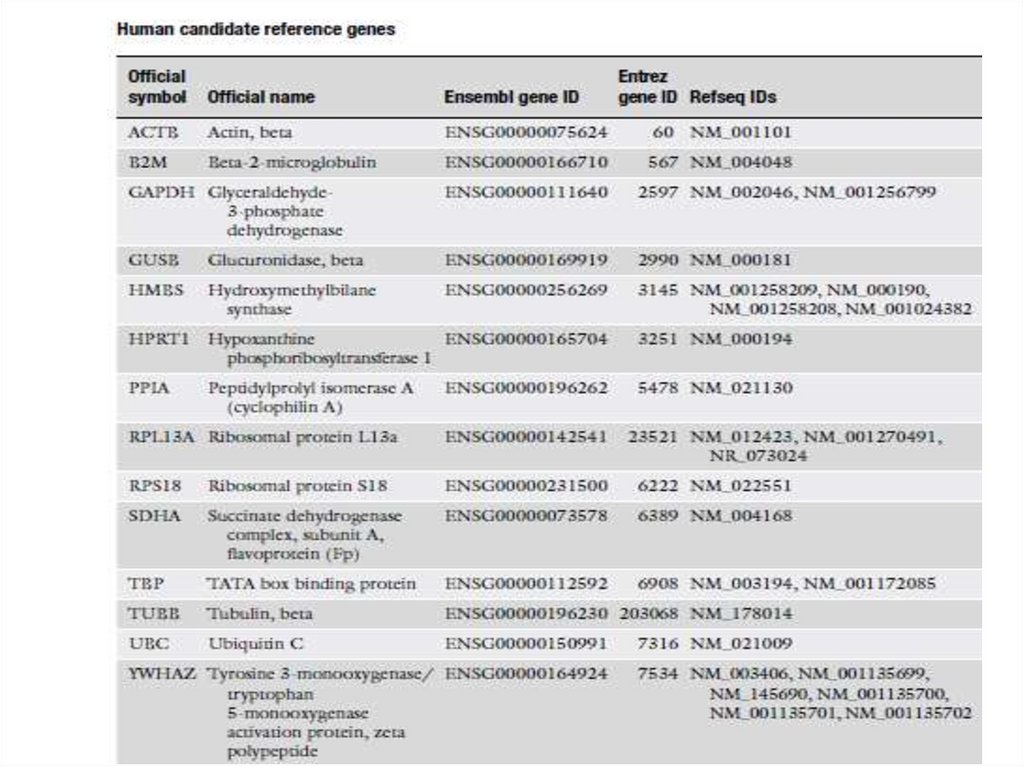





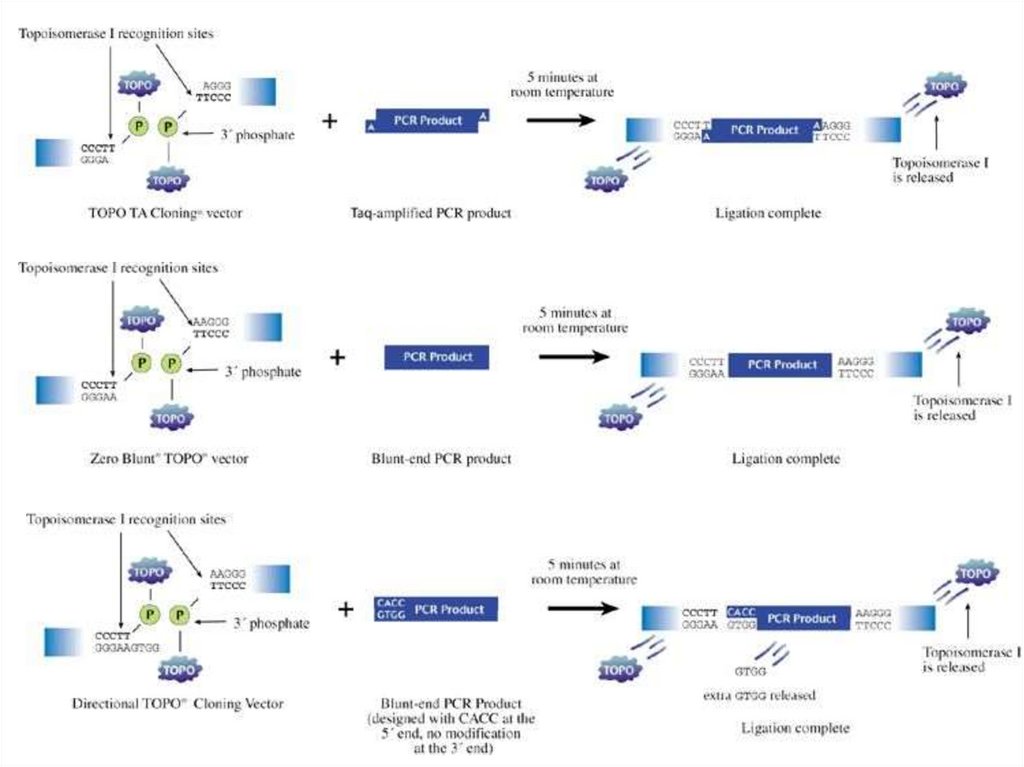
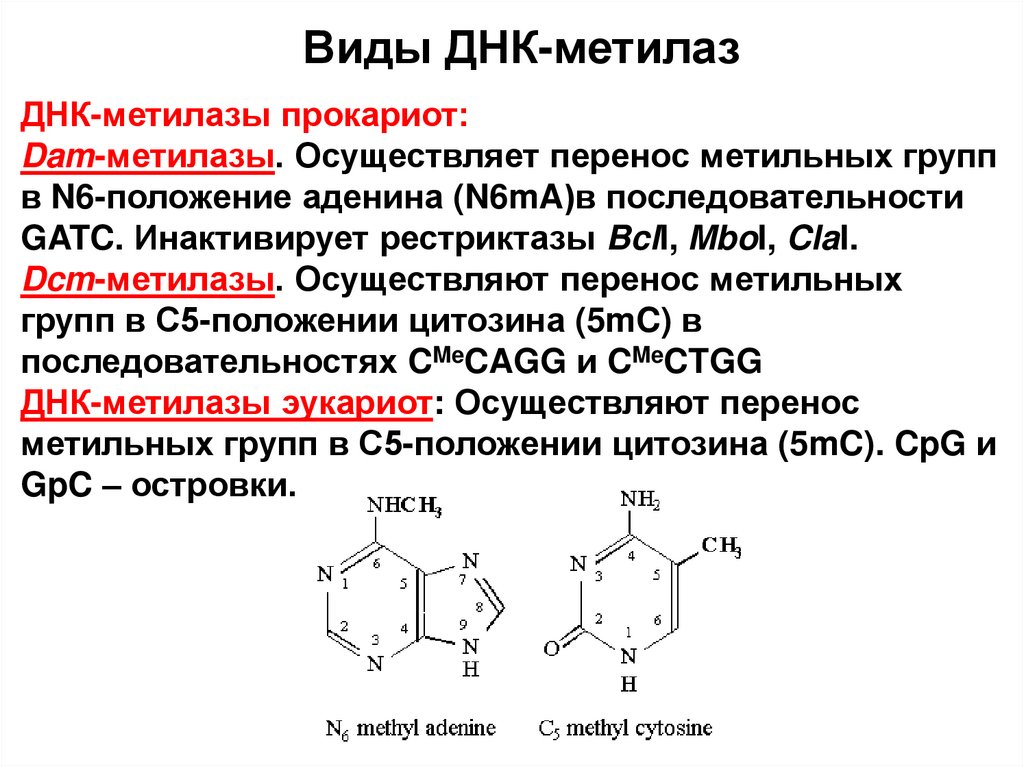

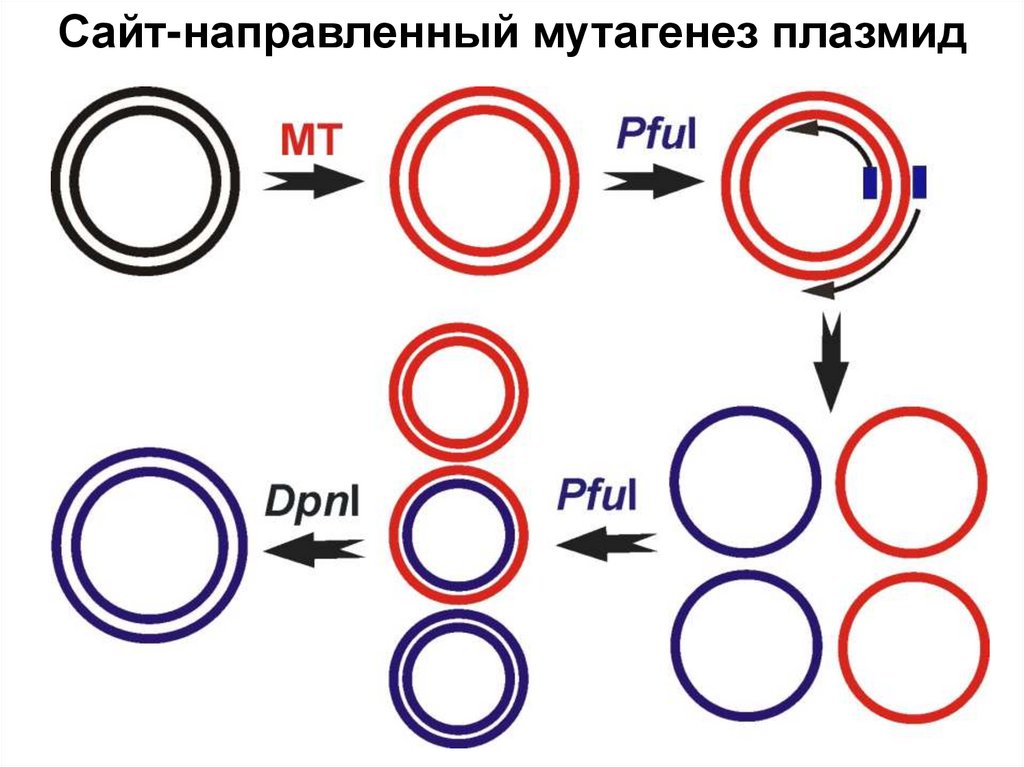










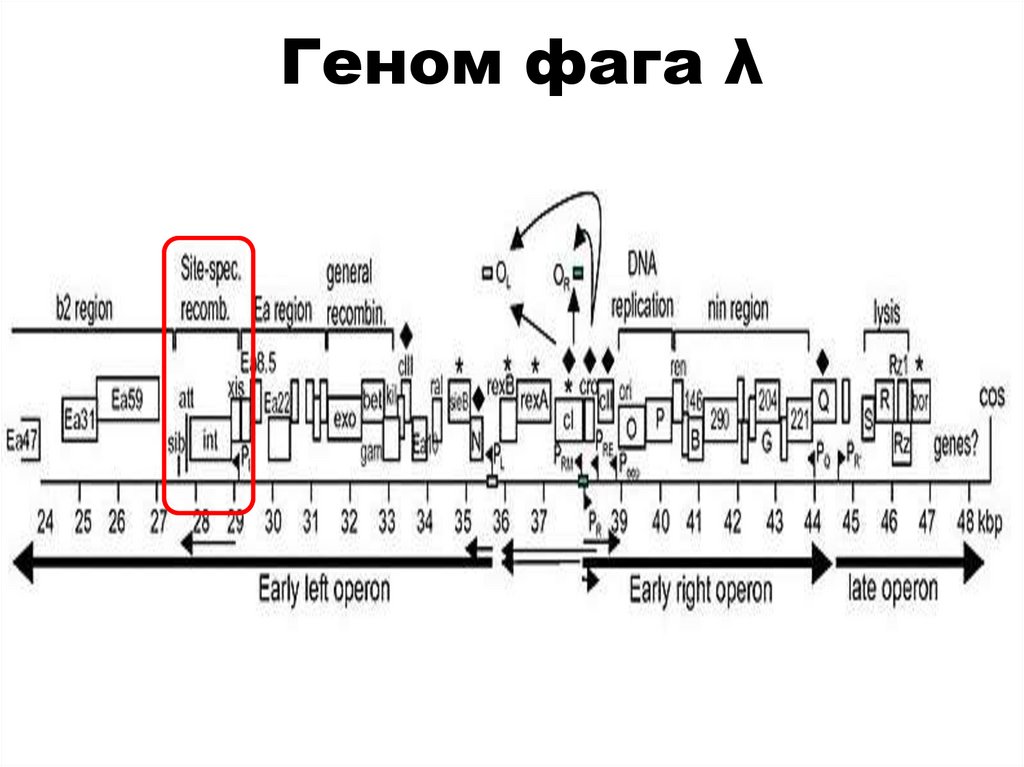
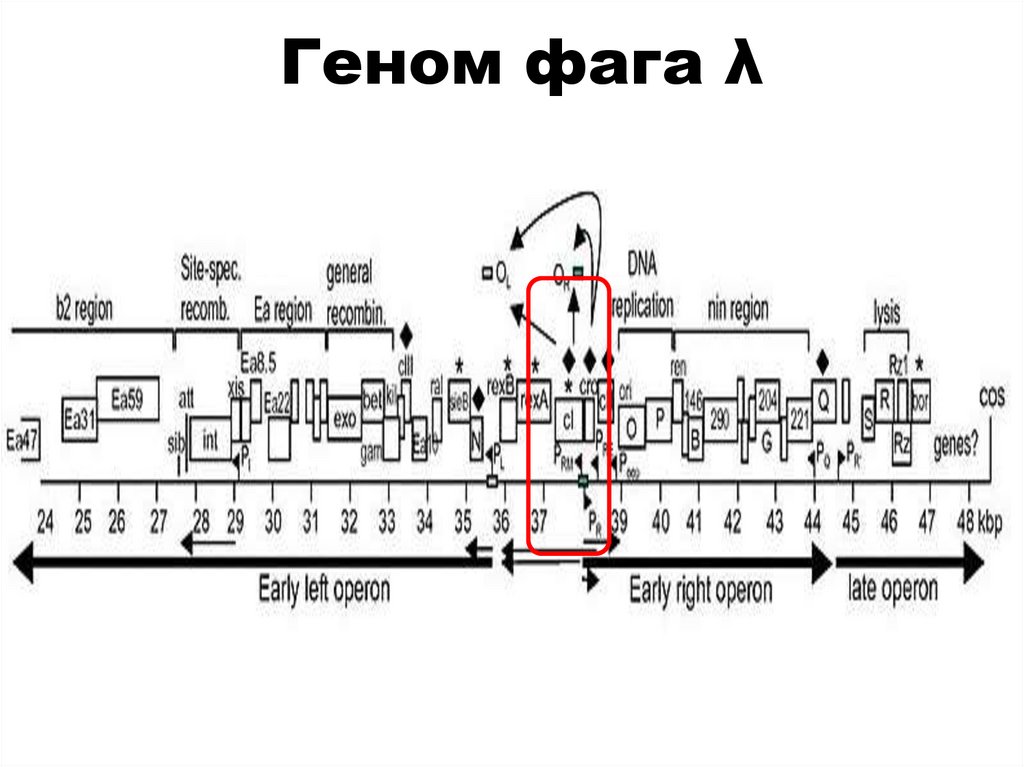
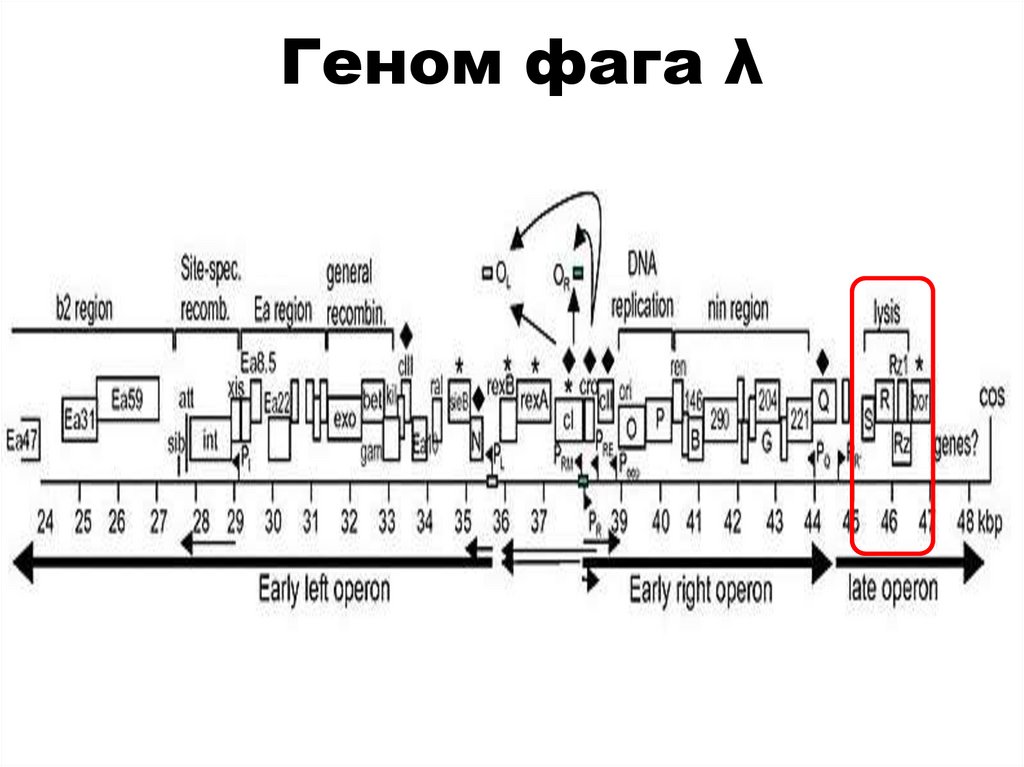
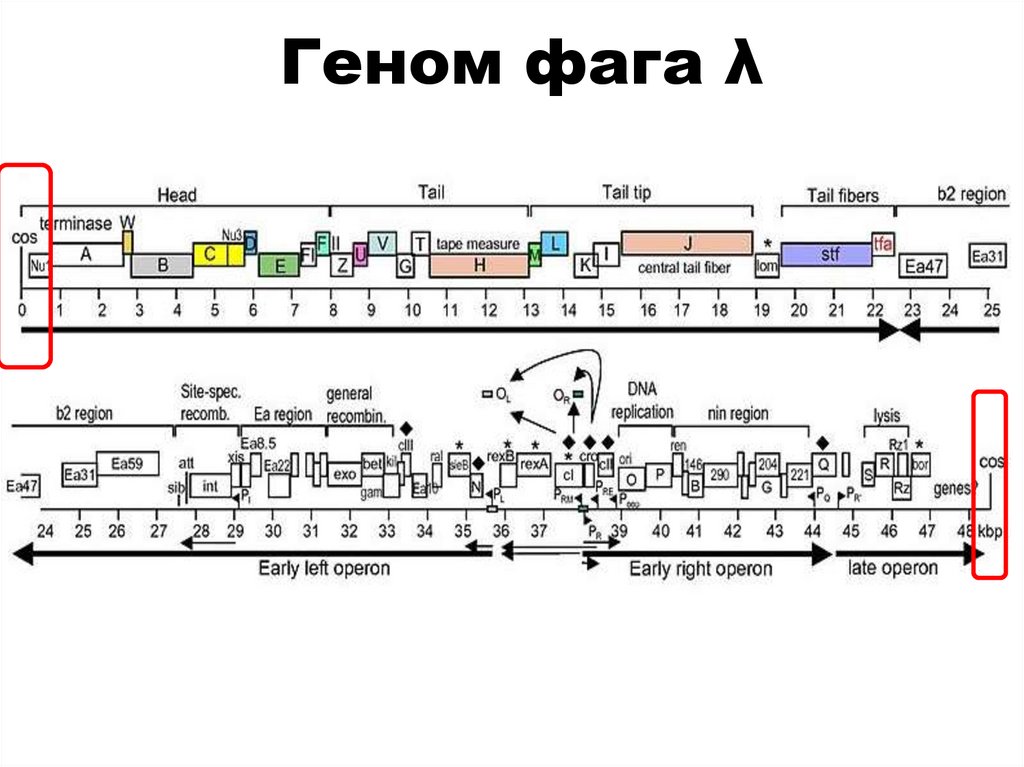












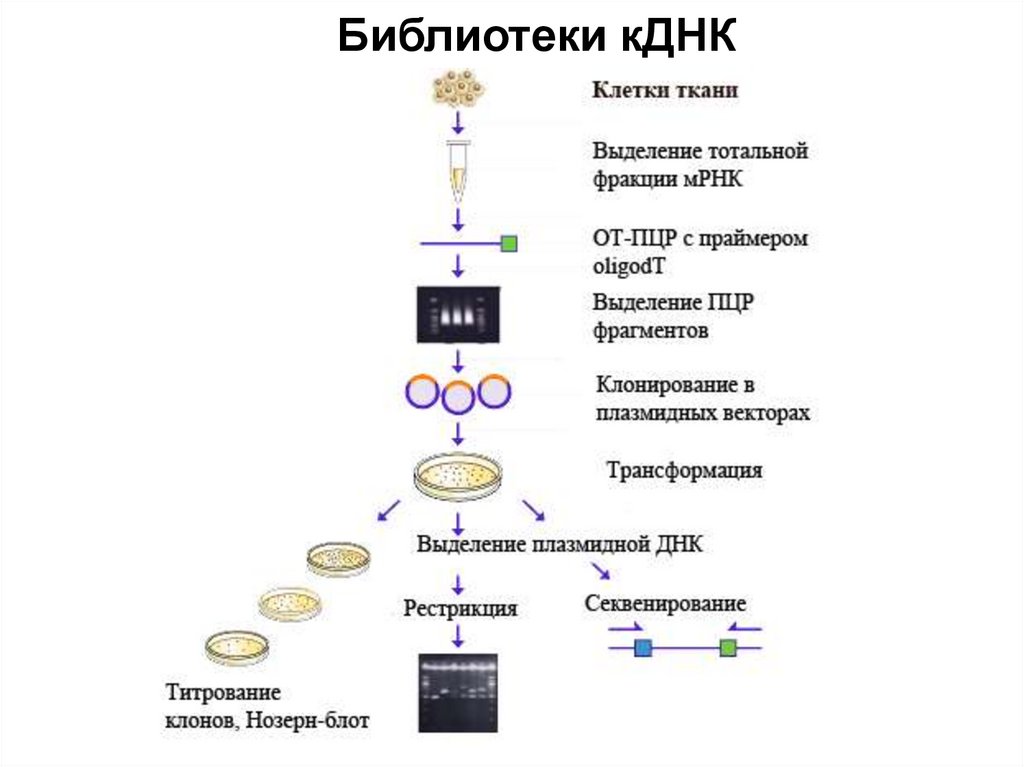
 biology
biology








