Similar presentations:
Современные подходы к молекулярной диагностике туляремии
1.
«САРАТОВСКИЙ НАЦИОНАЛЬНЫЙ ИССЛЕДОВАТЕЛЬСКИЙГОСУДАРСТВЕННЫЙ УНИВЕРСИТЕТ
ИМЕНИ Н.Г. ЧЕРНЫШЕВСКОГО»
ФКУН Российский противочумный институт “Микроб”
Роспотребнадзора
Современные подходы к молекулярной
диагностике туляремии
МАГИСТЕРСКАЯ РАБОТА
Студентки 3 курса 331 группы
Направления подготовки магистратуры 06.04.01 Биология
Биологического факультета
Чекмаревой Светланы Сергеевной
Научный руководитель:
к.б.н., доцент Е.В. Глинская
Научный консультант:
к.б.н., зав. отделом микробиологии
ФКУН Российский противочумный
институт “Микроб” Роспотребнадзора
Н.А. Осина
Саратов 2023
2.
АктуальностьТуляремия
–
зоонозная
природно-очаговая
инфекционная
болезнь,
характеризующаяся лихорадкой, интоксикацией и клиническими проявлениями в
зависимости от механизма заражения (поражение кожи, глаз, слизистой
ротоглотки,
легких,
кишечника,
лимфатических
узлов).
Возбудитель туляремии относится к микроорганизмам II группы патогенности.
Важное значение имеет своевременное
выявление патогена в биологическом материале и объектах
окружающей среды.
2
3.
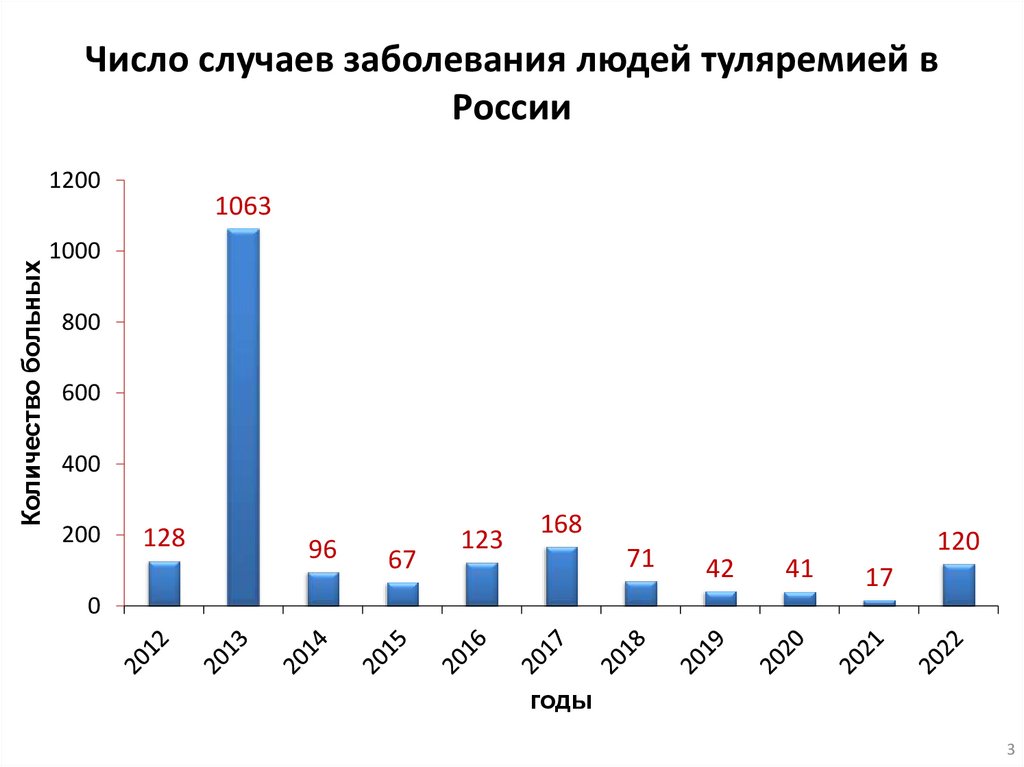
Число случаев заболевания людей туляремией вРоссии
Количество больных
1200
1063
1000
800
600
400
200
128
96
67
123
168
71
42
41
120
17
0
годы
3
4.
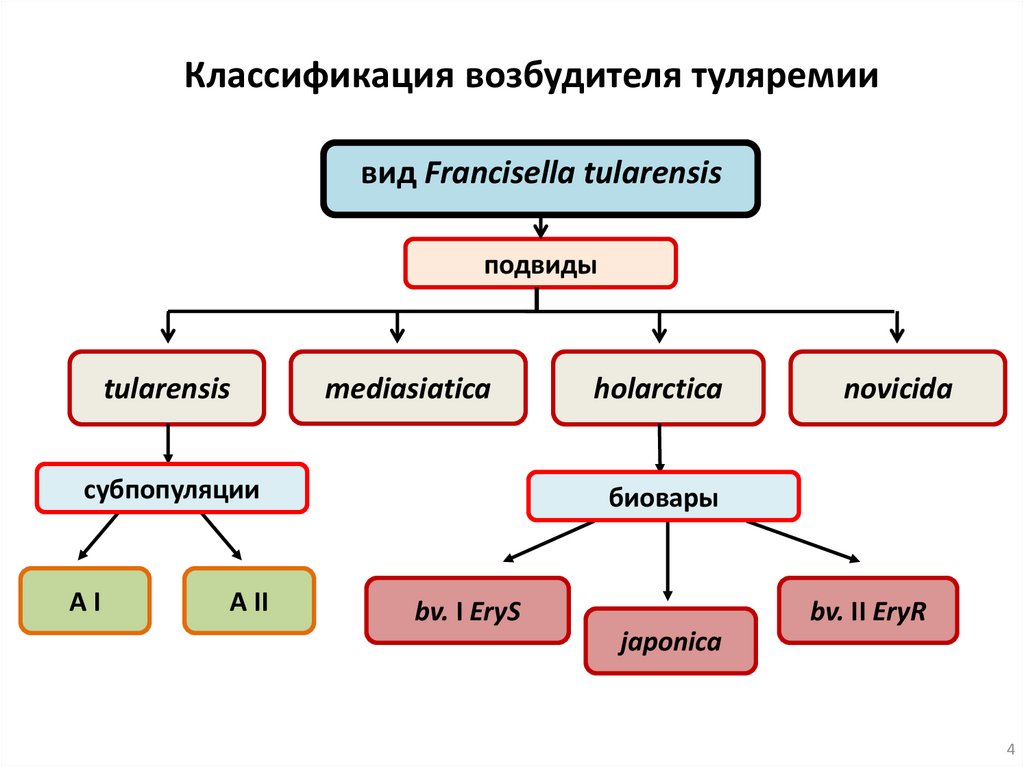
Классификация возбудителя туляремиивид Francisella tularensis
подвиды
tularensis
mediasiatica
субпопуляции
АI
A II
holarctica
novicida
биовары
bv. I EryS
bv. II EryR
japonica
4
5.
На территории Российской Федерациираспространен голарктический подвид
• резервуар – грызуны и зайцеобразные;
• переносчики – иксодовые клещи.
Заражение:
Рисунок 1 - Лесная мышь
(https://dezbox.ru/sredstva/
zookumarin-sredstvo-otgryzunov/)
• алиментарное – инфицированные
грызунами продукты питания и воду;
• контактное – при разделке зайцев,
пойманных охотниками в энзоотичных
территориях).
Рисунок 2 - Пастбищный
клещ
(https://mavink.com/explore/
Dermacentor-Reticulatus)
5
6.
Лабораторная диагностика туляремии• Бактериологические и биологические методы – для выделения культуры
возбудителя F. tularensis – предварительное накопление возбудителя в
органах
лабораторных
животных
увеличивает
продолжительность
исследования + загрязнение посевов посторонней микрофлорой.
• Иммунологические методы: метод флуоресцирующих антител (МФА),
иммуноферментный анализ (ИФА), реакция нейтрализации антител (РНАт)
– для обнаружения антигена и антител. Чувствительность данных методов
составляет 1×104 - 1×106 м.к./мл, продолжительность исследования – 2-5
часов.
• Молекулярная диагностика: полимеразная цепная реакция (ПЦР) –
перспективный подход быстрый (в течение 2-3 ч) выявления патогена в
пробах биологического материала и объектах окружающей среды с
высокой чувствительностью и специфичностью.
6
7.
Молекулярная диагностика туляремииЗарегистрированные генодиагностические препараты
для выявления ДНК F. tularensis методом ПЦР
“ Ген Francisella tularensis – РЭФ”
“ Ген Francisella tularensis – РГФ ”
“ Ген Francisella tularensis подвид – РЭФ”
“ Ген Francisella tularensis подвид – РГФ”
Производитель ФКУН Российский противочумный
институт “ Микроб ” Роспотребнадзора.
Наборы реагентов “ОМ-Скрин-Туляремия-РВ” производства ФБУН
ЦНИИ эпидемиологии, г. Москва.
• Не позволяют определять субпопуляции F. tularensis tularensis.
7
8.
Таблица 1 - Определение подвидов туляремийного микроба на основанииопределения вариабельности RD1 области, FT-M19, ISFtu2 по ВОЗ
Подвид
Размер ампликонов,
образуемых в ПЦР с
праймерами на основе
RD1-области
FT-М19
ISFtu2
1522
250
390
1453
250
390
1136
220
1248
holarctica bv.
EryS/R
924
220
1248
novicida
-
tularensis
mediasiatica
Рисунок 3 - RD-1
область
(фото Чекмаревой С.С.)
Трудоемко,
продолжительно
по времени 7-8 ч.
holarctica bv.
japonica
250
390
FT-M19
ISFtu2
Рисунок 4 - Области
FT-M19 и ISFtu2
(фото Чекмаревой С.С.)
Не идентифицирует
все подвиды –
только holarctica.
8
9.
Цель исследованияРазработка способов внутривидовой дифференциации
Francisella tularensis с помощью амплификационных и
секвенационных технологий.
9
10.
Задачи исследования1 Разработать мультилокусную систему для одновременного определения
подвидов, биоваров и субпопуляций F. tularensis методом ПЦР с учетом
результатов в режиме реального времени: выбор ДНК-мишеней, подбор
праймеров и зондов, оптимизация условий амплификации.
2 Определить внутривидовую принадлежность штаммов из Государственной
коллекции патогенных бактерий ФКУН Российский противочумный институт
«Микроб» с помощью предложенного подхода.
3 Оценить эффективность дифференциации эритромицинчувствительного и
эритромицинрезистентного биоваров F. tularensis holarctica на основании
выявления точечных мутаций (SNP) методом фрагментного секвенирования по
Сэнгеру.
4 Разработать алгоритм расширенной идентификации штаммов F. tularensis с
помощью молекулярно-генетических методов.
10
11.
Материалы исследованияГруппы штаммов из Государственной коллекции патогенных бактерий
ФКУН Российского противочумного института “Микроб“
1. F. tularensis subsp. tularensis AI.
2. F. tularensis subsp. tularensis AII.
3. F. tularensis subsp. mediasiatica.
4. F. tularensis subsp. holarctica, bv. EryS/R.
5. F. tularensis subsp. holarctica bv. japonica.
6. F. tularensis subsp. novicida.
11
12.
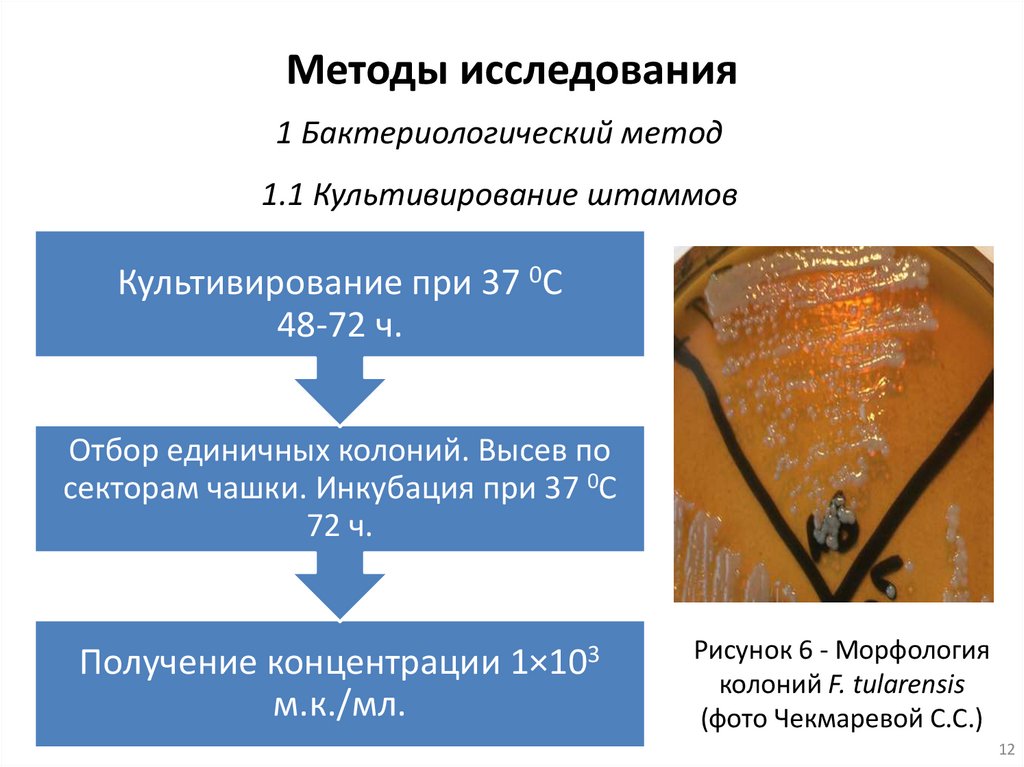
Методы исследования1 Бактериологический метод
1.1 Культивирование штаммов
Культивирование при 37 0С
48-72 ч.
Отбор единичных колоний. Высев по
секторам чашки. Инкубация при 37 0С
72 ч.
Получение концентрации 1×103
м.к./мл.
Рисунок 6 - Морфология
колоний F. tularensis
(фото Чекмаревой С.С.)
12
13.
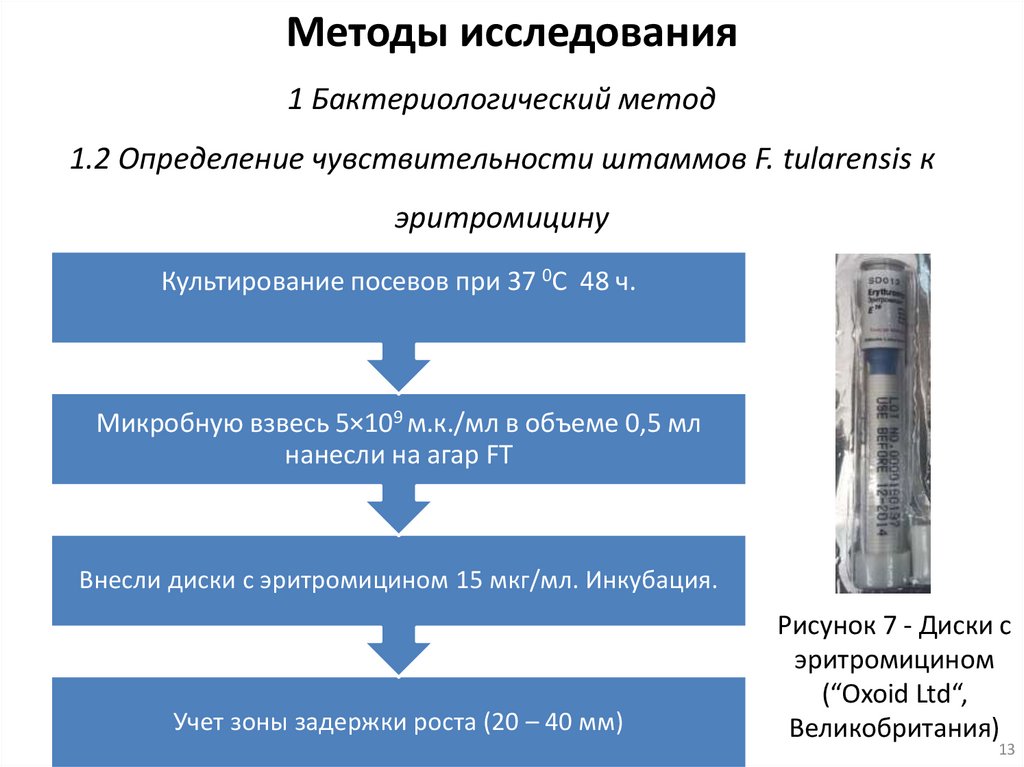
Методы исследования1 Бактериологический метод
1.2 Определение чувствительности штаммов F. tularensis к
эритромицину
Культирование посевов при 37 0С 48 ч.
Микробную взвесь 5×109 м.к./мл в объеме 0,5 мл
нанесли на агар FТ
Внесли диски с эритромицином 15 мкг/мл. Инкубация.
Учет зоны задержки роста (20 – 40 мм)
Рисунок 7 - Диски с
эритромицином
(“Oxoid Ltd“,
Великобритания)
13
14.
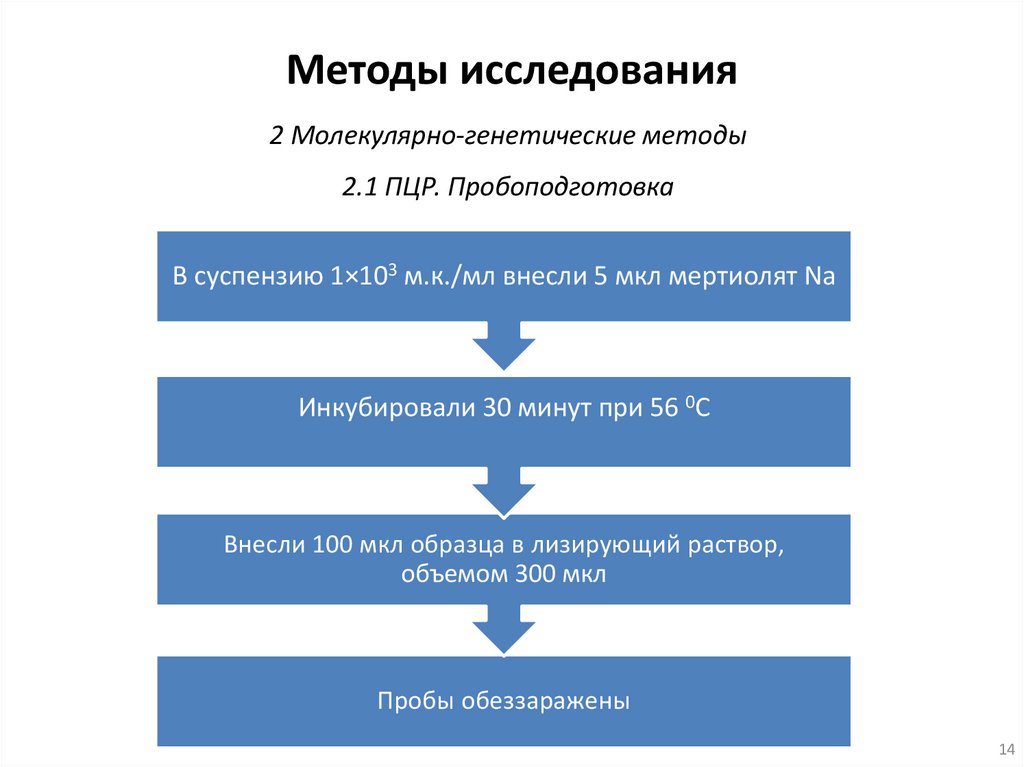
Методы исследования2 Молекулярно-генетические методы
2.1 ПЦР. Пробоподготовка
В суспензию 1×103 м.к./мл внесли 5 мкл мертиолят Na
Инкубировали 30 минут при 56 0С
Внесли 100 мкл образца в лизирующий раствор,
объемом 300 мкл
Пробы обеззаражены
14
15.
Методы исследования2 Молекулярно-генетические методы
2.2 ПЦР. Выделение ДНК
Выделение ДНК проводили с
помощью набора “ДНК-сорб В”
(“АмплиСенс”,
Москва)
в
соответствии с инструкцией к
препарату
Рисунок 7 - Компоненты набора “ДНКсорб В” (“АмплиСенс”, Москва)
(http://www.vetlabs.ru/katalog/naborreagentov/19569/)
15
16.
Методы исследования2 Молекулярно-генетические методы
2.3 Постановка ПЦР
Индикация F. tularensis – с использованием набора реагентов
“Ген F. tularensis – РГФ” с гибридизационно-флуоресцентной
детекцией.
Подвидовую принадлежность определяли на основании
анализа вариабельности области дифференциации RD1
методом ПЦР с электрофоретической детекцией.
16
17.
Методы исследования2 Молекулярно-генетические методы
2.4 Фрагментное секвенирование
Выявление полиморфных нуклеотидов в гене sdhA методом
фрагментного секвенирования по Сэнгеру – относительно гена
sdhA штамма F. tularensis Schu S4 в программе “MEGA X64” ,
выравнивание в “NCBI GenBank”, алгоритм “BLAST”.
Таблица 2 - Анализ нуклеотидных последовательностей
Подвид
Нуклеотиды
F. tularensis tularensis AI
441G, 465A, 480C, 750T, 795 C
F. tularensis mediasiatica
441G, 465G, 480T, 750T, 795C
F. tularensis tularensis AII;
441G, 465G, 480C, 750T, 795С
F. tularensis holarctica bv. Ery R/S
F. tularensis holarctica bv. japonica
441G, 465G, 480C, 750C, 795C
F. tularensis novicida
441A, 465G, 480T, 750T, 795T
17
18.
Результаты исследования1 Разработка мультилокусной ПЦР для внутривидовой
дифференциациии F. tularensis
18
19.
1 Анализ генетической вариабельности штаммов F. tularensis по даннымпроведенного анализа
RD-8 обнаружена:
F. tularensis holarctica japonica,
F. tularensis novicida, (делеция 19 п.н.).
F. tularensis tularensis (ранее не отмечено)
Ген FTW2084 содержит RD-12 + VNTR15;
Присутствует у штаммов патогена всех
подвидов и субпопуляций
Выявлена новая RDhm 346 п.н..:
F. tularensis holarctica и mediasiatica;
Делетирована у F. tularensis
tularensis
20.
2 Разработка мультилокусной ПЦР для внутривидовойдифференциации F. tularensis
Таблица 3 - Подбор ДНК-мишеней и праймеров
Наименование
праймеров/зондов
FTT1122
FTT1670c
FTT1067c
FTW2084
Нуклеотидная последовательность, 5' – 3'
ATGCTGCTAAAGCACATGGA
GCGCAGAGTCTCTACCTGAA
Зонд FAM-ACCAAAGTGGTGATAATTCTTC-RTQ1
ATCCAAGCAATACCTTTAGT
TCACTAATGCTTGTGGTACT
Зонд R6G-TCCAGCGTATCATATGCTAA- BHQ1
AGCAAATTCTAATTCTCTATCTAA
GGCTATATAATTGATGATGTCT
Зонд ROX-TCATCACCCAATCTAATGAACTTACCBHQ2
CAATGACCAAGGTAATAATG
ATACAGTTTGAGGATTCCAT
Зонд Cy5-ACCAAAGTGGTGATAATTCTTC-BHQ2
20
21.
2 Разработка мультилокусной ПЦР для внутривидовойдифференциациии F. tularensis (продолжение)
2.1 Оптимизация состава реакционной смеси
Таблица 4 - Сборка реакционной смеси
Реагент
На одну
реакцию
5 мкл
Смесь Т (праймеры (8
Пмоль) – 8 пМоль, зонды –
4 пМоль, дНТФ)
Смесь РС (2,5хБ, 6,25
10 мкл
мМоль MgCl2)
Taq-полимераза, 1 мкл/5 ед 1 мкл
ДНК
10 мкл
ВСЕГО
25 мкл
Рисунок 9 - Оборудование в
ламинарном боксе
(фото Чекмаревой С.С.)
21
22.
2 Разработка мультилокусной ПЦР для внутривидовойдифференциациии F. tularensis (продолжение)
2.2 Оптимизация программы амплификации
Таблица 5 - Программа амплификации
Этап
Температура
Время
Денатурация
95 ⁰С
95 ⁰С
59 ⁰С
72 ⁰С
95 ⁰С
56 ⁰С
(детекция)
72 ⁰С
5 мин
10 с
30 с
20 с
10 с
30 с
Первый
цикл
Второй цикл
Количество
повторов
1
10
35
20 с
Рисунок 10 - Амплификатор Оптимизировали: количество циклов – 10 циклов без
“Rotor Gene Q ”
учета флуоресценции, 35 циклов – с учетом, температура
(“QIAGEN ”, Германия)
отжига праймеров и зондов в первые 10 циклов – 59 ⁰С,
в последующие 35 – 56 ⁰С, время каждого шага
22
23.
ШтаммКанал Green (FTT1122)
Канал Yellow (FTT1670c)
Канал Orange (FTT1067c)
Канал Red (FTW2084)
F.
tularensis
tularensis
AI
F.
tularensis
tularensis
AII
F.
tularensis
mediasiatic
a
F.
tularensis
holarctica
Ery
F.
tularensis
holarctica
japonica
F.
tularensis
novicida
Рисунок 11 - Графики со специфичным свечением по соответствующим каналам
для каждого подвида, субпопуляции и биовара штаммов F. tularensis
(фото Чекмаревой С.С.)
23
24.
Таблица 6 - Таблица интерпретации полученных результатовFAM
HEX
ROX
Cy5
(Green)
FTT1122
(Yellow)
FTT1670c
(Orange)
FTT1067c
(Red)
FTW2084
F. tularensis tularensis AI
+
+
-
-
F. tularensis tularensis AII
+
+
-
+
F. tularensis mediasiatica
+
+
+
-
F. tularensis holarctica bv
Ery S/R
+
-
-
-
F. tularensis holarctica bv
japonica
+
-
+
-
F. tularensis novicida
-
+
-
+
Проба
24
25.
Таблица 7 - Чувствительность и специфичность мультилокуснойтест-системы ПЦР «F. tularensis 4c»
Наименование
Определение подвидовой принадлежности с
штаммов
использованием подходов
Анализ RD1 Анализ sdhA гена Мультилоку-сная
(ВОЗ)
ПЦР
“F. tularensis-4c”
КМ-7 (O-328);
Результаты
идентификации
tularensis
tularensis AI
tularensis AI
tularensis AI
tularensis
holarctica EryS/R
tularensis AII
tularensis AII
mediasiatica
mediasiatica
mediasiatica
mediasiatica
КМ-8 (М-498); 9; 84; 7;
holarctica
holarctica EryS/R
holarctica EryS/R
holarctica EryS/R
359…
EryS/R
Utah112
Не идент.
novicida
novicida
novicida
Невада № 14; О-402
КМ-6 (E-261); О/284 ;
В-399; A.Coli; О-419
КМ-4 (А-61(117));
А-142(112); А-148(120)
25
26.
3 Разработка способа дифференциации эритромицинчувствительного иэритромицинрезистентного биоваров F. tularensis методом фрагментного
секвенирования
• ДНК-мишени – два участка гена 23S рДНК (единичные полиморфные
нуклеотиды A449G и A2052C, T2123C, С588T).
• Подбор специфичных праймеров + оптимизация условий амплификации.
• Во всех случаях подтверждена специфичность выбранных мутаций.
Таблица 8 - Выявление мутаций для определения биоваров
F. tularensis sp. holarctica
Подвид
Мутации в гене 23S рДНК
F. tularensis holarctica bv. japonica
T2123C
F. tularensis holarctica bv. EryR
С588T; A449G; A2052C; T2123C
F. tularensis holarctica bv. Ery S
С588T; T2123С
26
27.
3 Разработка способа дифференциации эритромицинчувствительного иэритромицинрезистентного биоваров F. tularensis методом фрагментного
секвенирования
A449G
A2052C
T2123C
С588T
Рисунок 12 - Выявленные мутации в гене 23S рДНК подвидов F. tularensis
(фото Чекмаревой С.С.)
27
28.
4 Применение комплексного подхода для дифференциацииколлекционных штаммов F. tularensis с помощью молекулярногенетических методов
ПЦР-РВ
“F. tularensis 4с”
дифференциация всех
внутривидовых групп
(локусы FTT122, FTT1670c,
FTT1067c, FTW2084)
+
Фрагментное
секвенирование
23 рДНК
определение биоварной
принадлежности
F. tularensis sp. holarctica
(SNP A449G, С588T,
A2052C, T2123C)
28
29.
Таблица 9 - Результаты идентификации штаммов F. tularensis из фонда ГКПБ«Микроб» с использованием различных подходов
Наименование
штамма
Источник /
место
выделения
О-328
Клещи
/Канада
74
503/850
Miura
Utah112
Внутривидовая принадлежность
Определени
По
Система
Секвенирование
е
анализу “F. tularensis23S
sdhA
чувствительн
RD1
4c”
рДНК
ости к
эритромици
ну
н/и
Результаты
идентификации
tularensis
tularensis AI
н/и
tularensis AI
tularensis AI
Полевка
Чувствител holarctica
обыкновенн
ен
EryS/R
ая/
Московская
обл
Клещи D. Резистенте holarctica
pictus /
н
EryS/R
Московская
обл.
Человек/ Чувствител holarctica
Япония
ен
japonica
holarctica
EryS/R
bv.
EryS
holarctica
EryS/R
holarctica EryS
holarctica
EryS/R
bv.
EryR
holarctica
EryS/R
holarctica EryR
holarctica
japonica
н/и
holarctica
japonica
holarctica japonica
Озеро/ США
novicida
н/и
novicida
novicida
н/и
Не идент.
29
30.
Выводы1. На основании проведенного анализа получены новые данные о
структуре и встречаемости областей дифференциации RD-8, RD-12,
RD-28, гена FTT1122с и гомологичных ему последовательностей у
штаммов F. tularensis различных подвидов, биоваров и
субпопуляций.
2. Выявлена новая RDhm область дифференциации размером 346 п.н.,
характерная для штаммов патогена subsp. mediasiatica, holarctica,
которая делетирована у subsp. tularensis и отсутствует у subsp.
novicida.
3. Подобраны локусы, перспективные для использования при
внутривидовой дифференциации возбудителя туляремии методом
ПЦР: FTT1670, FTT1122с, FTT1067, FTW2084.
4. Сконструирована мультилокусная ПЦР “F. tularensis 4c”
обеспечивает определение всех подвидов F. tularensis: tularensis,
mediasiatica, holarctica, novicida, отдельно биовара japonica
голарктического подвида и субпопуляции АI, AII подвида tularensis.
30
31.
Выводы5. Определены единичные полиморфные нуклеотиды, специфичные
для подвида holarctica и его биоваров методом фрагментного
секвенирования по Сэнгеру четырех мутаций в гене 23S рДНК
(A449G, С588T, A2052C, T2123C). Используя предложенный подход,
удалось определить биоварную принадлежность F. tularensis
holarctica.
6. Подтверждена высокая специфичность мультилокусной ПЦР
при исследовании штаммов F. tularensis различного происхождения
из фонда ГКПБ ФКУН Российский противочумный институт “Микроб”.
7. Разработанные современные подходы для определения подвидов,
биоваров и субпопуляций патогена позволяют с высокой
специфичностью проводить внутривидовую дифференциацию
F. tularensis .
31



















 medicine
medicine








