Similar presentations:
Секвенирование нового поколения (NGS)
1.
Секвенирование нового поколения (NGS)Васильев Геннадий Владимирович
Институт цитологии и генетики СО РАН
2.
Революция цены в геномных исследованиях454 GS20
1000 Геномов
SOLiD 3.0
Illumina GA2
3.
Второе поколение секвенаторов – NGS – создатели геномикиRoche FLX Titanium
SOLiD 5500
Illumina HiSeq
Ion Proton
4.
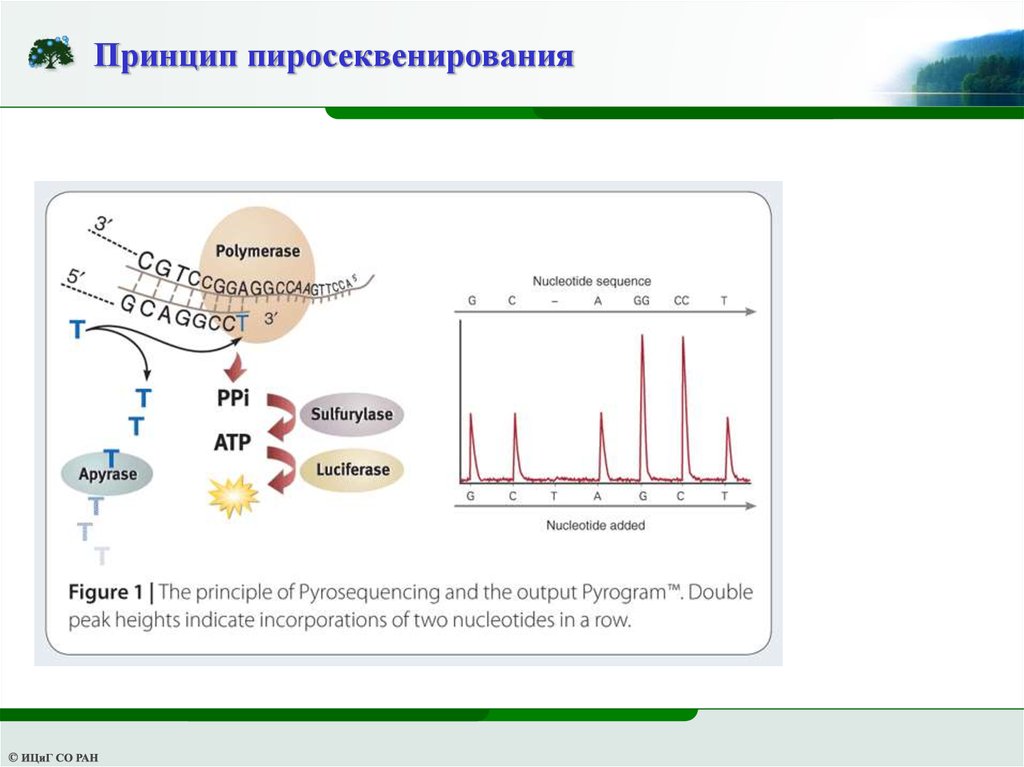
Принцип пиросеквенирования5.
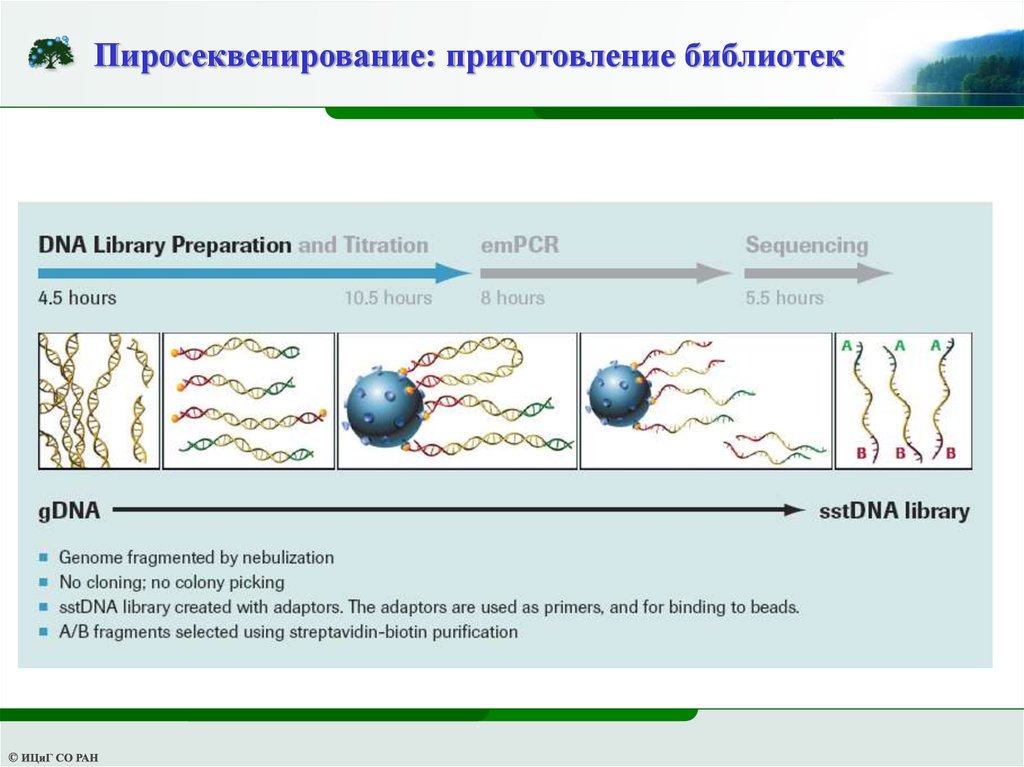
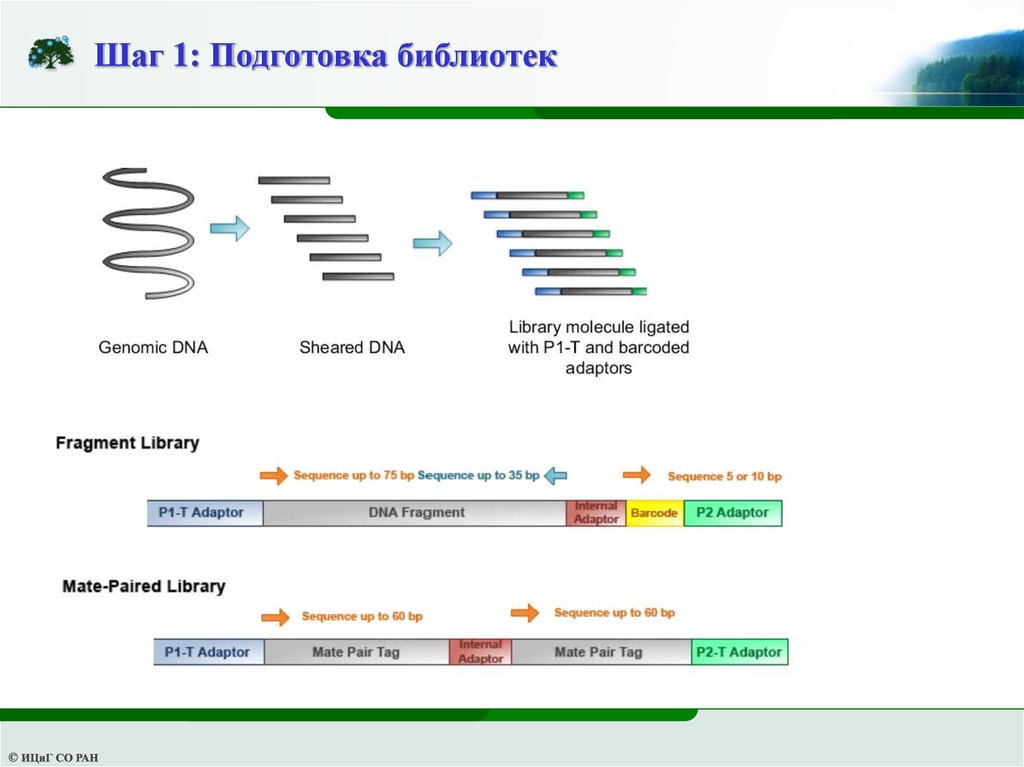
Пиросеквенирование: приготовление библиотек6.
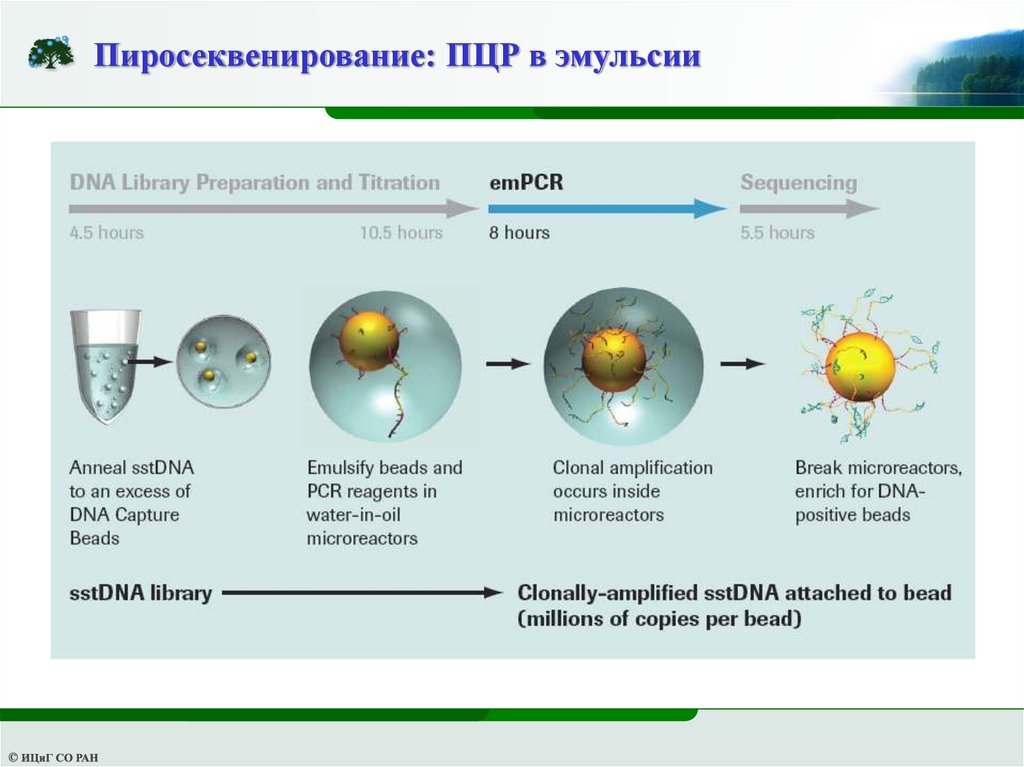
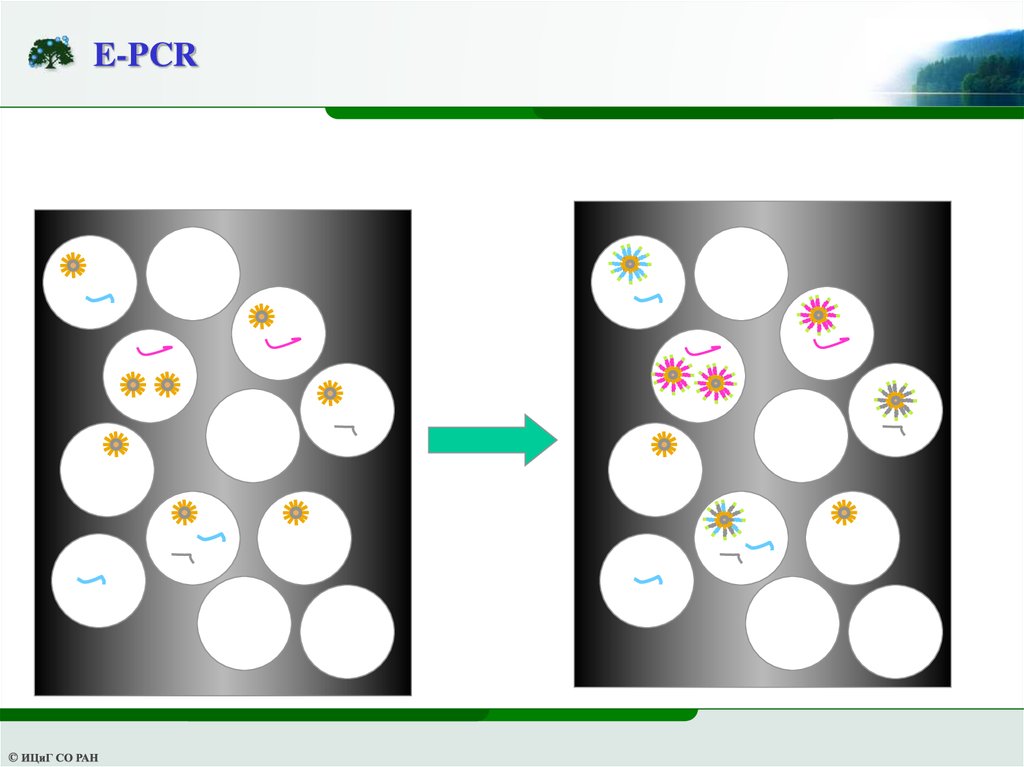
Пиросеквенирование: ПЦР в эмульсии7.
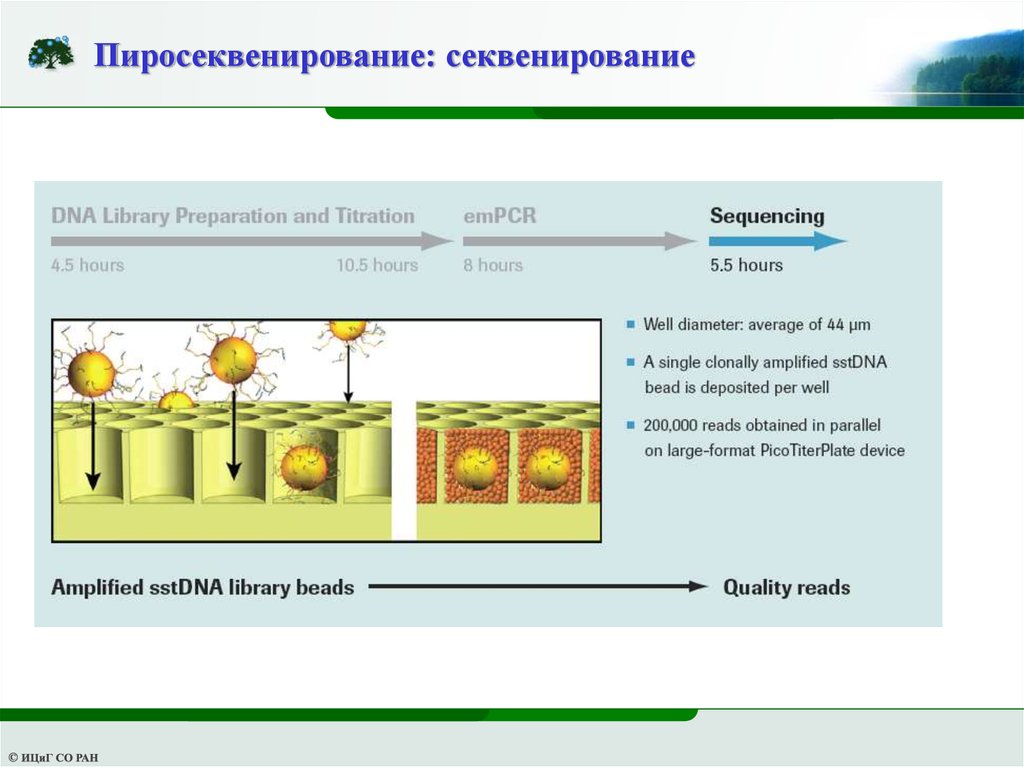
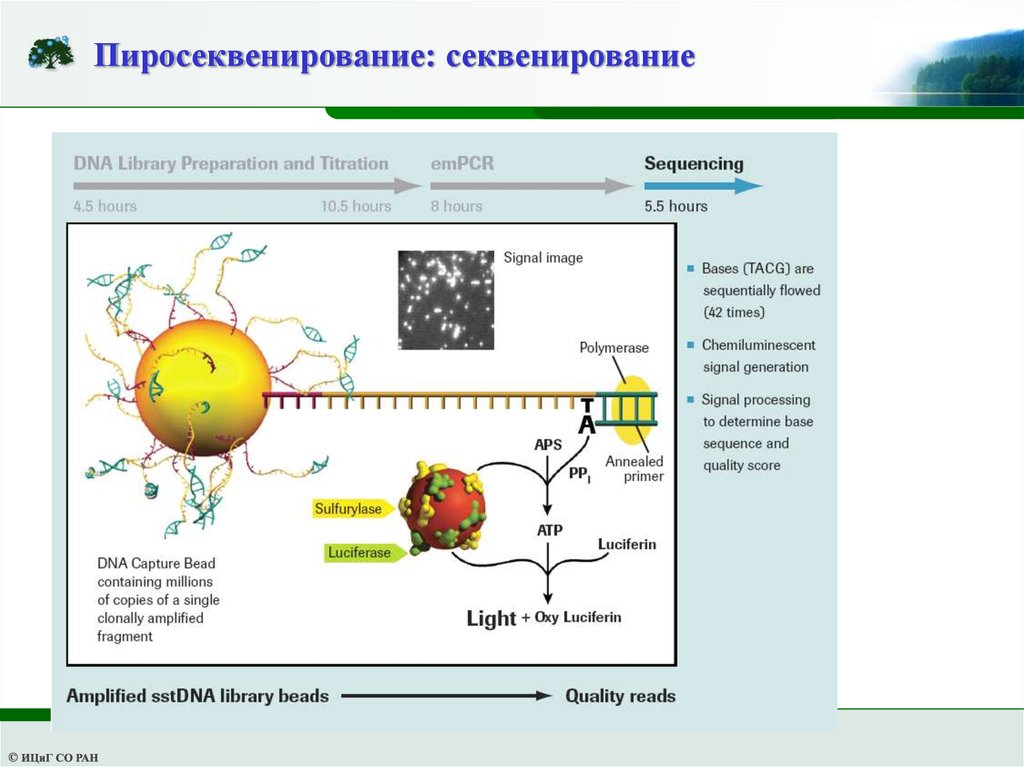
Пиросеквенирование: секвенирование8.
Пиросеквенирование: секвенирование9.
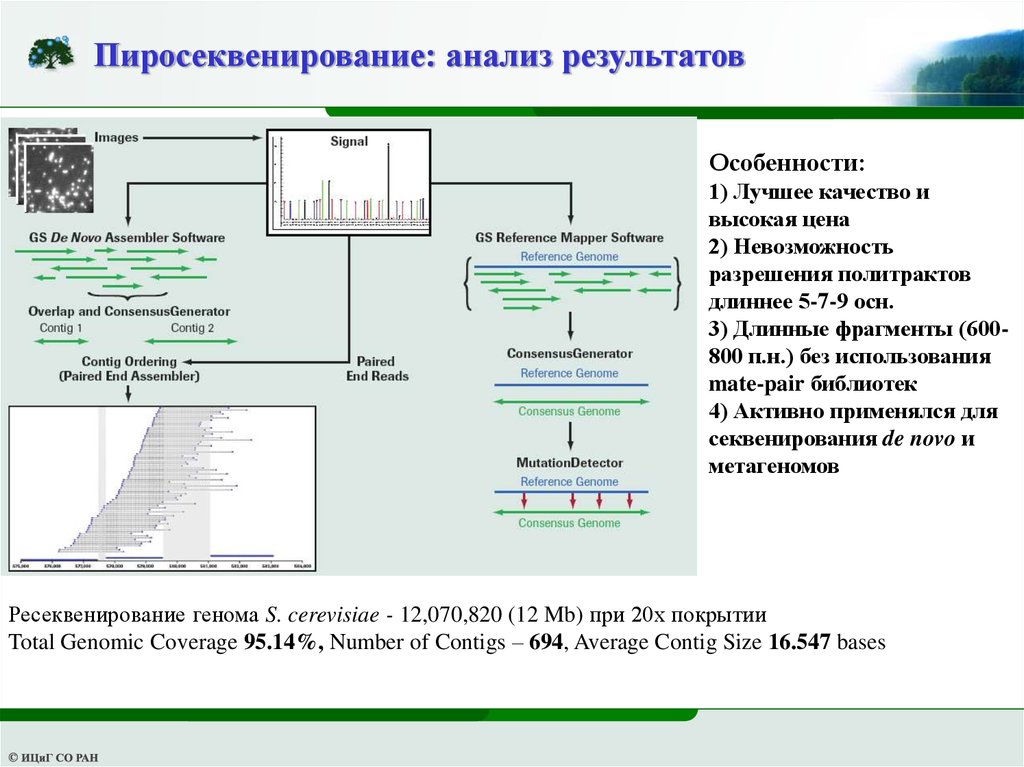
Пиросеквенирование: анализ результатовОсобенности:
1) Лучшее качество и
высокая цена
2) Невозможность
разрешения политрактов
длиннее 5-7-9 осн.
3) Длинные фрагменты (600800 п.н.) без использования
mate-pair библиотек
4) Активно применялся для
секвенирования de novo и
метагеномов
Ресеквенирование генома S. cerevisiae - 12,070,820 (12 Mb) при 20х покрытии
Total Genomic Coverage 95.14%, Number of Contigs – 694, Average Contig Size 16.547 bases
10.
SOLiDSOLiD 4
SOLiD 5500
Особенности:
1) Самые короткие чтения 50 или 75 п.н., используется при ресеквенировании и анализе
транскриптомов
2) Есть чтение mate-pair библиотек
3) Достаточно высокая точность при использовании специализированного ПО
11.
Шаг 1: Подготовка библиотек12.
SOLiD: результат ПЦР в эмульсии1.6 x 109
150 – 300 x 109
13.
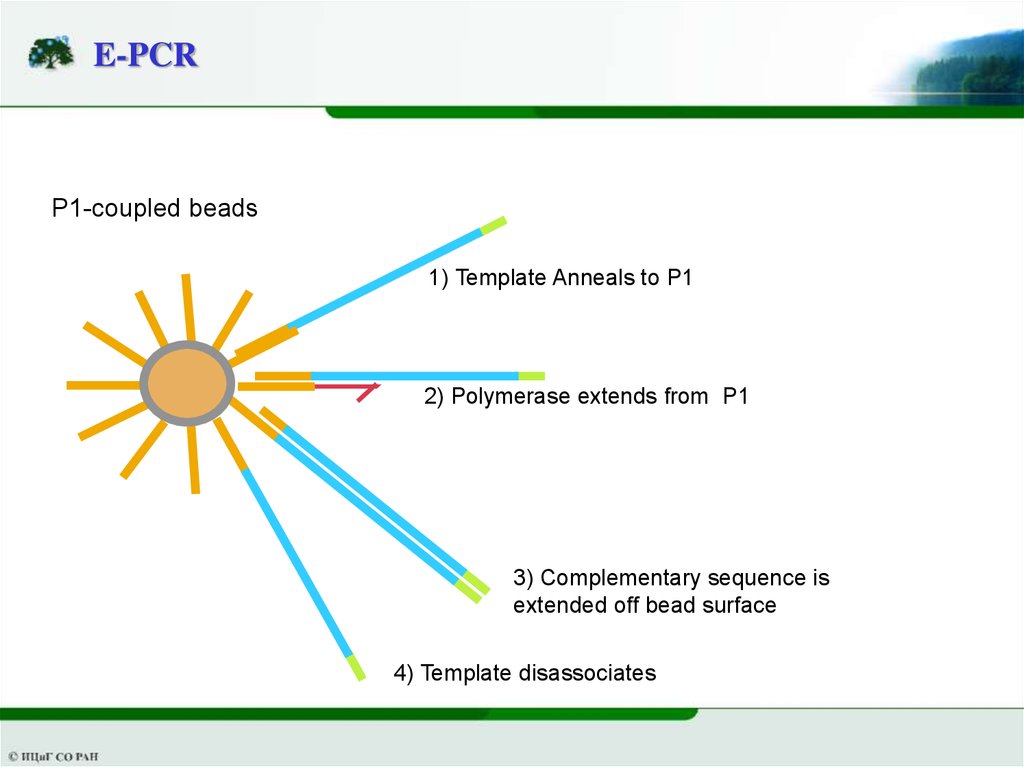
E-PCR14.
E-PCRP1-coupled beads
1) Template Anneals to P1
2) Polymerase extends from P1
3) Complementary sequence is
extended off bead surface
4) Template disassociates
15.
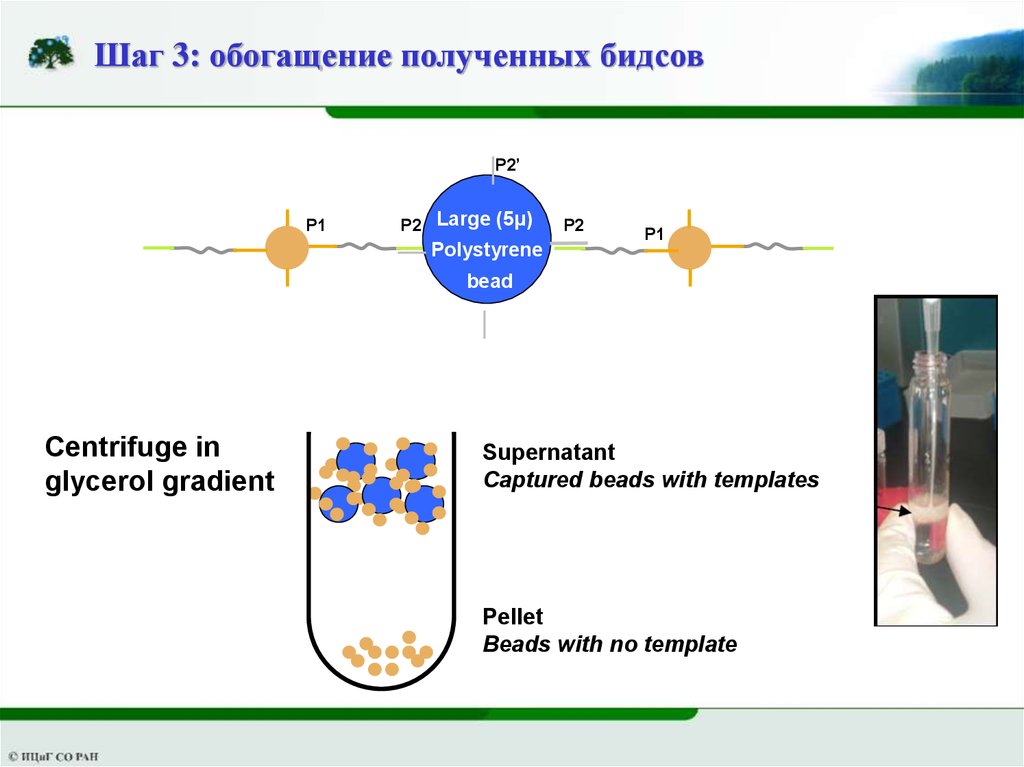
Шаг 3: обогащение полученных бидсовP2’
P1
P2 Large (5µ)
Polystyrene
P2
P1
bead
Centrifuge in
glycerol gradient
Supernatant
Captured beads with templates
Pellet
Beads with no template
16.
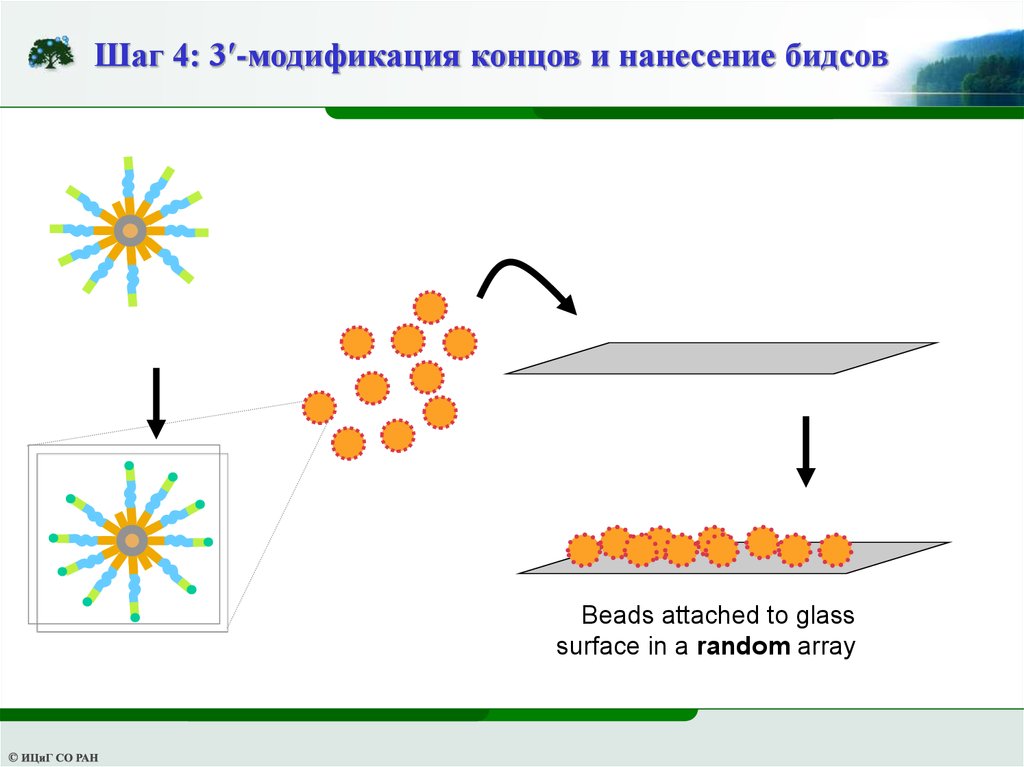
Шаг 4: 3′-модификация концов и нанесение бидсовBeads attached to glass
surface in a random array
17.
SOLiD: Пробы2 Base Pair Encoding
Using 4 Dyes
Red-probe
2nd Base
1st Base
A
C
G
5’
3’
n n n A T z z z
T
A
C
G
T
Blue-probe
3’
5’
n n n T T z z z
On our probes the 1st base encoded is
position 4
the 2nd base encoded is position 5
18.
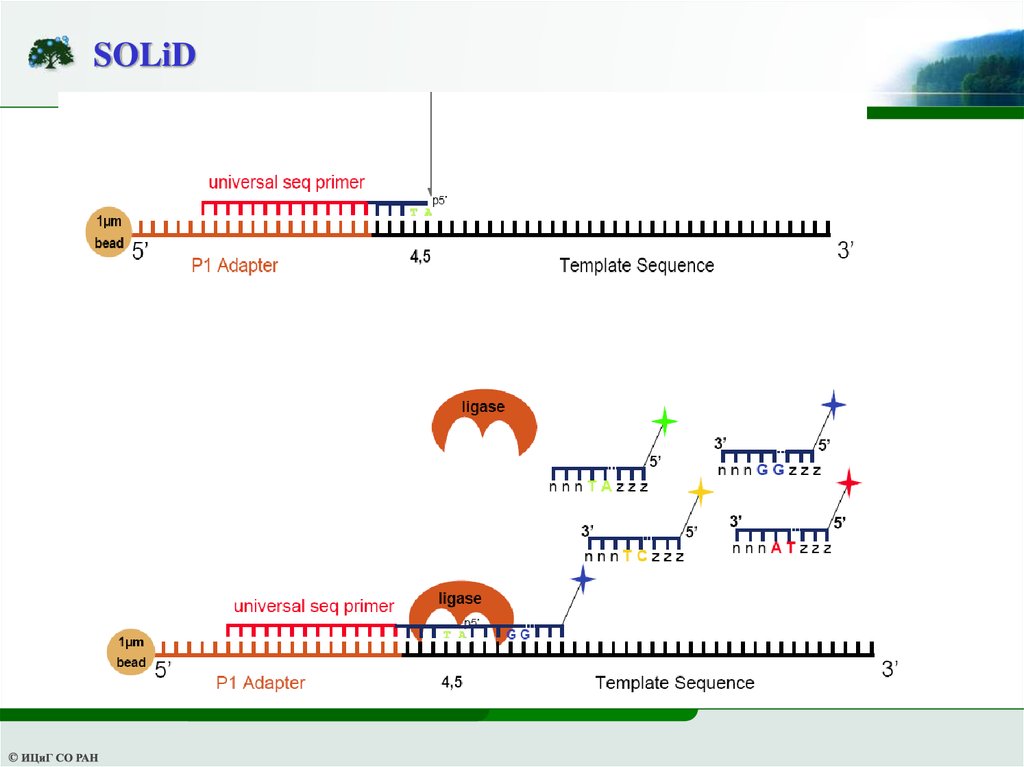
SOLiD19.
SOLiDP1 Adapter
Internal Adapter
P2 Adapter
1µm
bead
25 base
Tag #1
25 base
Tag #2
20.
SOLiD readACGGTCGTCGTGTGCGT
SNP
1st Base
ACGGTCGTCGTGTGCGT
Insertion / Deletion
A
C
G
T
2nd Base
A C G T
21.
SOLiD: SNP и ошибки чтенияSNP
98% raw base accuracy
99,99% consensus accuracy (~ 20x coverage)
error
Reference
SNP 2 colors change
22.
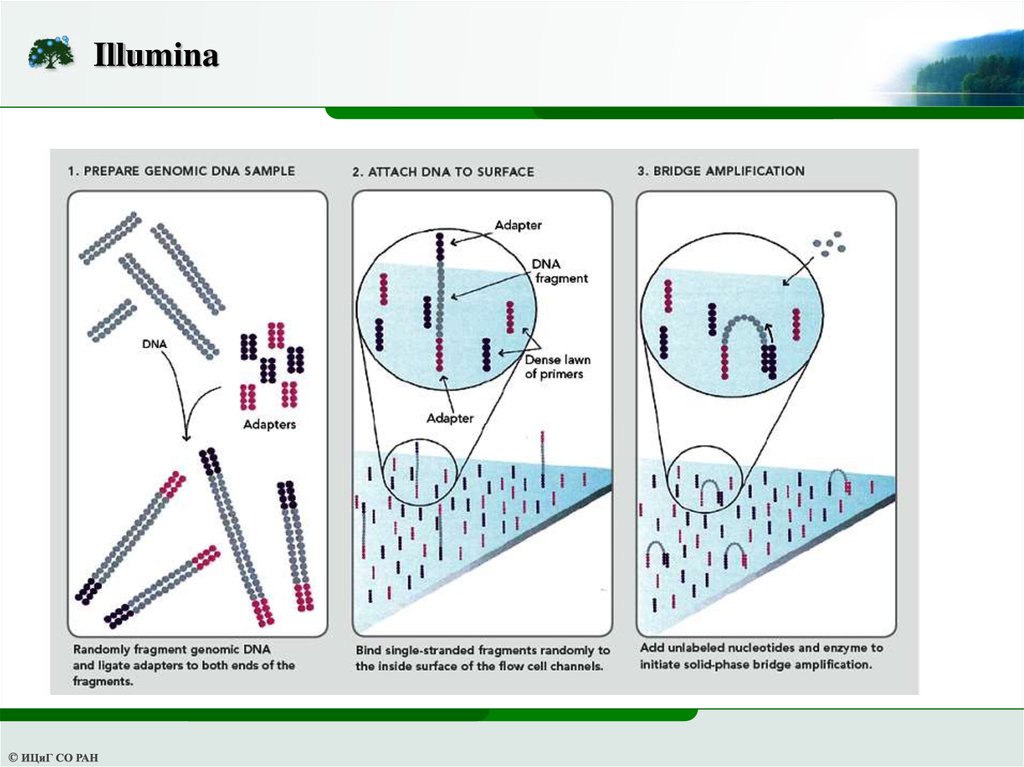
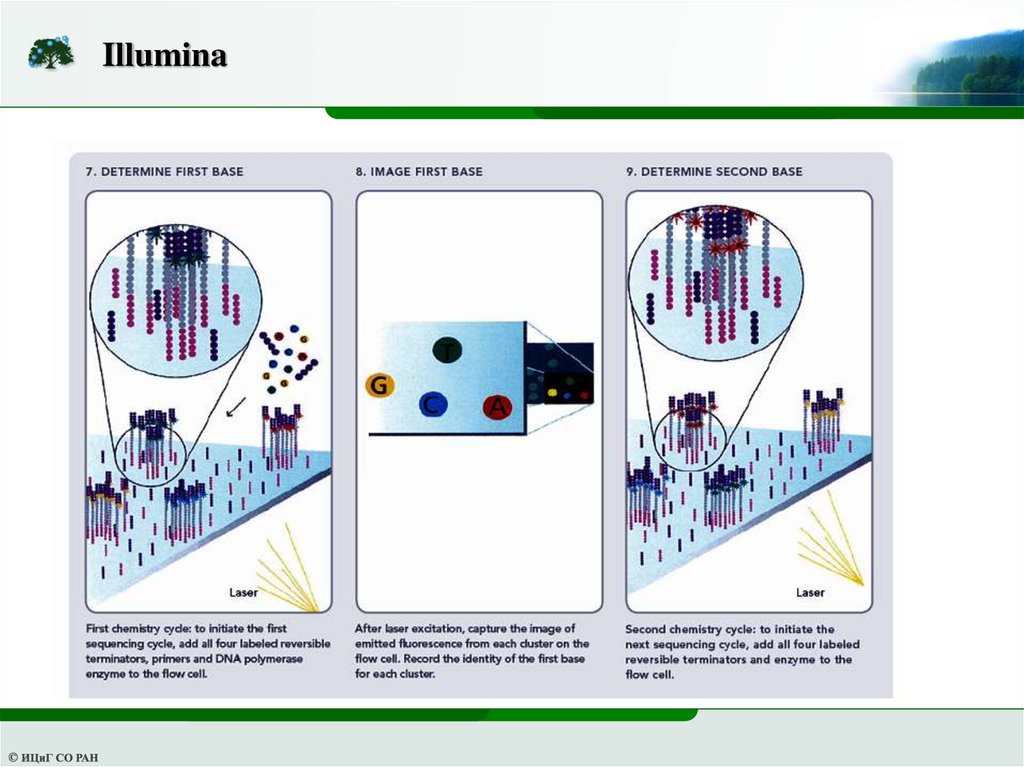
Технология фирмы Illumina – HiSeq, MySeq, NextSeq, NovaSeqОсобенности:
1) Чтения 50-300 п.н. прямые либо парные
2) Есть чтение mate-pair библиотек
3) Доминирующая на рынке (80-90%), применяется везде.
23.
Illumina24.
Illumina25.
Illumina26.
Illumina GA и HiSeq vs NovaSeq27.
Illumina NextSeq и Nova SeqИспользуются только два красителя и длины
волн
28.
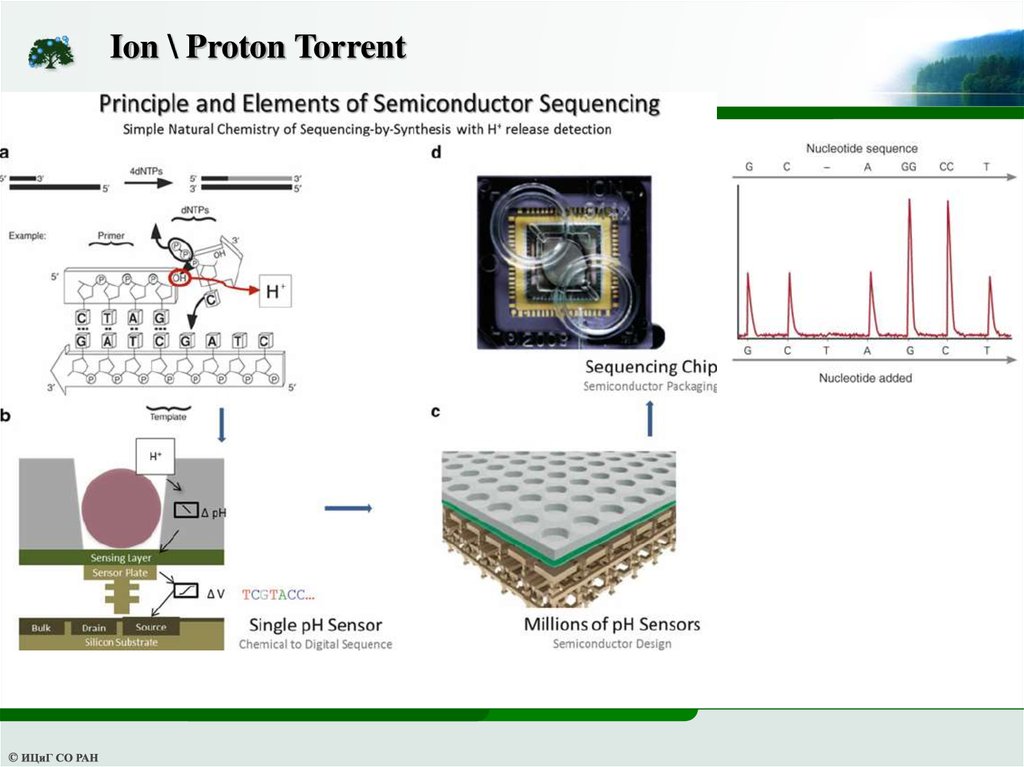
Ion \ Proton TorrentОсобенности:
1) Не используется высокочувствительная оптика
2) Длинное чтение (до 400 п.н. непостоянной длины)
3) Самая быстрая скорость работы
4) Меньшая точность
29.
Ion \ Proton Torrent - E-PCR30.
Ion \ Proton Torrent31.
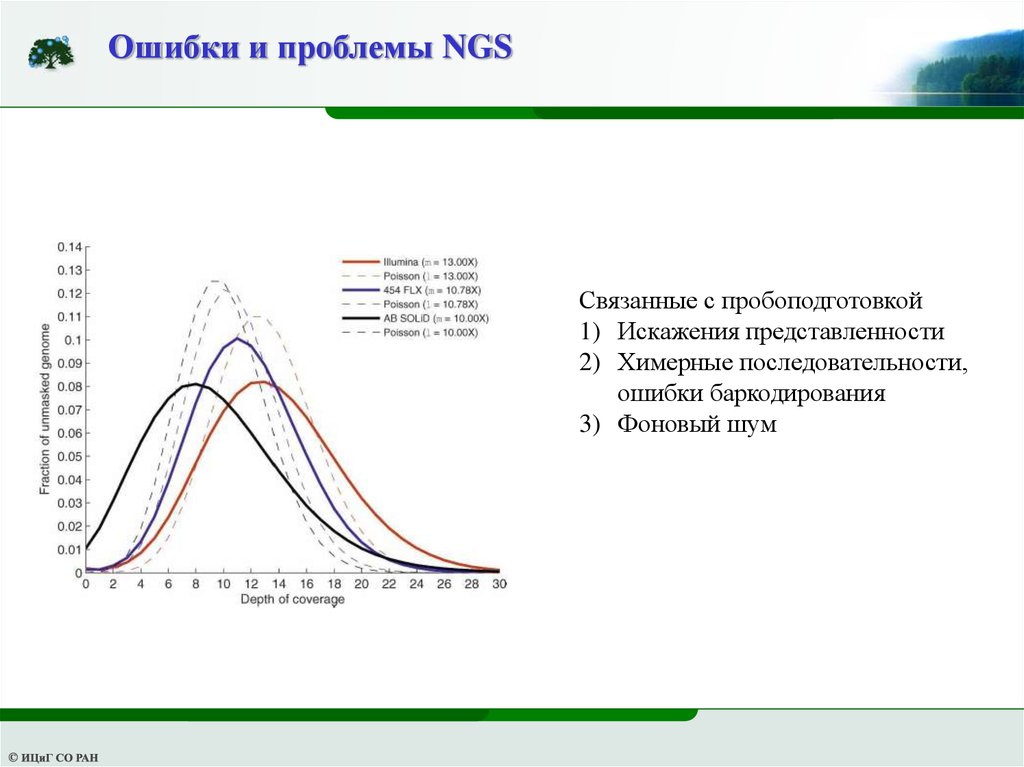
Ошибки и проблемы NGSСвязанные с пробоподготовкой
1) Искажения представленности
2) Химерные последовательности,
ошибки баркодирования
3) Фоновый шум
32.
Ошибки кластеризации и баркодирования, поликлональность33.
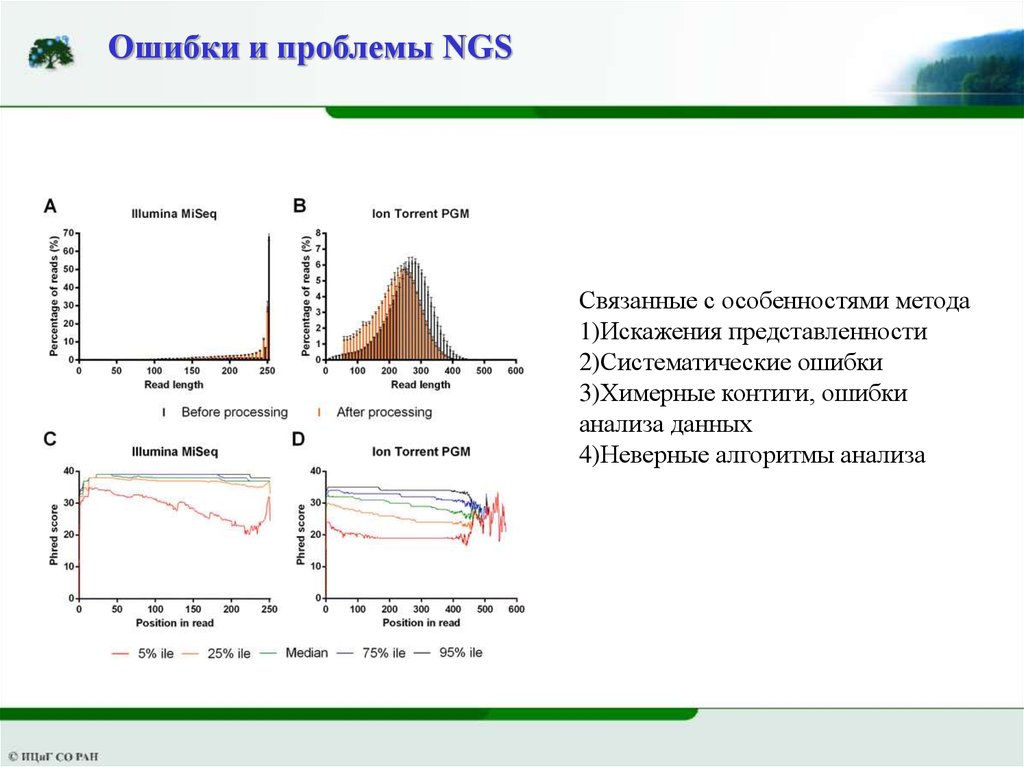
Ошибки и проблемы NGSСвязанные с особенностями метода
1)Искажения представленности
2)Систематические ошибки
3)Химерные контиги, ошибки
анализа данных
4)Неверные алгоритмы анализа
34.
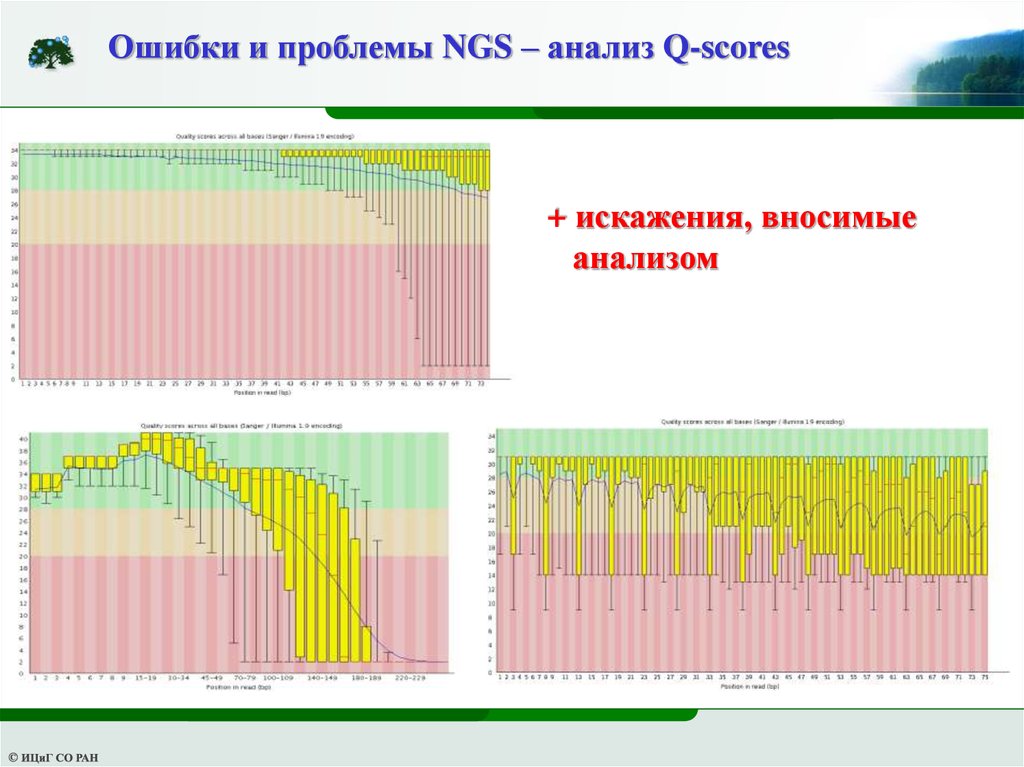
Ошибки и проблемы NGS – анализ Q-scores+ искажения, вносимые
анализом

















 biology
biology








