Similar presentations:
Технология секвенирования генома и сборка генома. Лекция 8
1. Технология секвенирования генома и сборка генома
Лекция 8Многие слайды и материалы используемые в презентации взяты из курса
«Введение в биоинформатику» Санкт-Петербурского государственного
университета
21.12.2019
1
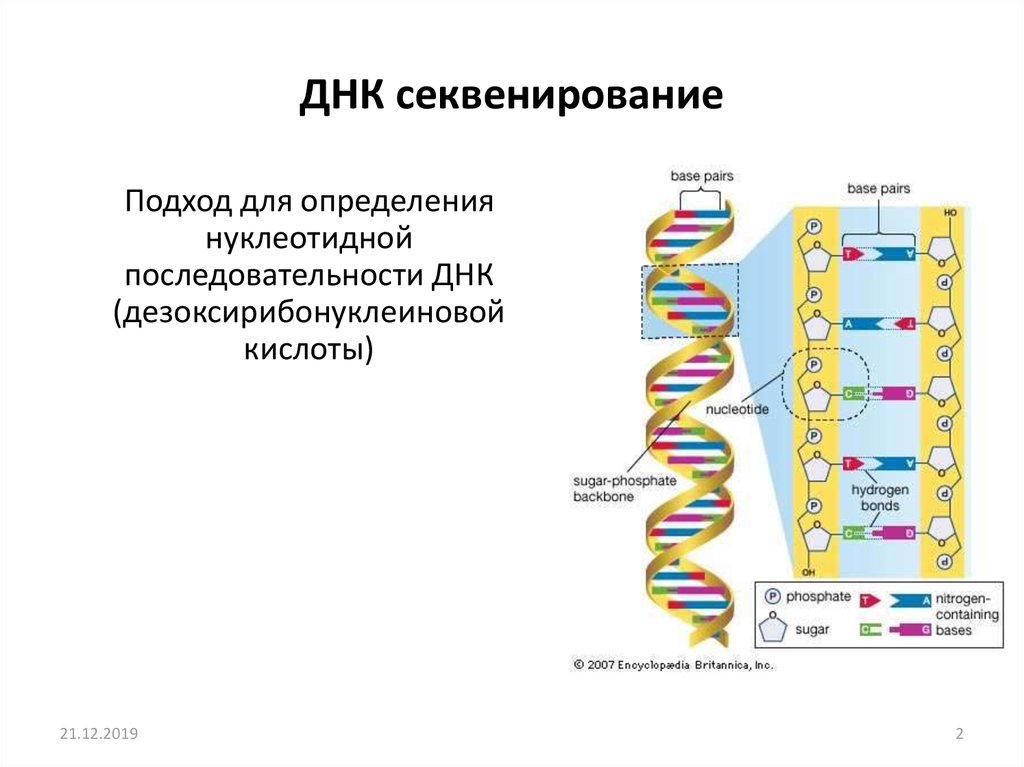
2. ДНК секвенирование
Подход для определениянуклеотидной
последовательности ДНК
(дезоксирибонуклеиновой
кислоты)
21.12.2019
2
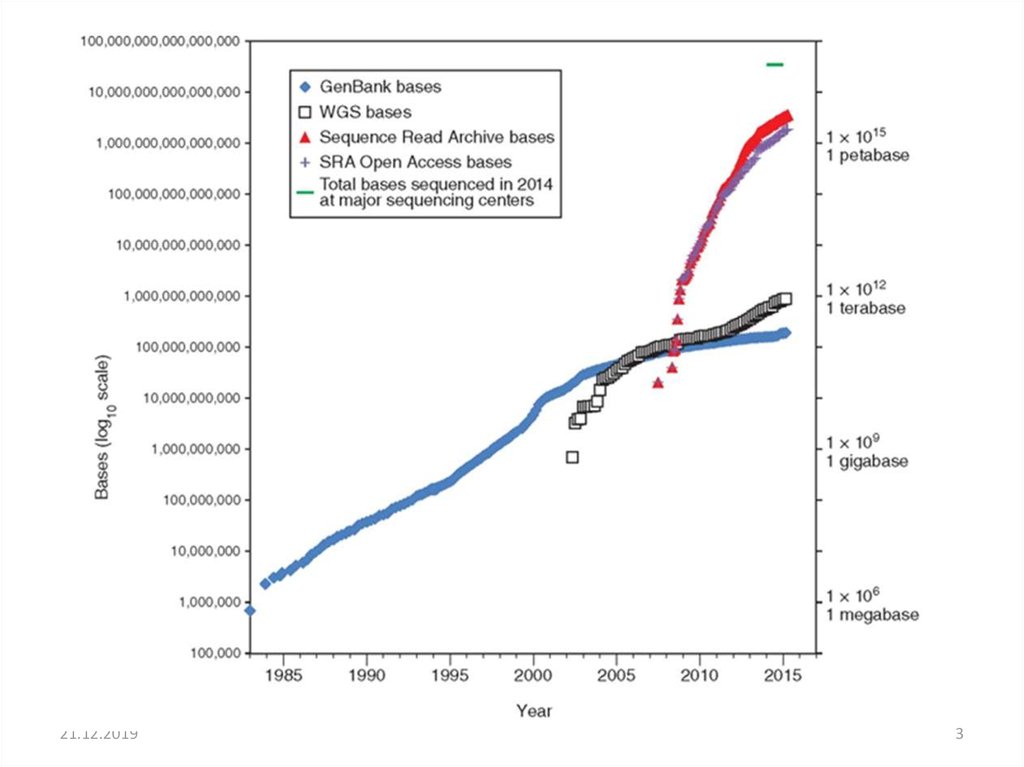
3.
21.12.20193
4.
21.12.20194
5. Применение NGS
ПрименениеРешаемая задача
Полногеномное секвенирование de novo
Реконструкция работы клетки и организма на
молекулярном уровне, эволюционная геномика
Полногеномное повторное секвенирование
Метагеномное секвенирование
Поиск генетических вариаций
Исследование биоценоза, поиск новых видов
живых систем
Исследование генной экспрессии, аннотация
генома
Исследование генной экспрессии
Поиск генетических вариаций
Исследование профиля ДНК
Секвенирование транскриптомов
Секвенирование малых РНК
Таргетное секвенирование
Секвенирование обработанной бисульфидом
ДНК
Секвенирование иммунопреципитированного
хроматина (ChIP)
Секвенирование единичных клеток
21.12.2019
Полногеномное картирование ДНК-белковых
взаимодействий
Исследование генной экспрессии,
секвенирование некультивируемых бактерий
5
6. Основные термины
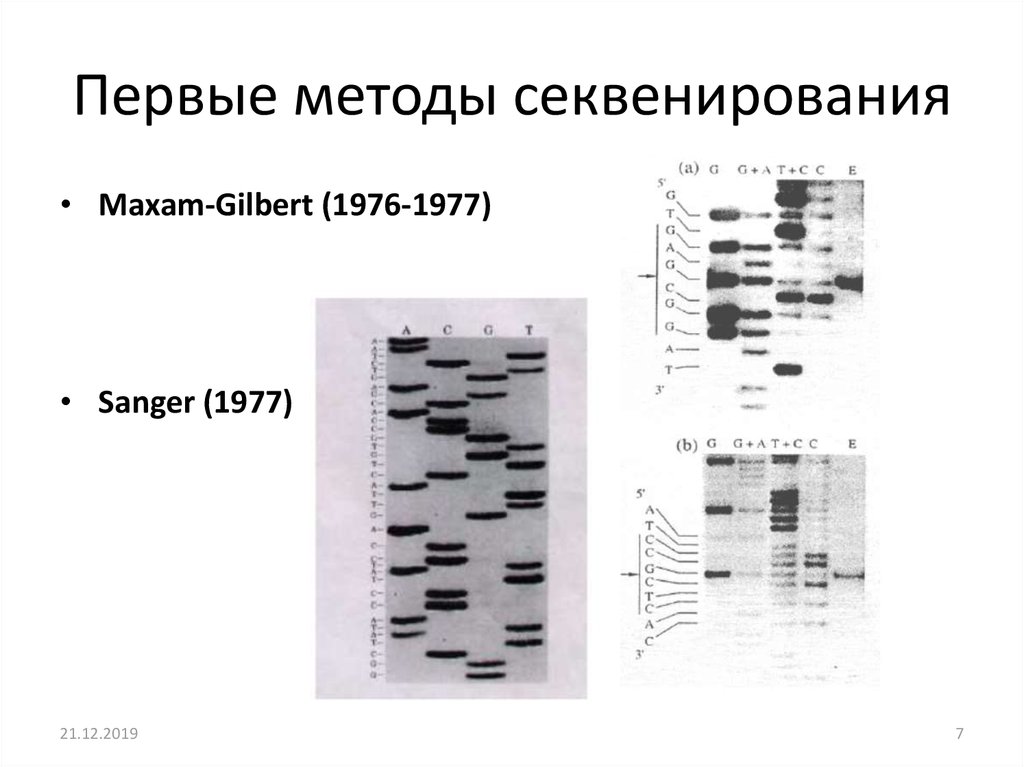
Первые методы секвенирования• Maxam-Gilbert (1976-1977)
• Sanger (1977)
21.12.2019
7
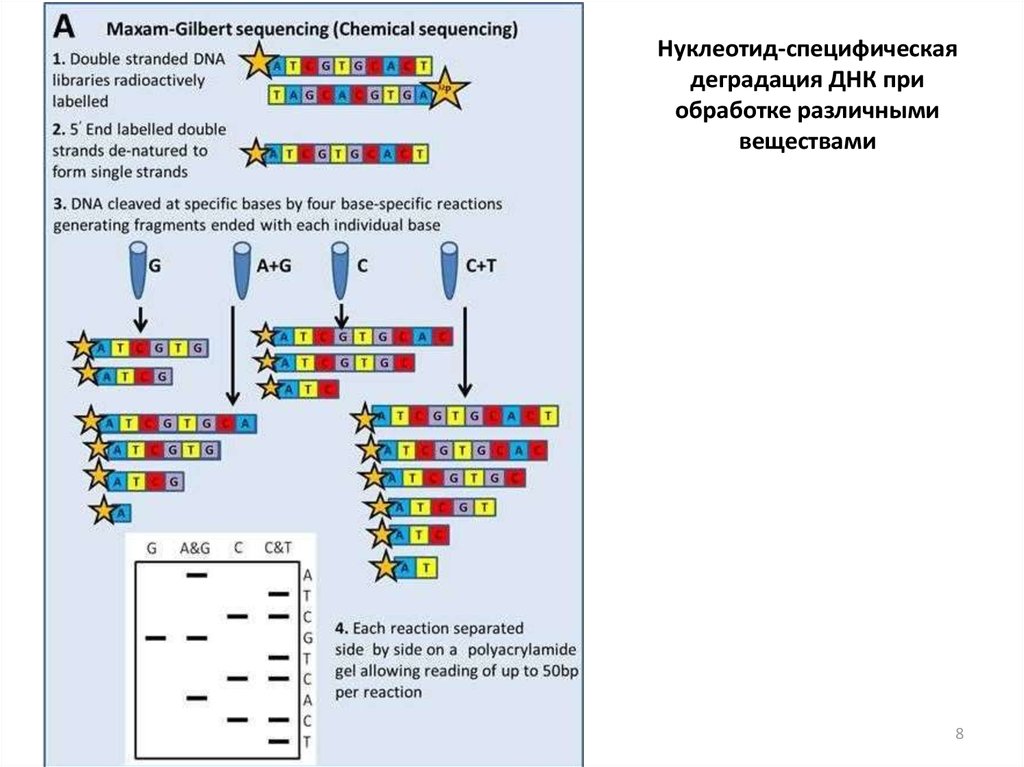
7. Первые методы секвенирования
Нуклеотид-специфическаядеградация ДНК при
обработке различными
веществами
21.12.2019
8
8.
Секвенирование по Сенгеру(Золотой стандарт)
Длинна секвенирования:
300-1000 bp
Ошибки: 0.1-1%
Phi X 174 (ΦX174) бактериофаг был
первым секвенированным ДНК геномом
(5386 нуклеотидов) в 1977 году
21.12.2019
9
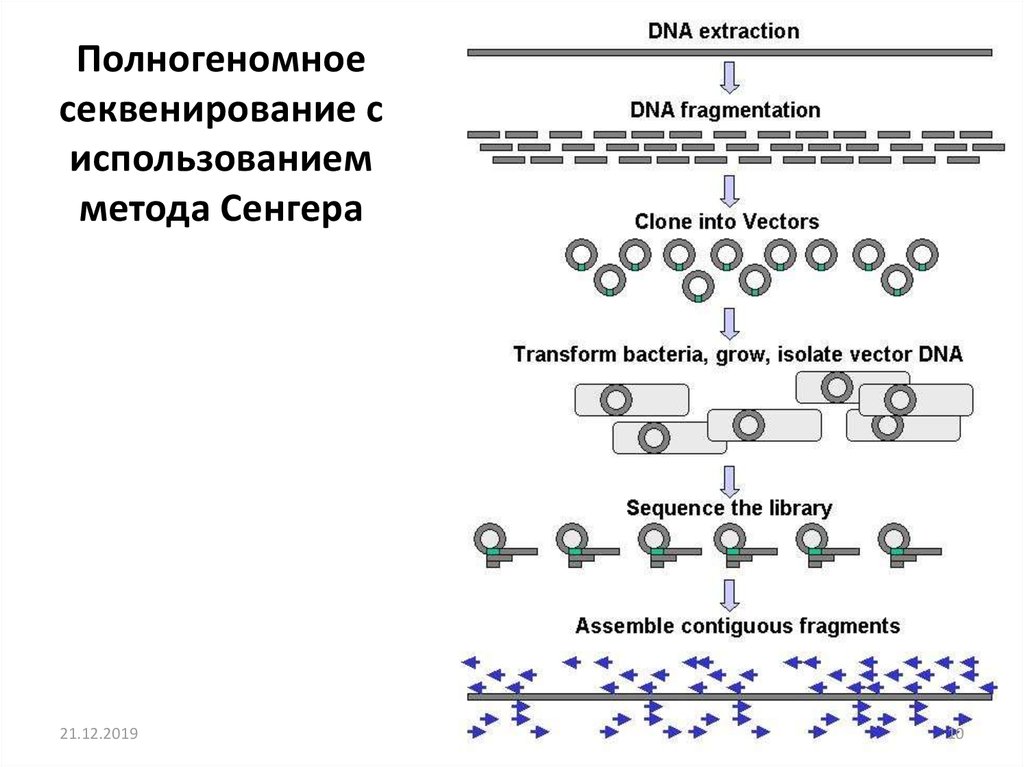
9. Секвенирование по Сенгеру (Золотой стандарт)
Полногеномноесеквенирование с
использованием
метода Сенгера
21.12.2019
10
10. Полногеномное секвенирование с использованием метода Сенгера
Проект геном человекаРазмер генома – 3.2 Гб
Длительность – 10 лет
1990 – 2000
Цена – 3 млрд. $
Метод - секвенирование
по Сенгеру
21.12.2019
11
11. Проект геном человека
Секвенирование по СенгеруПлюсы:
• Относительно низкий уровень ошибок
• Удобное и дешевое секвенирование небольших
фрагментов генома (16S РНК, Hsp65, и т.д.)
Минусы:
• Высокая стоимость полногеномного секвенирования
• Трудоемкость
• Низкая производительность
21.12.2019
12
12. Секвенирование по Сенгеру
21.12.201913
13.
New Generation SequencingПлюсы:
• Простая подготовка ДНК библиотек (пробоподготовка)
• Высокая производительность
• Низкая стоимость секвенирования
Минусы:
• Короткие риды
• Относительно высокий уровень ошибок
21.12.2019
14
14. New Generation Sequencing
Основные принципы подготовки ДНК библиотек1.
2.
3.
4.
21.12.2019
Фрагментация ДНК
Отбор размера
Лигирование адаптора
Амплификация
библиотеки
15
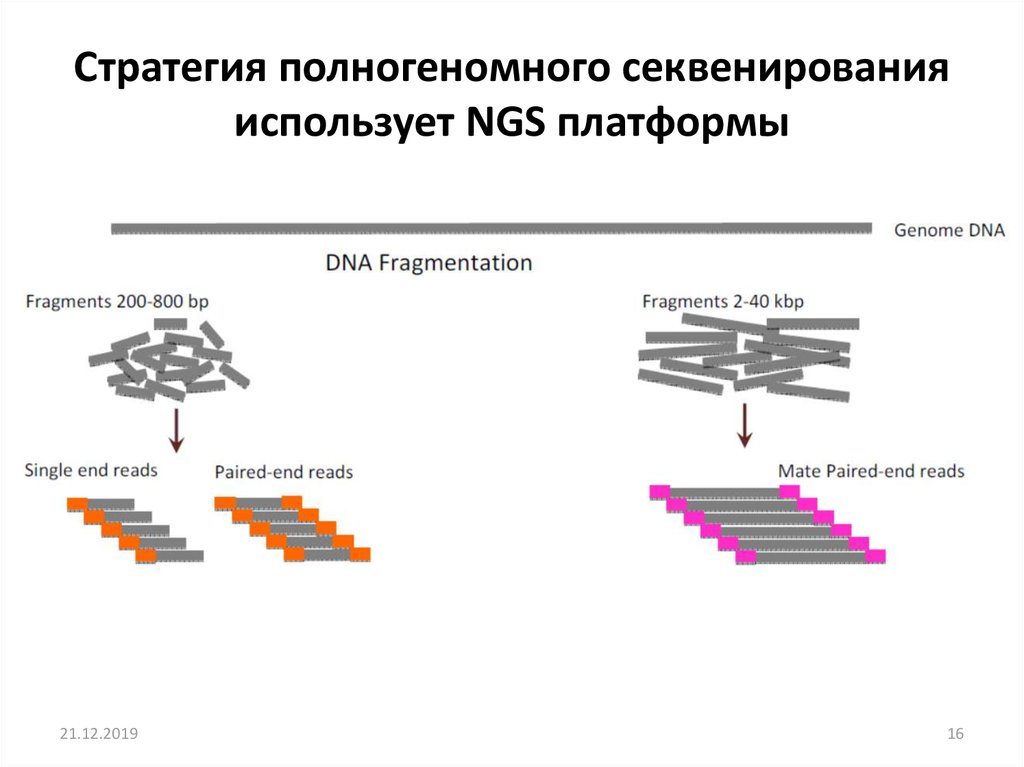
15. Основные принципы подготовки ДНК библиотек
Стратегия полногеномного секвенированияиспользует NGS платформы
21.12.2019
16
16. Стратегия полногеномного секвенирования использует NGS платформы
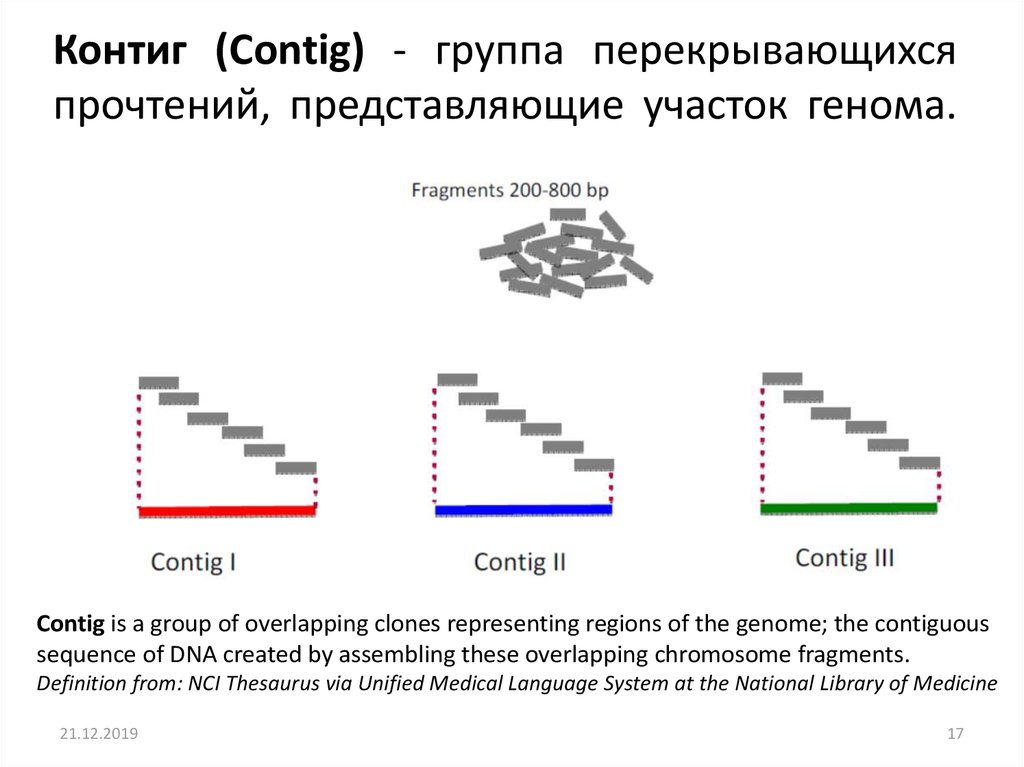
Контиг (Contig) - группа перекрывающихсяпрочтений, представляющие участок генома.
Contig is a group of overlapping clones representing regions of the genome; the contiguous
sequence of DNA created by assembling these overlapping chromosome fragments.
Definition from: NCI Thesaurus via Unified Medical Language System at the National Library of Medicine
21.12.2019
17
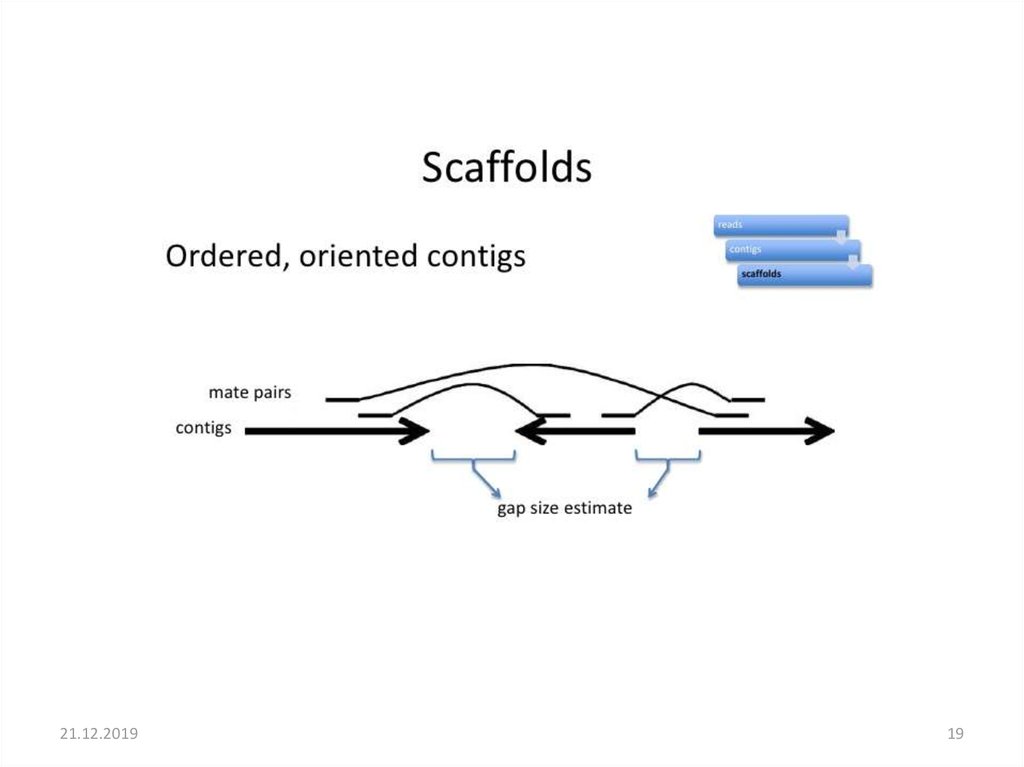
17. Контиг (Contig) - группа перекрывающихся прочтений, представляющие участок генома.
Scaffold (Скафолд) – реконструированная часть генома,полученная в результате анализа библиотек большого
размера и правильного взаимного расположения контигов
Scaffold is a portion of the genome sequence reconstructed from end-sequenced
whole-genome shotgun clones. Scaffolds are composed of contigs and gaps.
Definition from: http://genome.jgi-psf.org/help/scaffolds.html
21.12.2019
18
18. Scaffold (Скафолд) – реконструированная часть генома, полученная в результате анализа библиотек большого размера и правильного
21.12.201919
19.
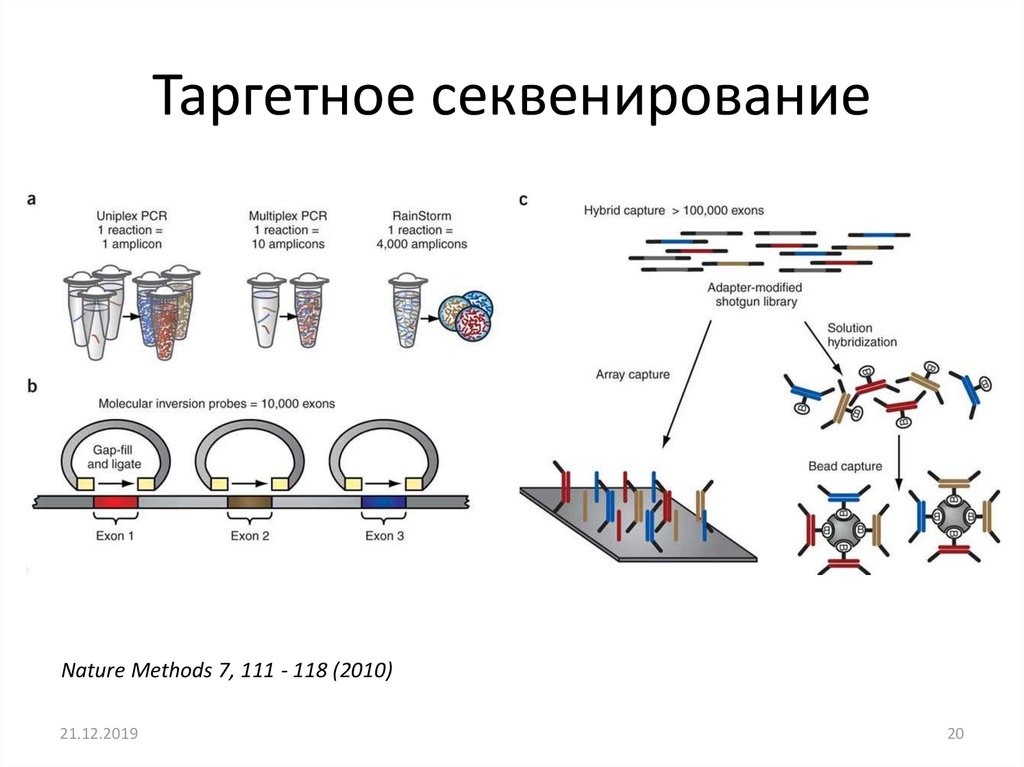
Таргетное секвенированиеNature Methods 7, 111 - 118 (2010)
21.12.2019
20
20. Таргетное секвенирование
Индексирование (Баркодинг)Можно за один запуск секвенатора прочитать
несколько геномов или геномных участков
Индексы – короткие олигонуклеотиды с
различной
последовательностью,
которые
фланкируют ДНК библиотеки и секвенируются
вместе с целевой ДНК. На основе известной
индексной
последовательности
можно
дифференцировать
несколько
образцов
секвенированных в одно время.
21.12.2019
21
21. Индексирование (Баркодинг)
Примеры индексов21.12.2019
22
22. Примеры индексов
Платформы21.12.2019
23
23. Платформы
21.12.201924
24.
454 Sequencing TechnologyФрагментация ДНК
Подготовка библиотеки
Пришивание адапторов к молекулам
ДНК с двух концов.
21.12.2019
25
25. 454 Sequencing Technology
Один фрагмент = одна бусина (bead)Библиотека фрагментов ДНК прикрепляется к
бусинам после денатурации ДНК. Каждая бусина
имеет уникальный фрагмент библиотеки. Шарики
эмульгируют с реагентами амплификации в смеси
вода-в-масле.
emPCR: Эмульсионная ПЦР-амплификация
Амплификация ведется в эмульсии параллельно,
чтобы создать миллионы клонных копий каждого
фрагмента библиотеки на каждом шарике. Если
фрагмент не присоединяется к шарику, то он
остается гладким.
21.12.2019
Секвенирование: один шарик = один рид
Шарик помещается в лунку, где дизайн поверхности
допускает только один шарик. Отдельные
нуклеотиды протекают последовательно через
лунки.
Каждое
включение
нуклеотида,
комплементарное к матричной нити, приводит к
хемилюминесцентному
световому
сигналу,
записанному камерой.
26
26.
Секвенирование начинается сприсоединения праймера, потом
присоединение комплементарного
нуклеотида приводит к
высвобождению пирофосфата,
который взаимодействуя с
сулфирилазой и люциферазой
приводит к образованию светового
сигнала, детектируемого камерой.
По интенсивности сигнала
определяется какое количество
нуклеотидов присоединяется. При
этом зная какие нуклеотиды
подаются в текущее время
определяют последовательность
ДНК.
21.12.2019
27
27.
21.12.201928
28.
Ion TorrentПодготовка библиотеки похожа на Roche 454
• фрагментация ДНК
• Прикрепление адаптера
• Эмульсионная ПЦР
Технология секвенирования отличается
21.12.2019
29
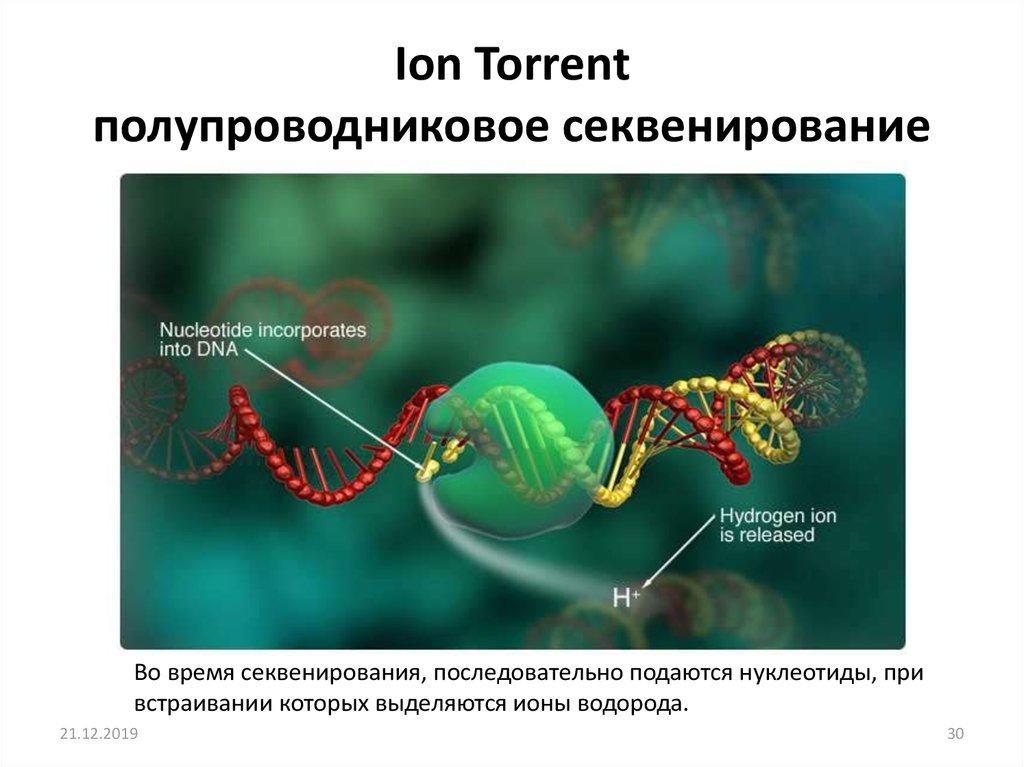
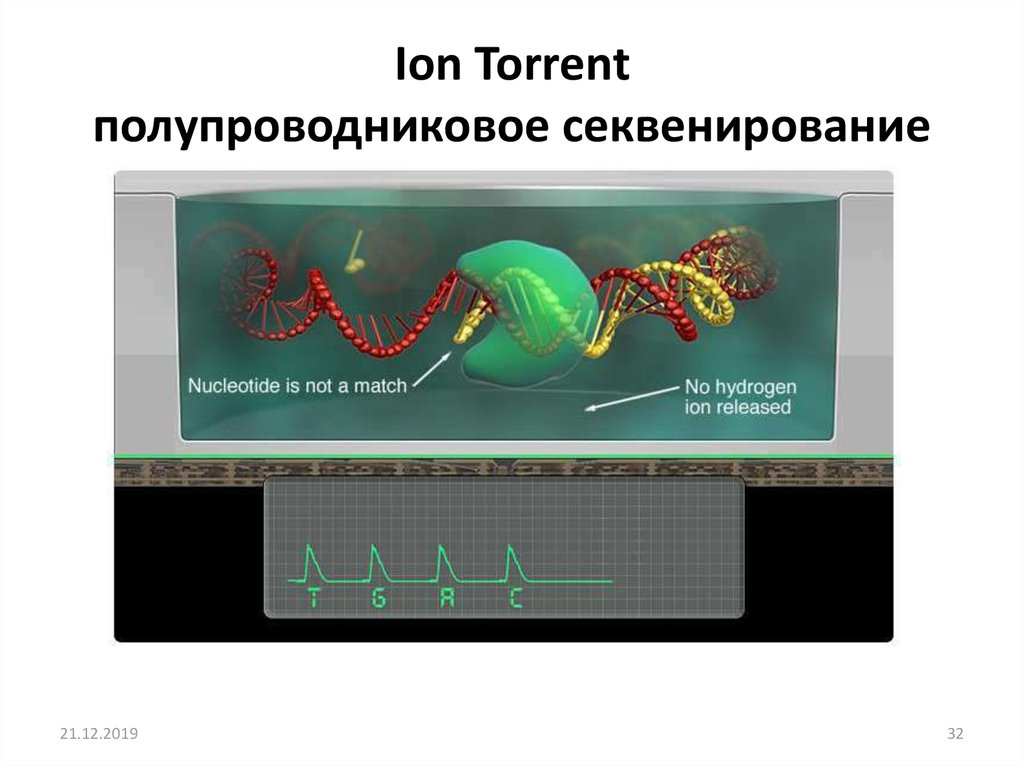
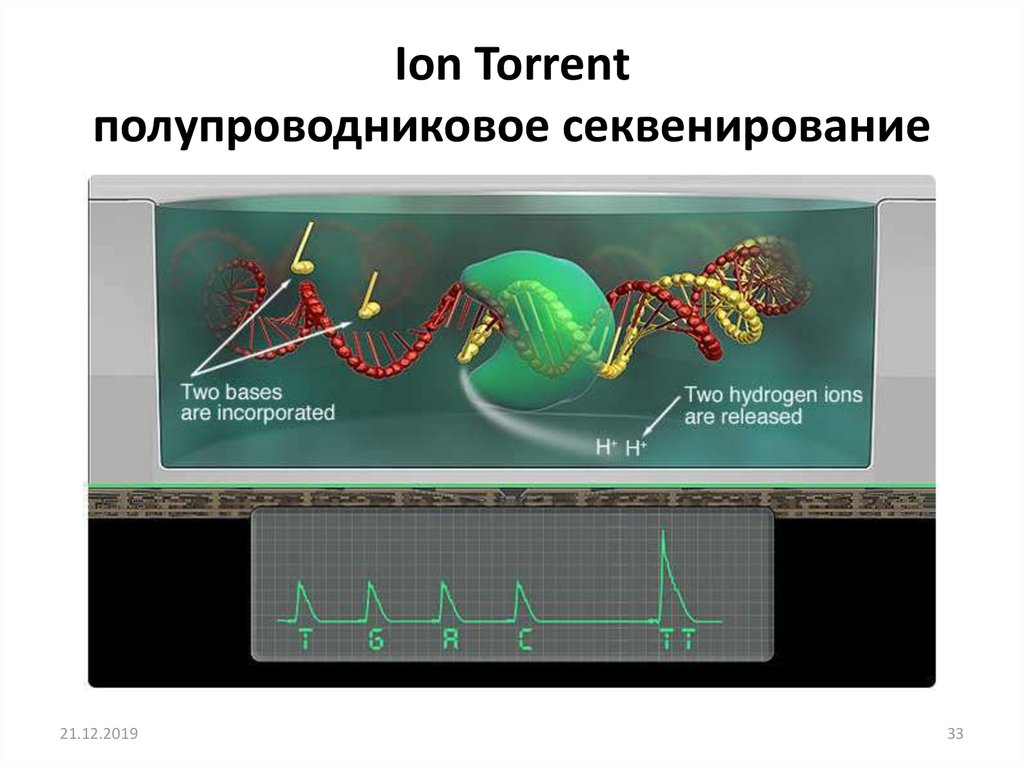
29. Ion Torrent
полупроводниковое секвенированиеВо время секвенирования, последовательно подаются нуклеотиды, при
встраивании которых выделяются ионы водорода.
21.12.2019
30
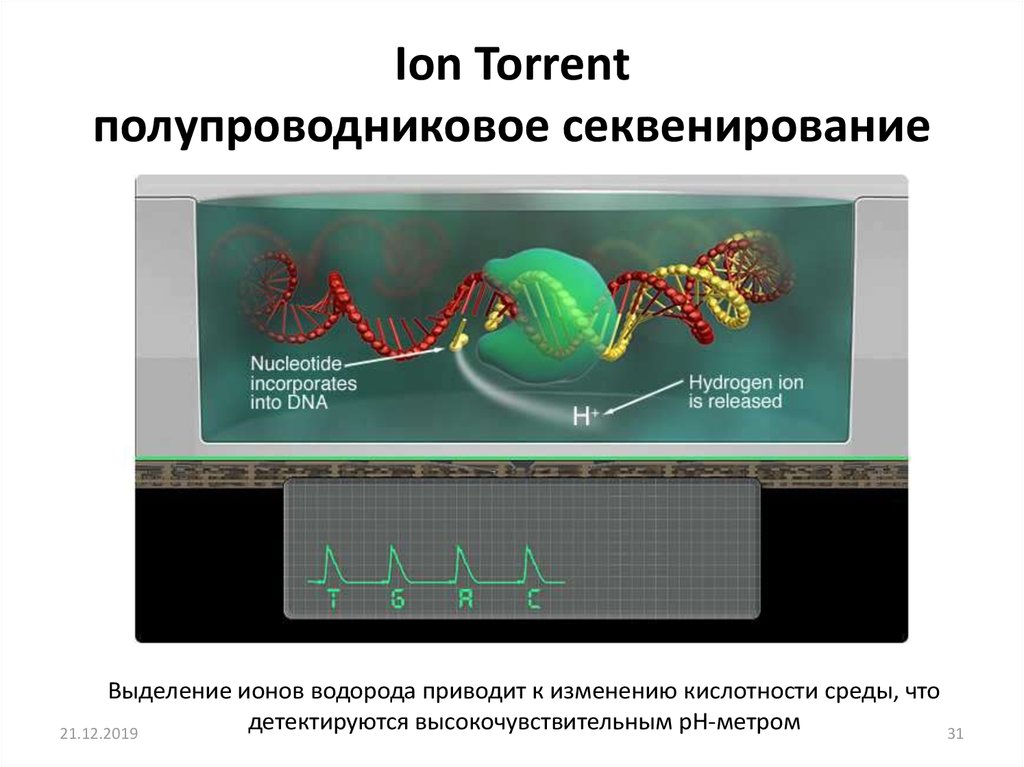
30. Ion Torrent полупроводниковое секвенирование
Выделение ионов водорода приводит к изменению кислотности среды, чтодетектируются высокочувствительным pH-метром
21.12.2019
31
31. Ion Torrent полупроводниковое секвенирование
21.12.201932
32. Ion Torrent полупроводниковое секвенирование
21.12.201933
33. Ion Torrent полупроводниковое секвенирование
21.12.201934
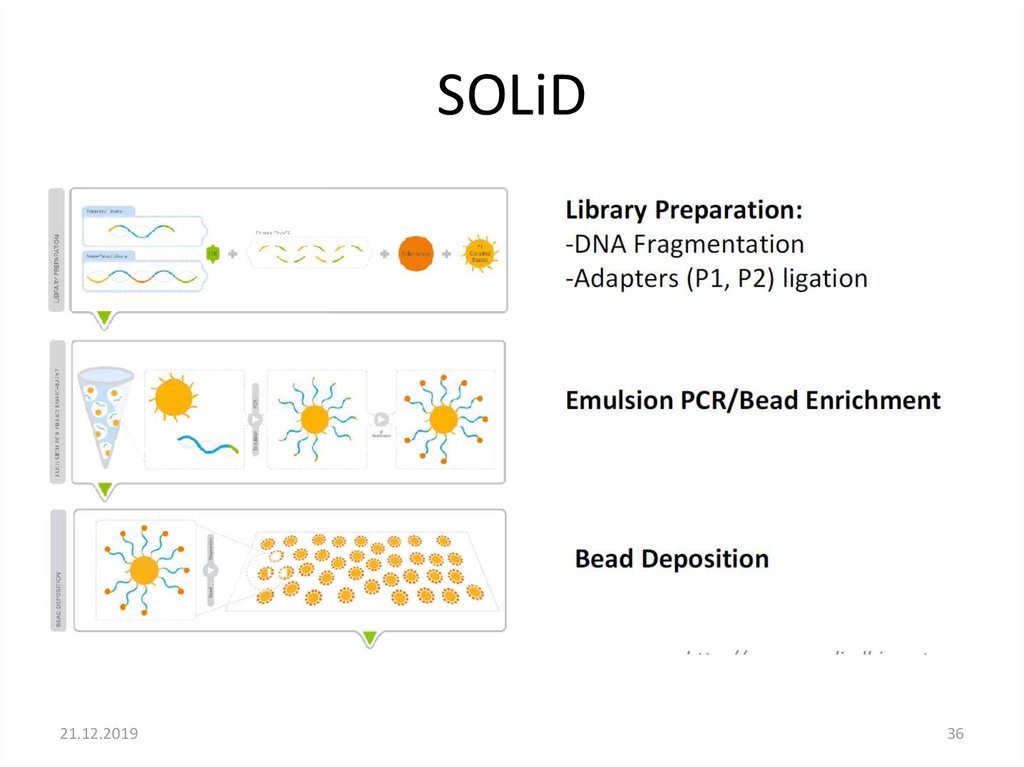
34.
SOLiDПодготовка библиотеки похожа на Roche 454
• фрагментация ДНК
• Прикрепление адаптера
• Эмульсионная ПЦР
• Технология секвенирования отличается секвенирование путем лигирования
олигонуклеотидов
21.12.2019
35
35. SOLiD
21.12.201936
36. SOLiD
Происходит последовательное взаимодействие олигонуклеотида, состоящего изспецифичного динуклеотида, пяти неспецифичных нуклеотидов и флуорафора, что
приводит к специфическому связыванию динуклеотида (лигирование) и отщеплению
флуорафора и детекция флуоресцентного сигнала.
21.12.2019
37
37. SOLiD
Для борьбы с неспецифичными нуклеотидами используют новые праймеры,которые короче на 1,2,3,4 нуклеотида (всего 5 раундов секвенирования). Это
увеличивает точность секвенирования, т.к. каждый нуклеотид прочитывается
дважды, но длина ридов небольшая.
21.12.2019
38
38. SOLiD
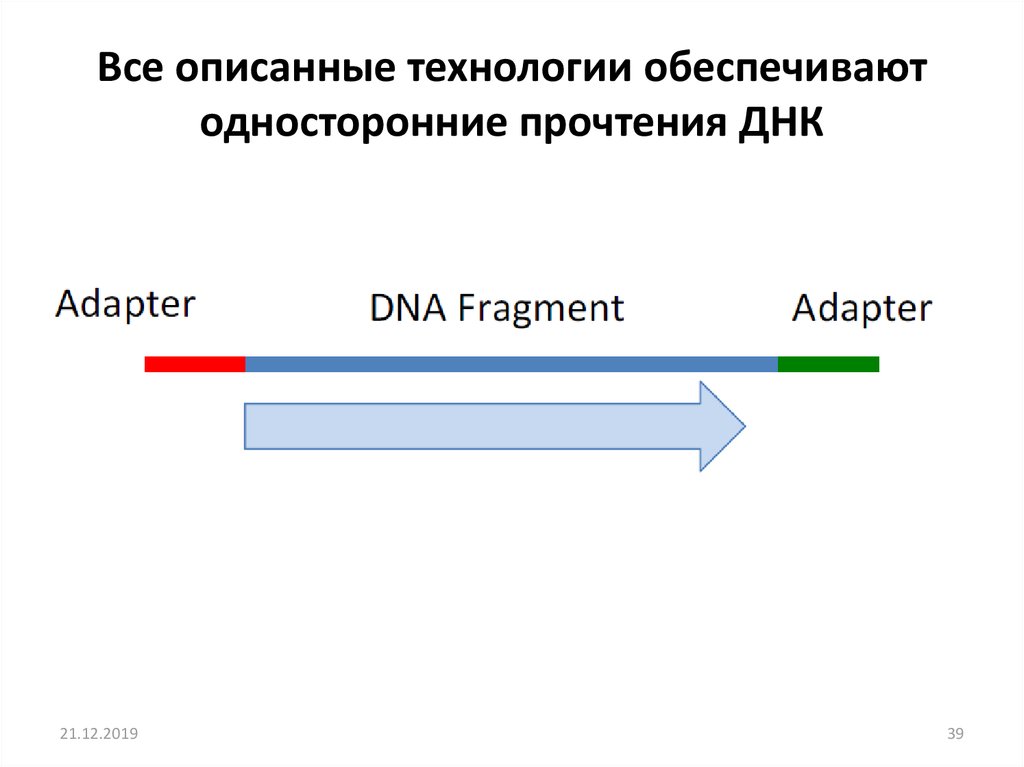
Все описанные технологии обеспечиваютодносторонние прочтения ДНК
21.12.2019
39
39. Все описанные технологии обеспечивают односторонние прочтения ДНК
21.12.201940
40.
Подготовка библиотеки ДНК21.12.2019
41
41. Подготовка библиотеки ДНК
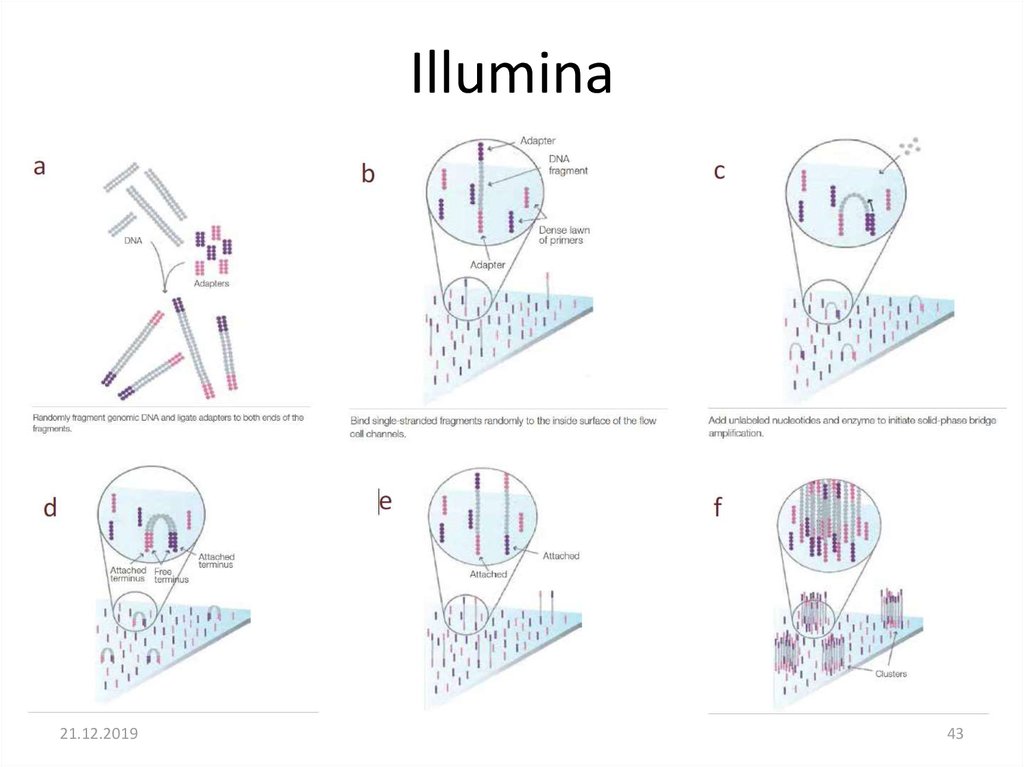
Illumina• Гибридизация ДНК-библиотек
• Генерация кластеров (ПЦР)
• Секвенирование синтезом
http://www.youtube.com/watch?v=HMyCqWhwB8E
21.12.2019
42
42. Illumina
21.12.201943
43.
Illumina21.12.2019
44
44.
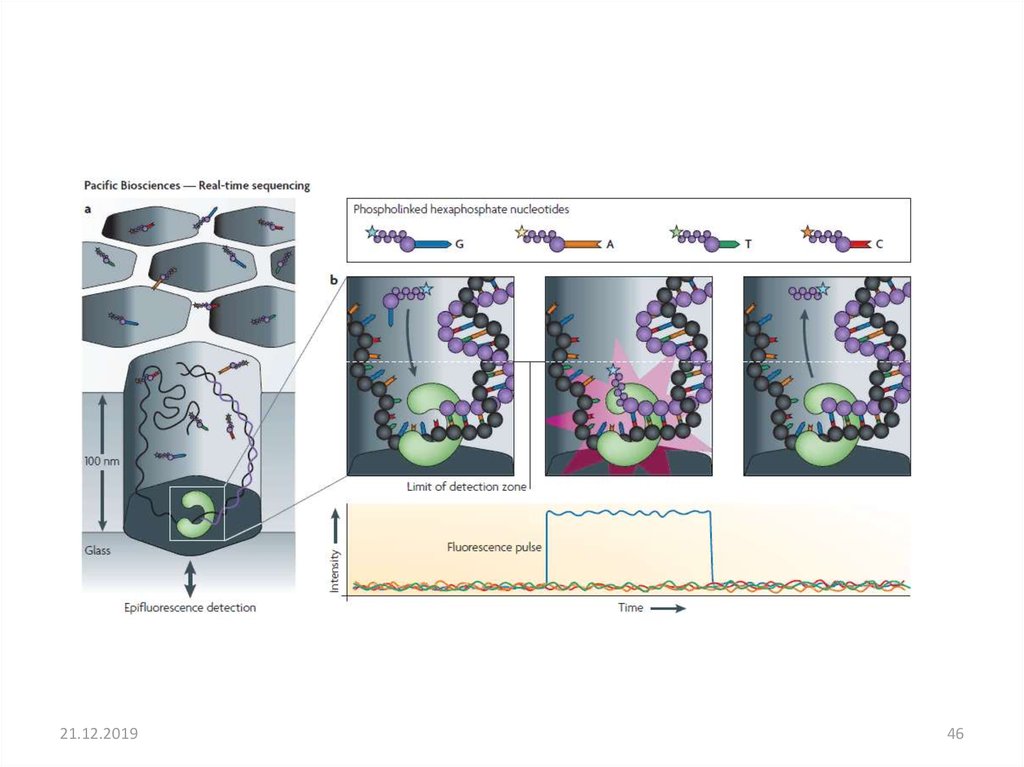
Pacific Biosciencessingle molecule real-time (SMRT) sequencing
Одномолекулярное секвенирование в реальном времени
• Секвенировании без амплификации
• Очень длинные риды
– Производит чтения со средней длиной от 10 000 до
15 000 пар оснований, причем самые длинные риды
могут быть более 30 000 пар оснований
21.12.2019
45
45. Pacific Biosciences single molecule real-time (SMRT) sequencing Одномолекулярное секвенирование в реальном времени
21.12.201946
46.
21.12.201947
47.
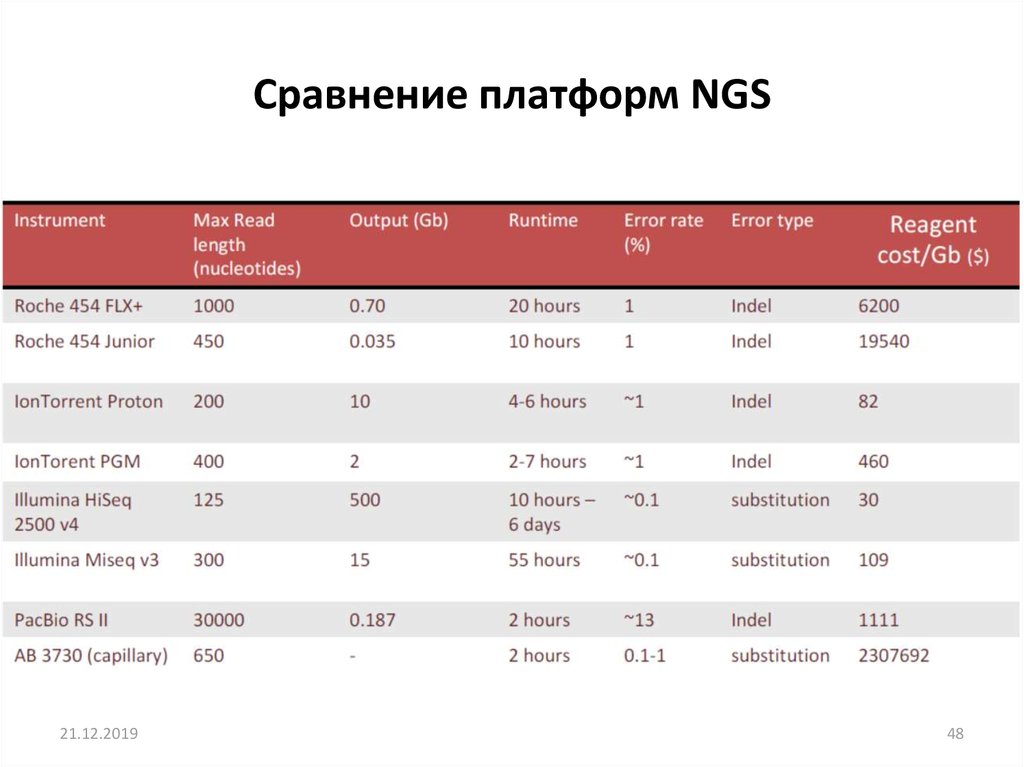
Сравнение платформ NGS21.12.2019
48
48. Сравнение платформ NGS
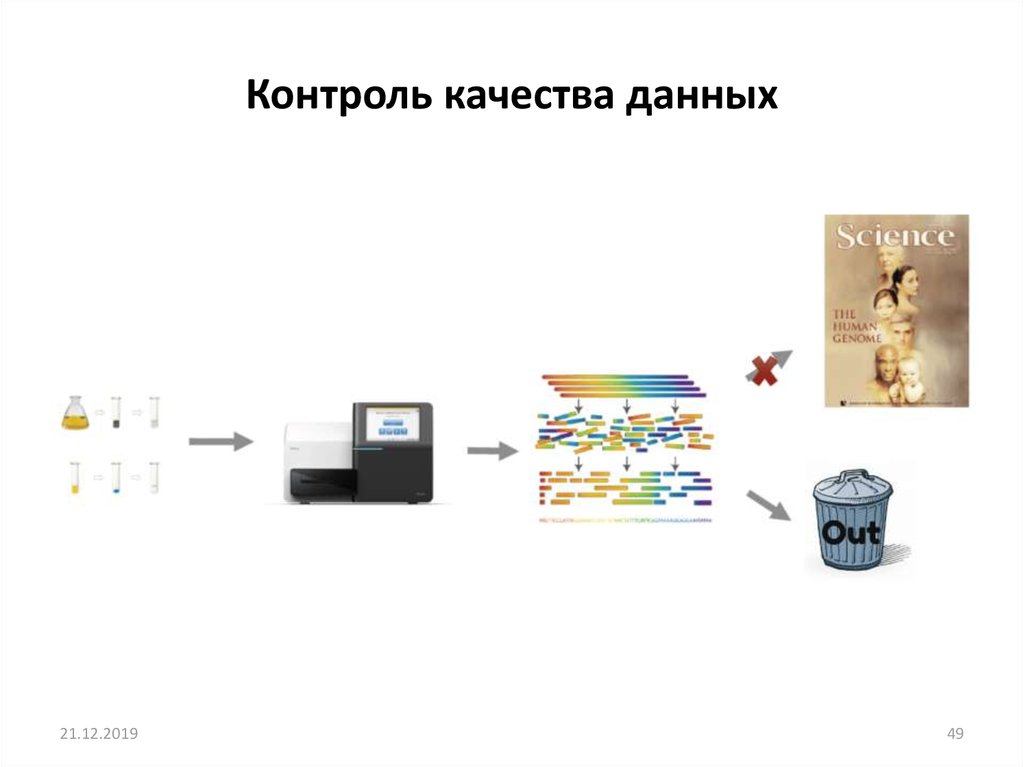
Контроль качества данных21.12.2019
49
49. Контроль качества данных
21.12.201950
50.
Алгоритм контроля качестваПроверка качества
Определение проблемы
Решение проблемы
Проверка качества
Последующий анализ
21.12.2019
51
51. Алгоритм контроля качества
Зачем чистить данные?• Риды низкого качества
• Контаминация (примесь ДНК другого организма)
• Служебные последовательности (адаптеры, индексы)
• Артефакты создания библиотек (некоторые
последовательности встречаются чаще, а не
равномерно)
• Различный формат данных
• Человеческий фактор
21.12.2019
52
52. Зачем чистить данные?
FASTA и FASTQ форматыFASTA
FASTQ
1.
2.
3.
4.
Линия начинающаяся с @ содержит идентификатор последовательности
Последовательность
Линия начинающаяся с + заполняется факультативно
Линия с величинами качества прочтения, кодируемые в ASCII формате
21.12.2019
53
53. FASTA и FASTQ форматы
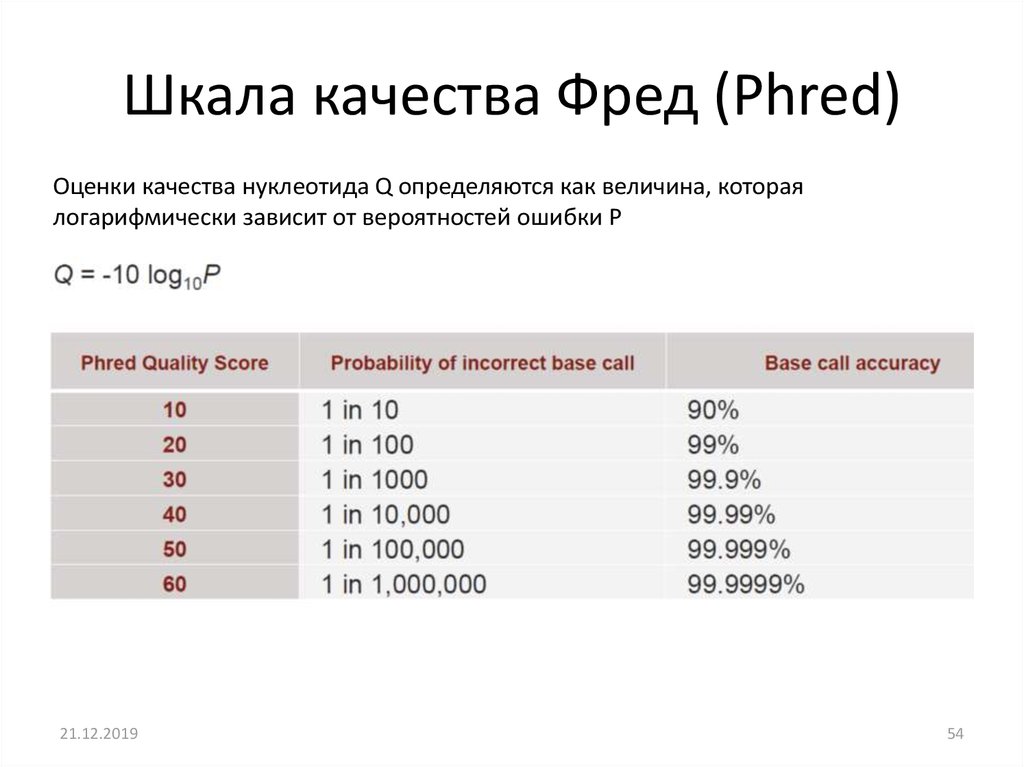
Шкала качества Фред (Phred)Оценки качества нуклеотида Q определяются как величина, которая
логарифмически зависит от вероятностей ошибки P
21.12.2019
54
54. Шкала качества Фред (Phred)
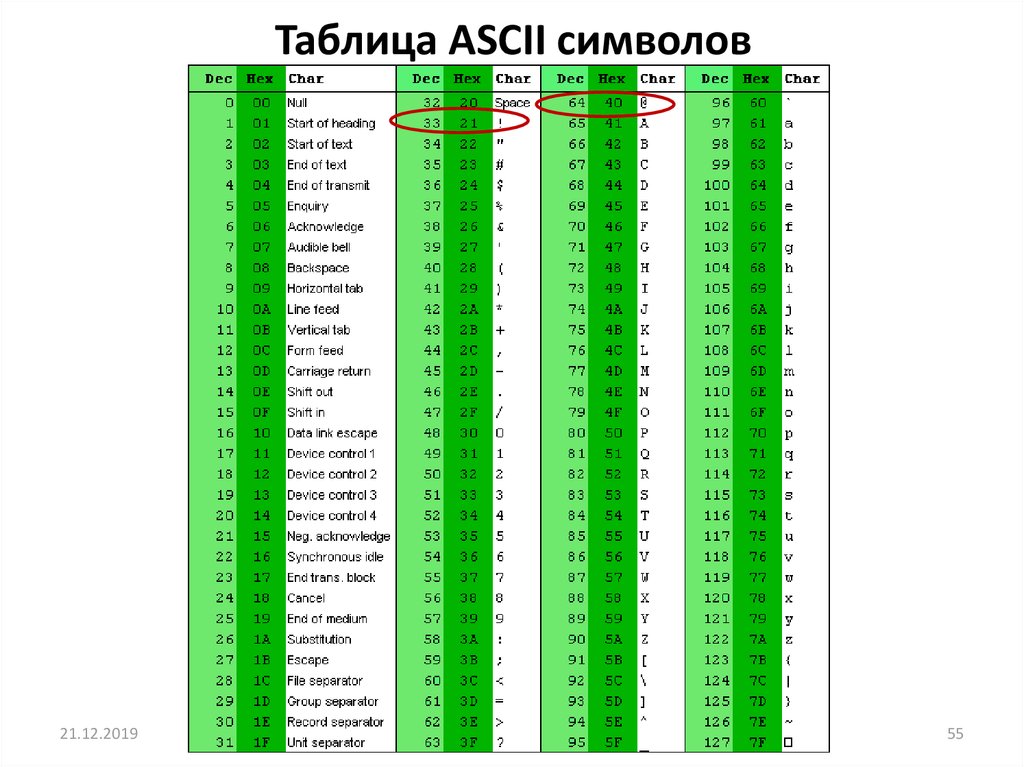
Таблица ASCII символов21.12.2019
55
55. Таблица ASCII символов
Разные Phred шкалы21.12.2019
56
56. Разные Phred шкалы
Cборка генома21.12.2019
57
57. Cборка генома
21.12.201958
58.
FastQC – инструмент для контроля качестваданных
• На вход – исходные данные с секвенатора
• HTML отчет
• Графический интерфейс и версия с командной
строкой
www.bioinformatics.babraham.ac.uk/projects/fastqc
21.12.2019
59
59. FastQC – инструмент для контроля качества данных
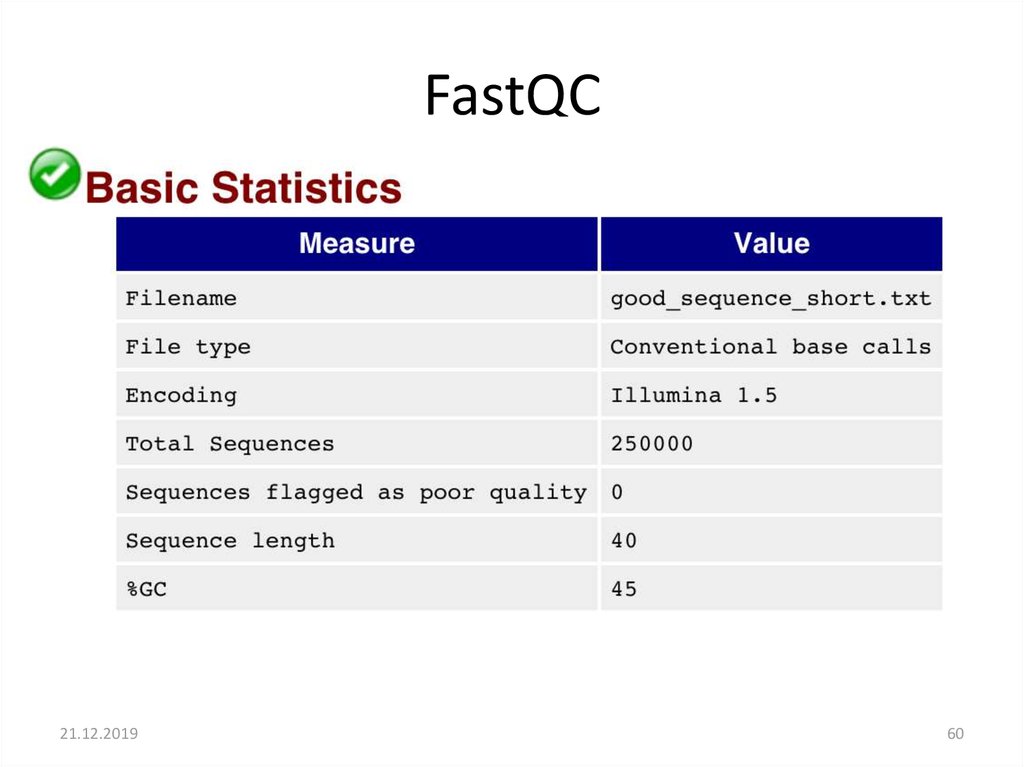
FastQC21.12.2019
60
60. FastQC
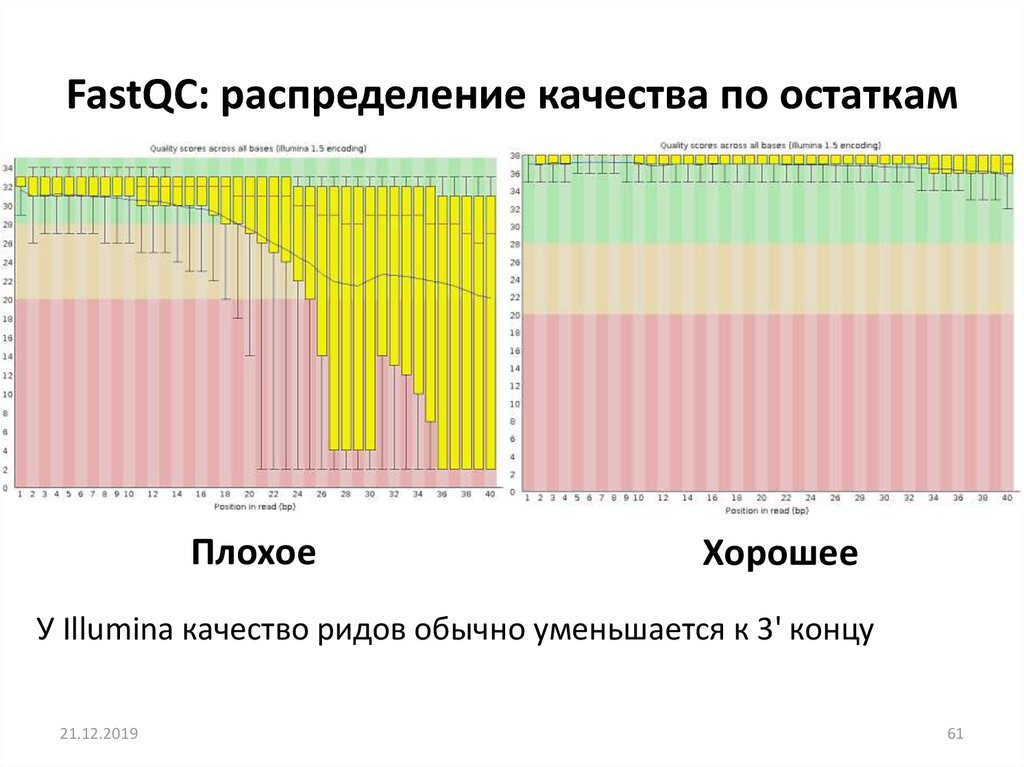
FastQC: распределение качества по остаткамПлохое
Хорошее
У Illumina качество ридов обычно уменьшается к 3' концу
21.12.2019
61
61. FastQC: распределение качества по остаткам
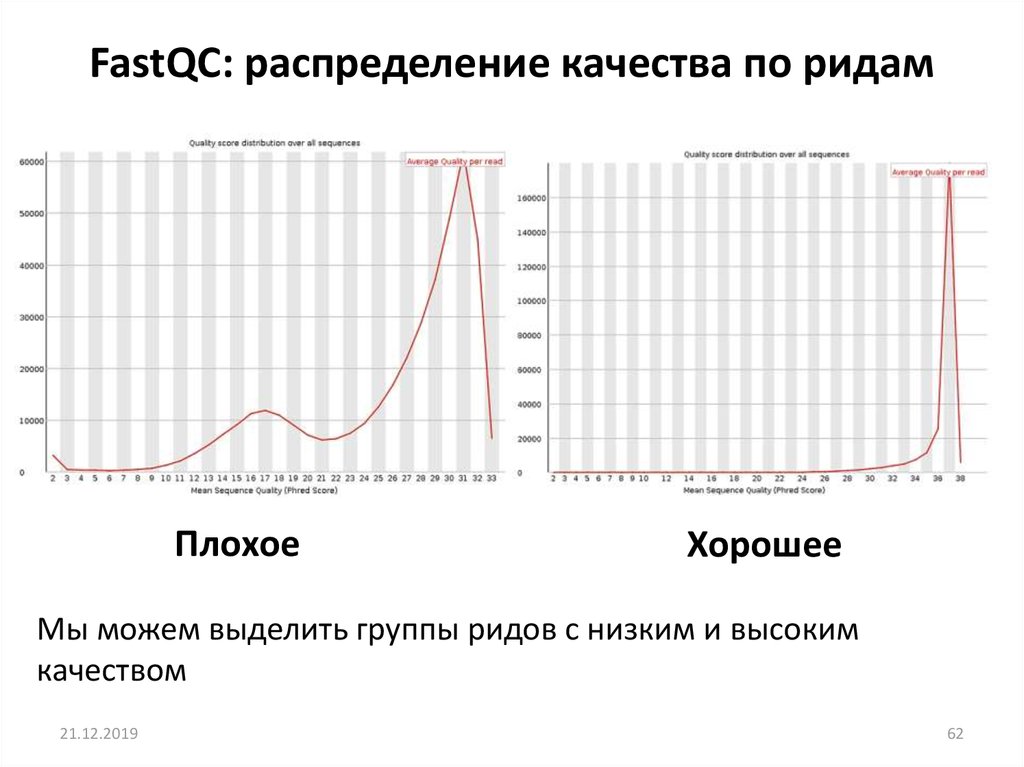
FastQC: распределение качества по ридамПлохое
Хорошее
Мы можем выделить группы ридов с низким и высоким
качеством
21.12.2019
62
62.
FastQC: распределение качества по составу остатковПлохое
Хорошее
Мы можем определить адаптеры или сдвиг
21.12.2019
63
63.
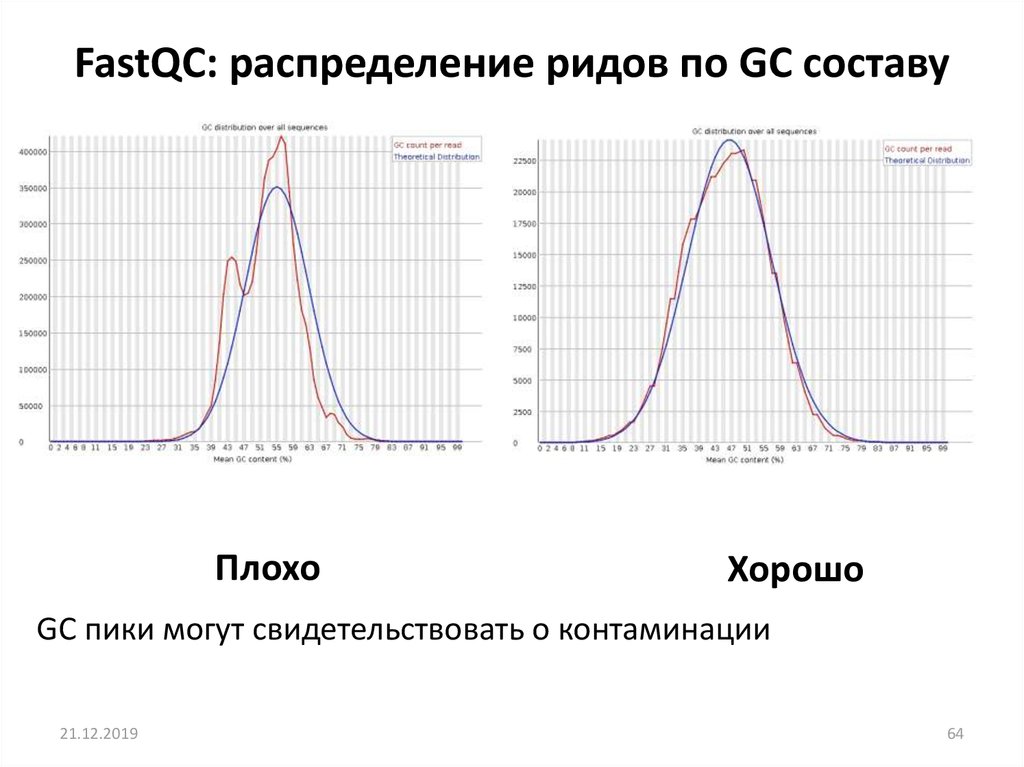
FastQC: распределение ридов по GC составуПлохо
Хорошо
GC пики могут свидетельствовать о контаминации
21.12.2019
64
64.
FastQC: уровни дупликаций последовательностейПлохо
Хорошо
Высокий уровень дупликации свидетельствует об
оверамплификации некоторых последовательностей при PCR
21.12.2019
65
65.
FastQC: Overrepresented sequencesПлохо
Перепредставленные последовательности
могут показывать источник контаминации
Хорошо
21.12.2019
66
66.
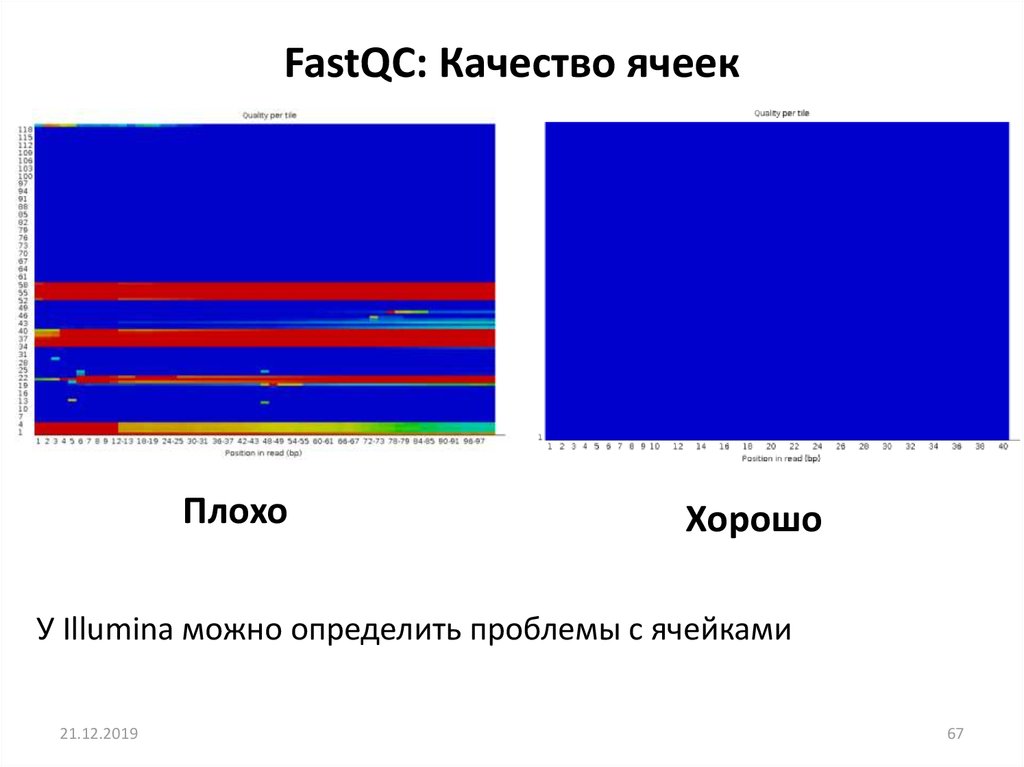
FastQC: Качество ячеекПлохо
Хорошо
У Illumina можно определить проблемы с ячейками
21.12.2019
67
67.
Шаги препроцессинга• Фильтрация данных по качеству
– Удаление ридов, качество которых ниже
определенного порога;
– Обрезание части ридов, где качество плохое
• Удаление контаминации
– Биологическая контаминация: определение и
удаление ридов
– Контаминация адапторами: вырезание
адапторов и удаление поврежденных ридов
21.12.2019
68
68. Шаги препроцессинга
У нас есть очищенные данные. Что дальше?• Сборка de novo
• Сборка по референсному геному
• Выравнивание с референсным геномом
21.12.2019
69
69. У нас есть очищенные данные. Что дальше?
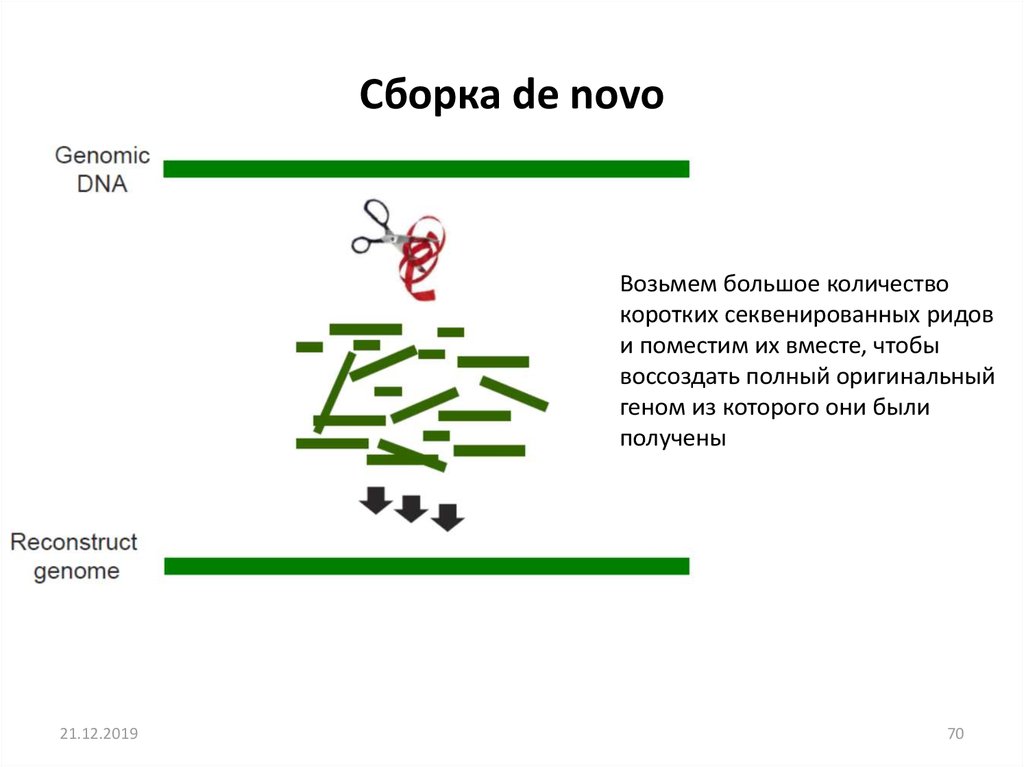
Сборка de novoВозьмем большое количество
коротких секвенированных ридов
и поместим их вместе, чтобы
воссоздать полный оригинальный
геном из которого они были
получены
21.12.2019
70
70. Сборка de novo
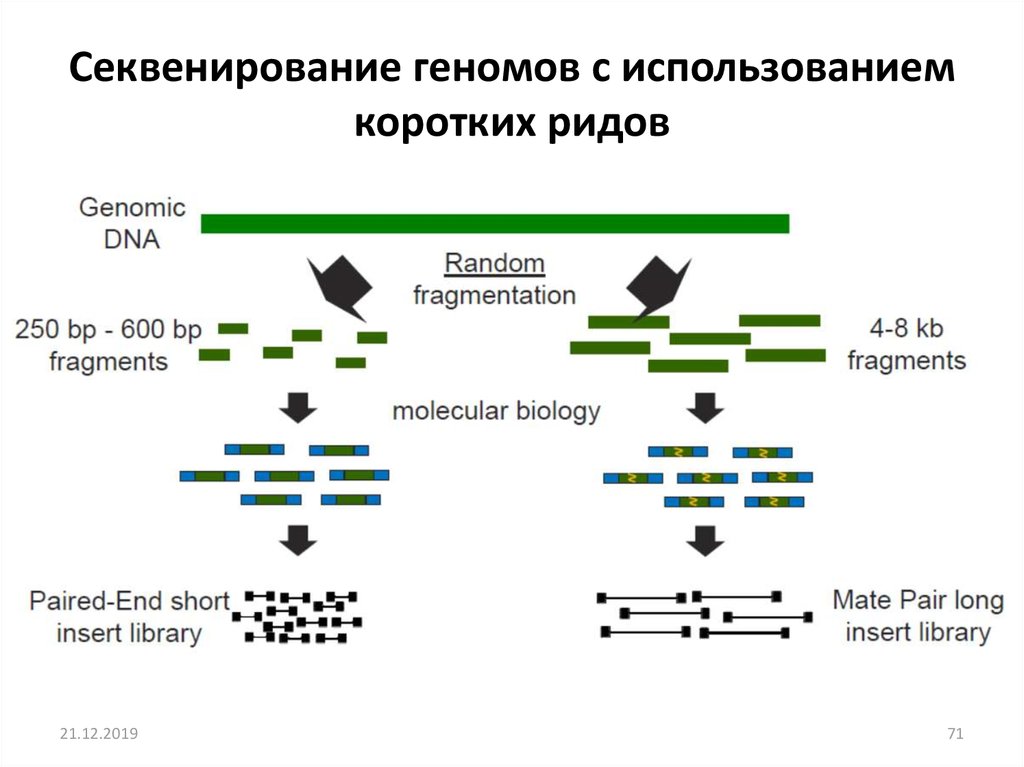
Секвенирование геномов с использованиемкоротких ридов
21.12.2019
71
71. Секвенирование геномов с использованием коротких ридов
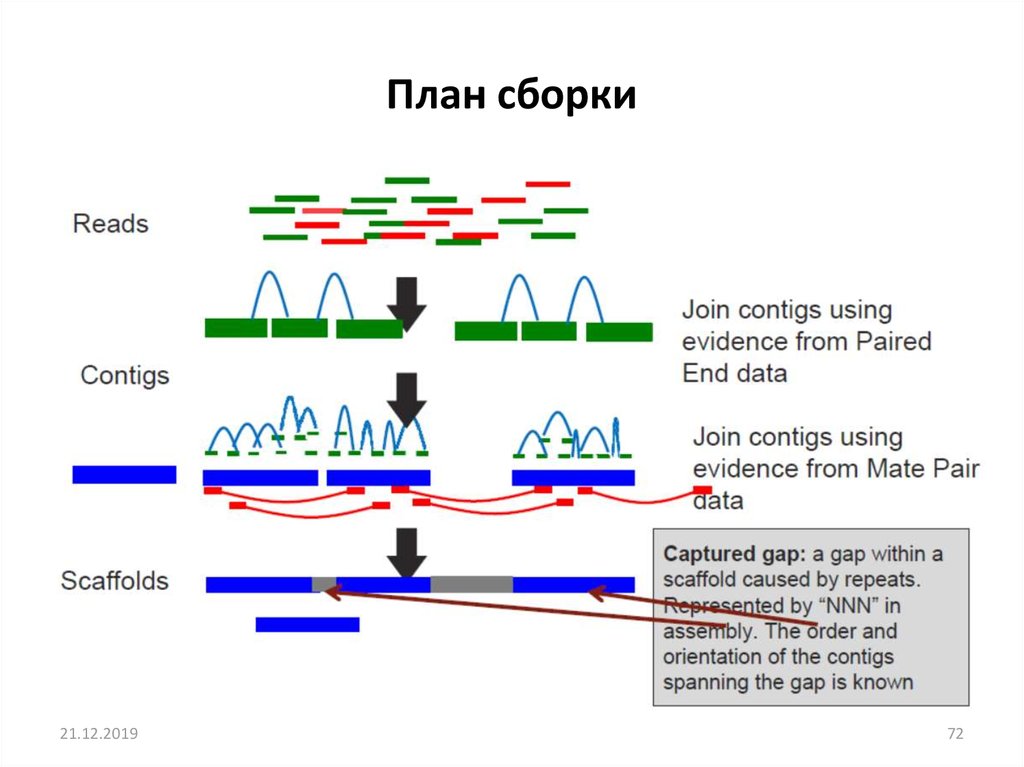
План сборки21.12.2019
72
72. План сборки
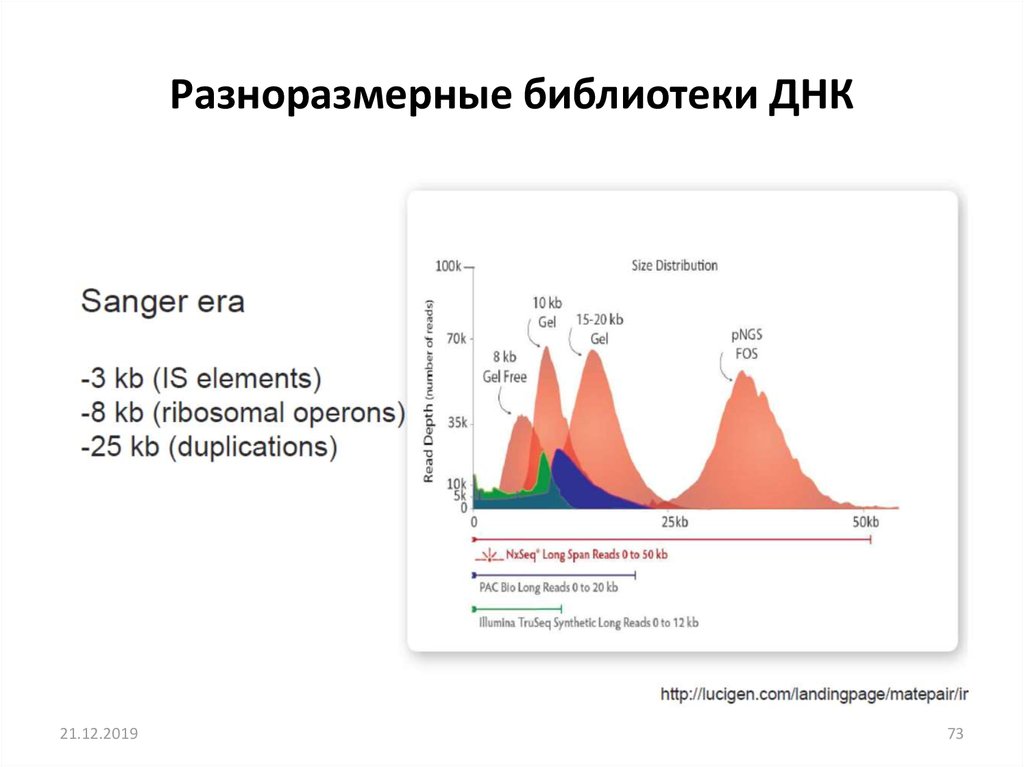
Разноразмерные библиотеки ДНК21.12.2019
73
73. Разноразмерные библиотеки ДНК
http://lucigen.com/landingpage/matepair/21.12.2019
74
74.
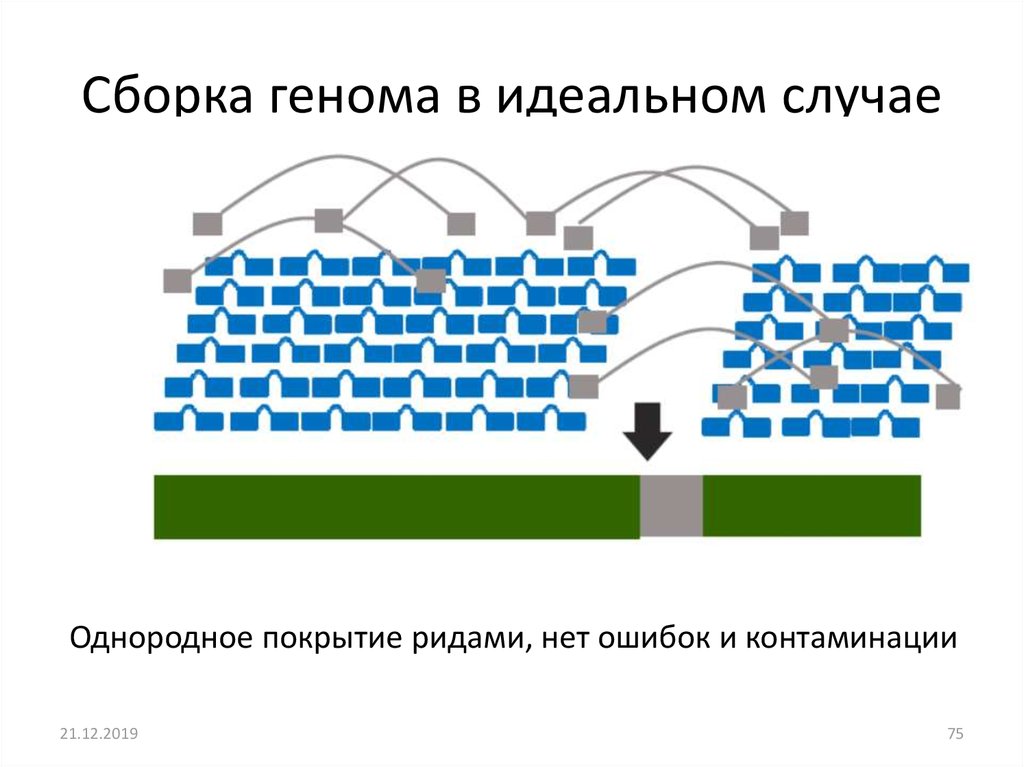
Сборка генома в идеальном случаеОднородное покрытие ридами, нет ошибок и контаминации
21.12.2019
75
75. Сборка генома в идеальном случае
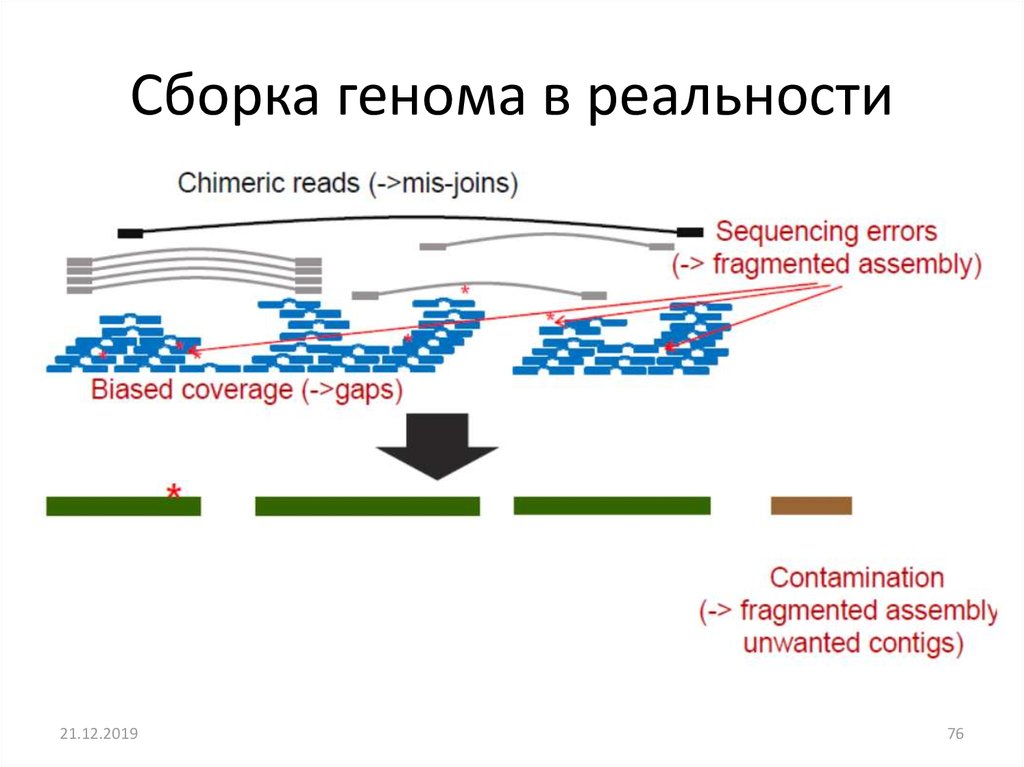
Сборка генома в реальности21.12.2019
76
76. Сборка генома в реальности
21.12.2019Кафедра биоинформатики МБФ РНИМУ
77
77.
Выбор правильной программы - сборщикагеномов (ассемблер)
• На сколько большой геном?
• Существуют ли известные особенности этого генома
(например, наличие большого числа повторов, GC состав)?
• Какое количество данных ожидается?
• Какого типа данные у вас есть?
• Какое качество данных и необходим ли их препроцессинг
перед сборкой генома?
21.12.2019
78
78. Выбор правильной программы - сборщика геномов (ассемблер)
Сборщики геномов21.12.2019
79
79. Сборщики геномов
Оценка качества сборки геномаКоличество контигов
Общая длинна всех контигов
Длинна наибольшего контига
Количество неправильно собранных
контигов
• Количество идентифицированных генов
• GC состав %
• N50
21.12.2019
80
80. Оценка качества сборки генома
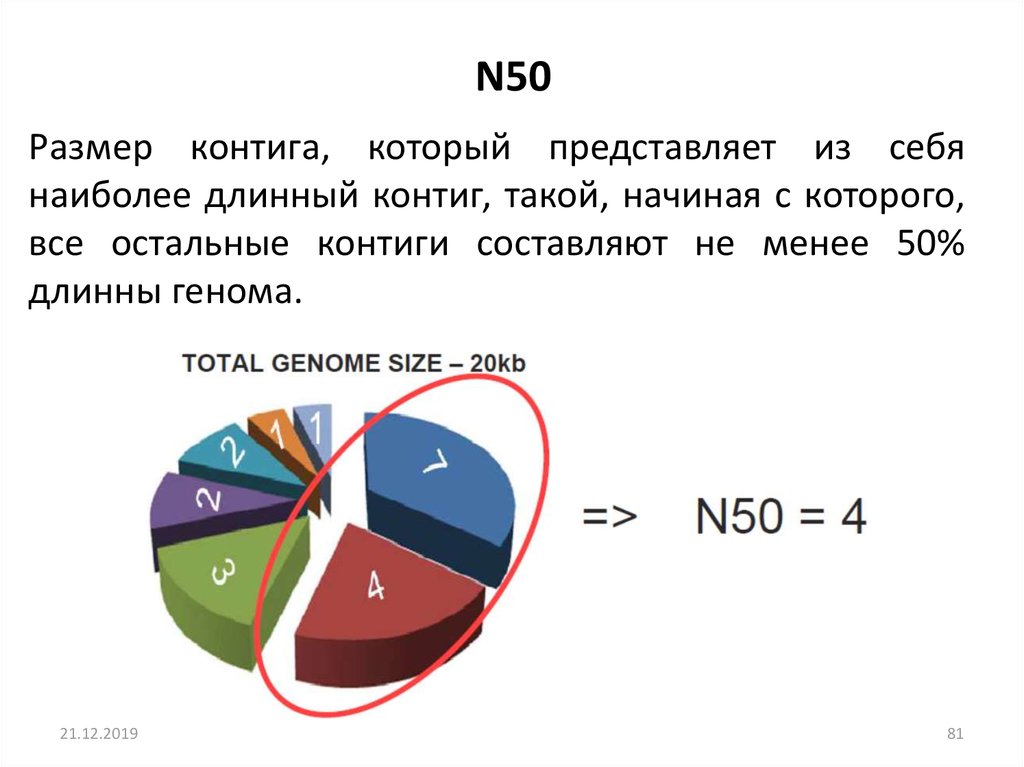
N50Размер контига, который представляет из себя
наиболее длинный контиг, такой, начиная с которого,
все остальные контиги составляют не менее 50%
длинны генома.
21.12.2019
81
81. N50
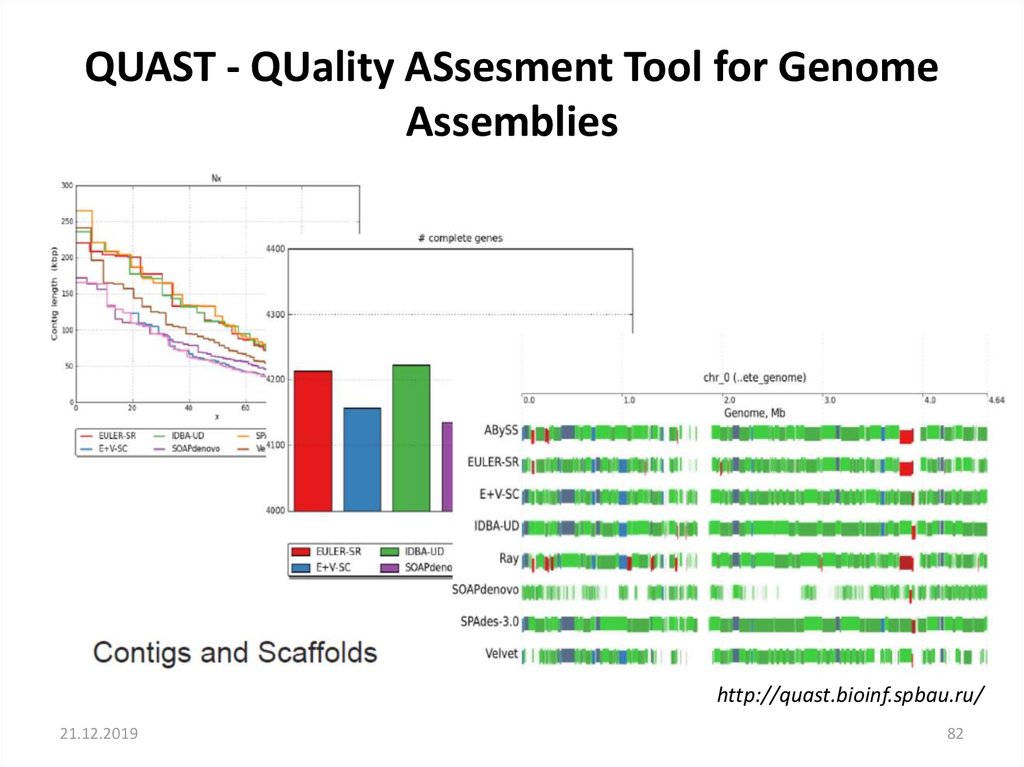
QUAST - QUality ASsesment Tool for GenomeAssemblies
http://quast.bioinf.spbau.ru/
21.12.2019
82
82. QUAST - QUality ASsesment Tool for Genome Assemblies
21.12.201983
83.
Реальные графы де Брюйна21.12.2019
84
84. Реальные графы де Брюйна
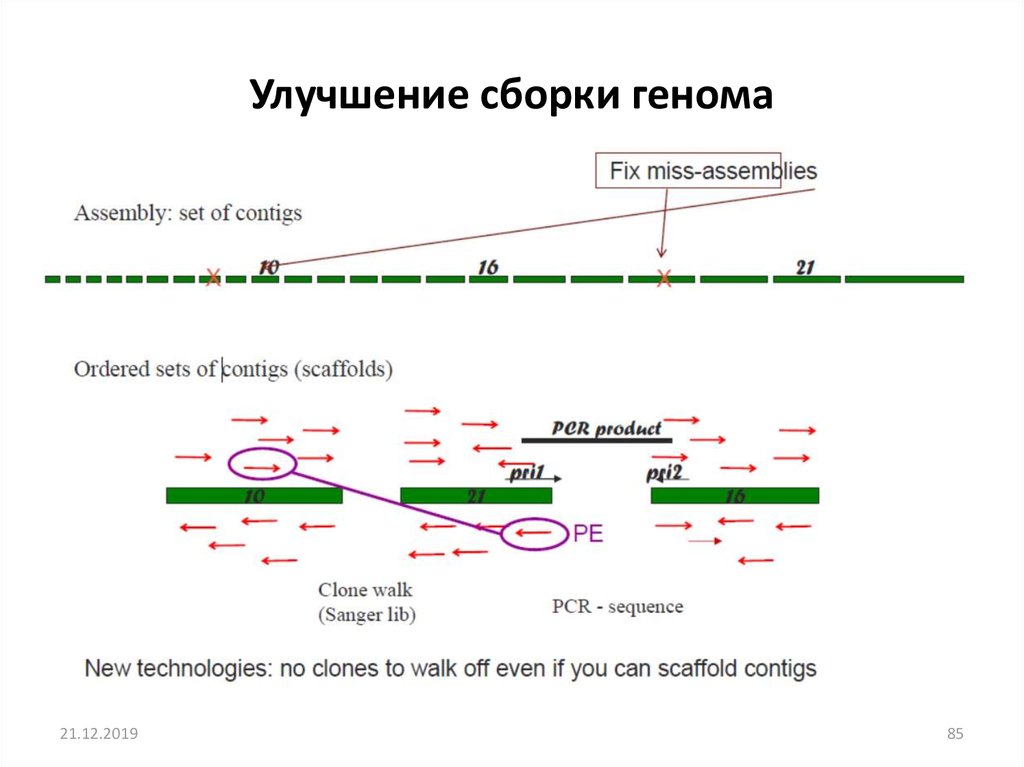
Улучшение сборки генома21.12.2019
85
85. Улучшение сборки генома
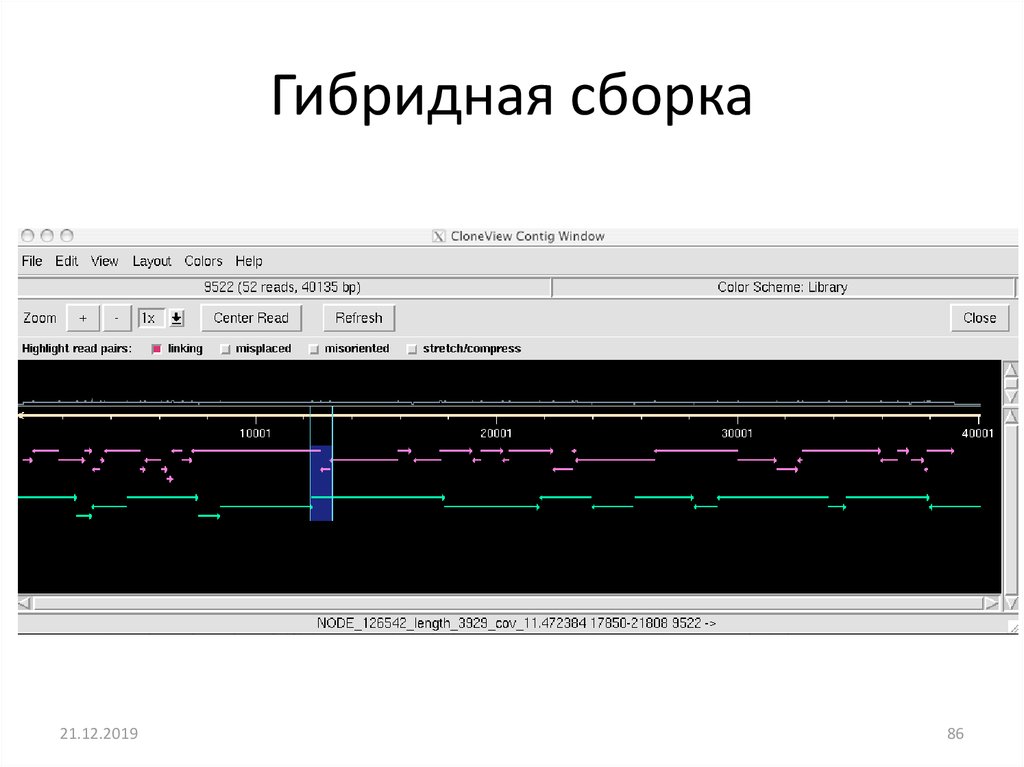
Гибридная сборка21.12.2019
86
86. Гибридная сборка
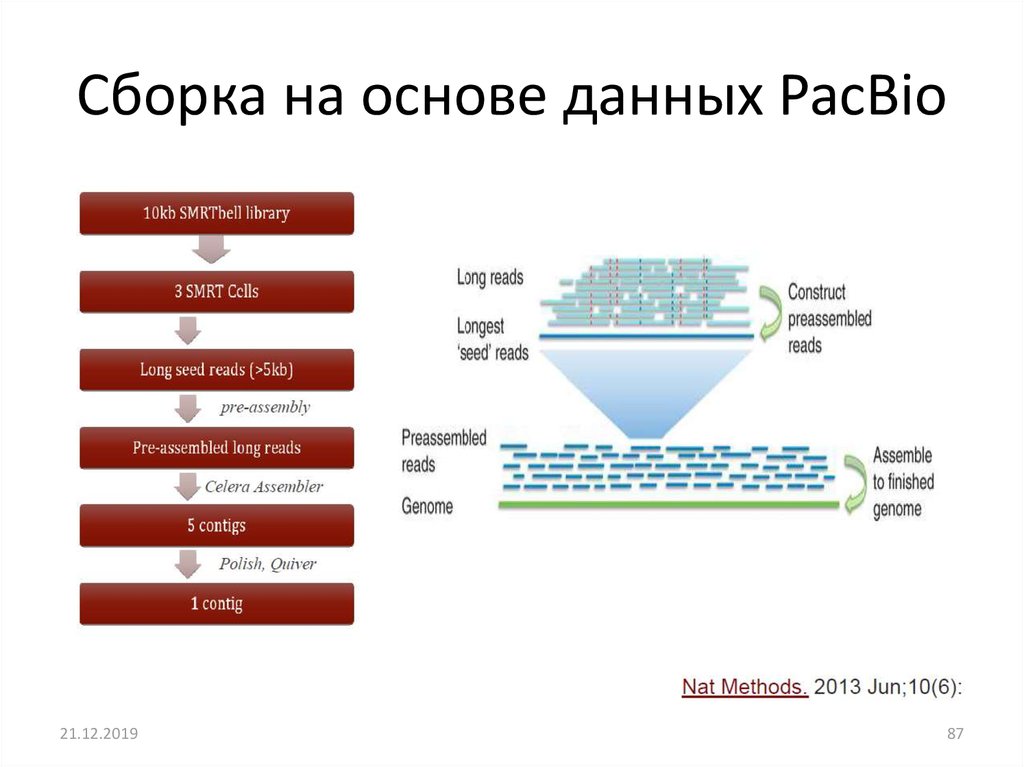
Сборка на основе данных PacBio21.12.2019
87
87. Сборка на основе данных PacBio
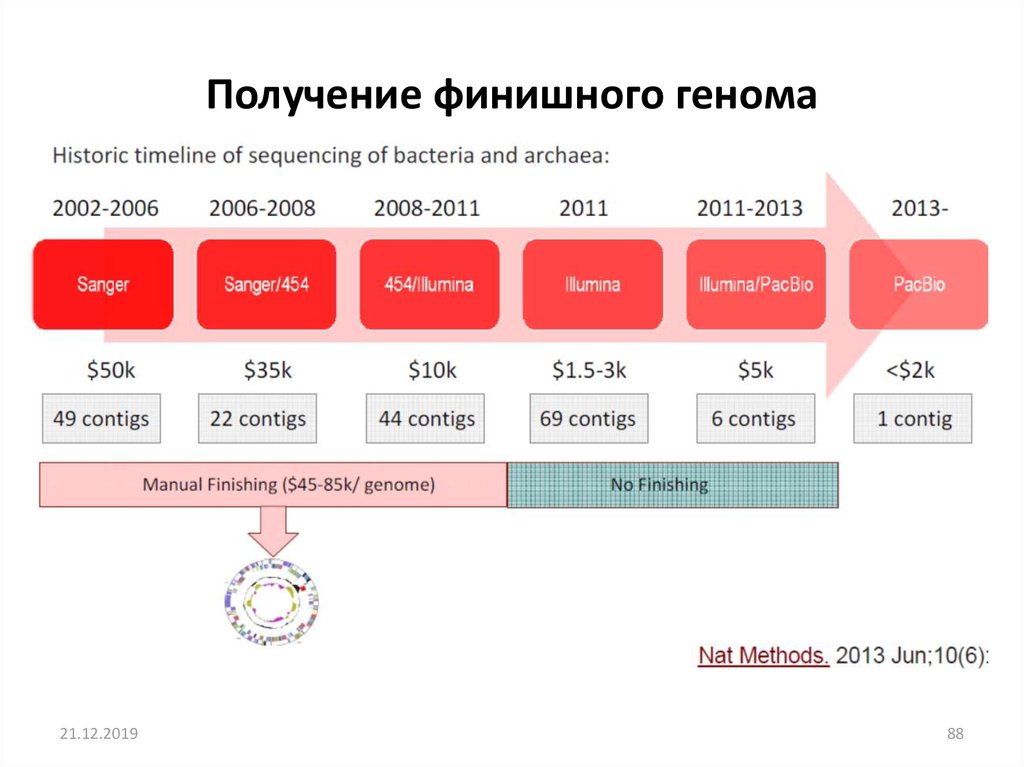
Получение финишного генома21.12.2019
88
88. Получение финишного генома
Зачем нужны финишные геномы?• Функциональные геномные исследования требуют высококачественной,
полной последовательности генома в качестве отправной точки
• Сравнительная геномика имеет смысл только в терминах полных
последовательностей генома
• Исследования бактериальных геномов требует по крайней мере одной
полной эталонной последовательности генома
• Финишные геномы помогают в идентификации источника вспышки
инфекций и филогенетическом анализе
• Полный геном - это постоянный научный ресурс
• Полный геном человека является наилучшим источником для улучшения
лечения пациентов (переход к персонализированной медицине)
21.12.2019
89
89. Зачем нужны финишные геномы?
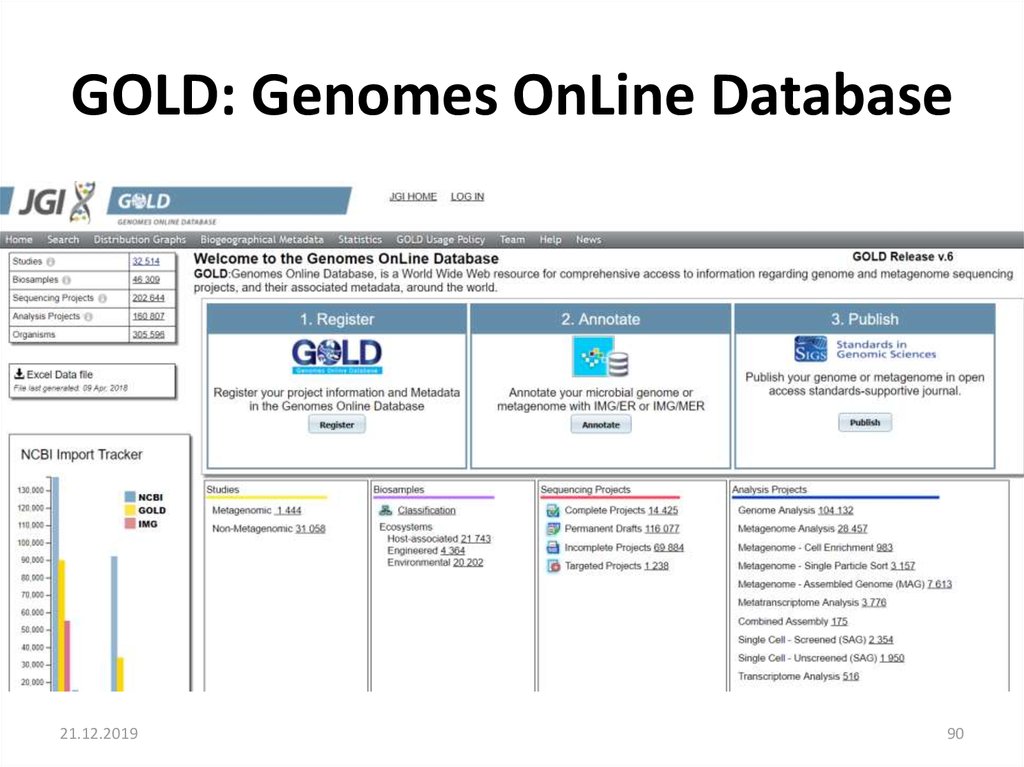
GOLD: Genomes OnLine Database21.12.2019
90
90. GOLD: Genomes OnLine Database
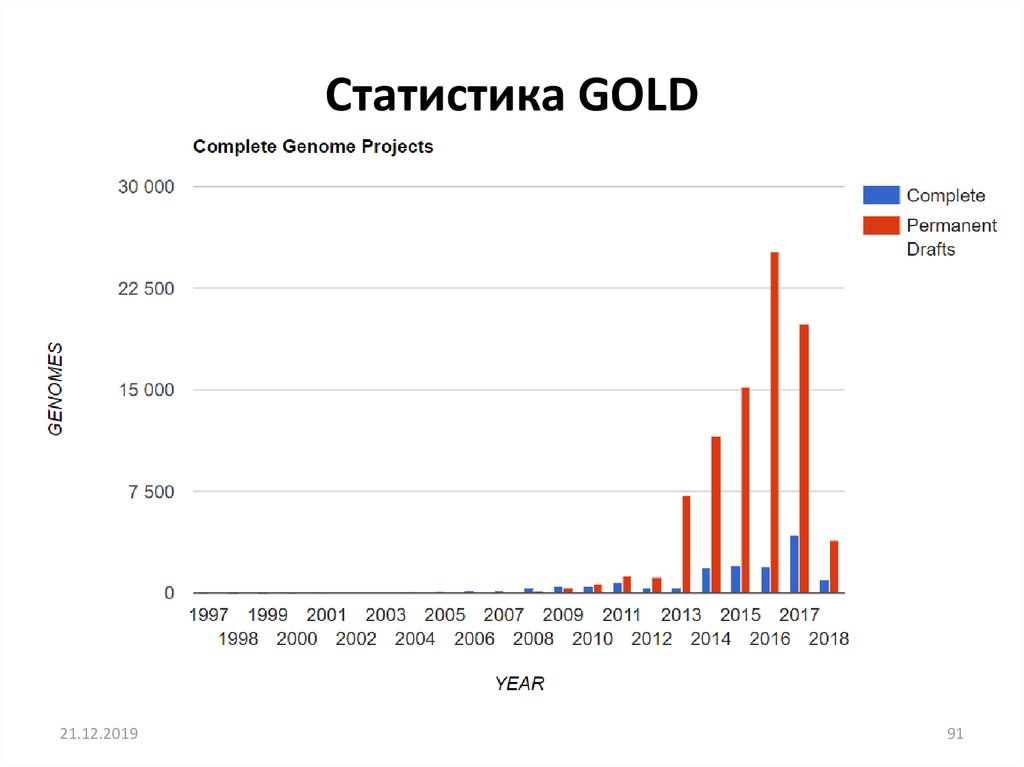
Статистика GOLD21.12.2019
91
91. Статистика GOLD
21.12.201992
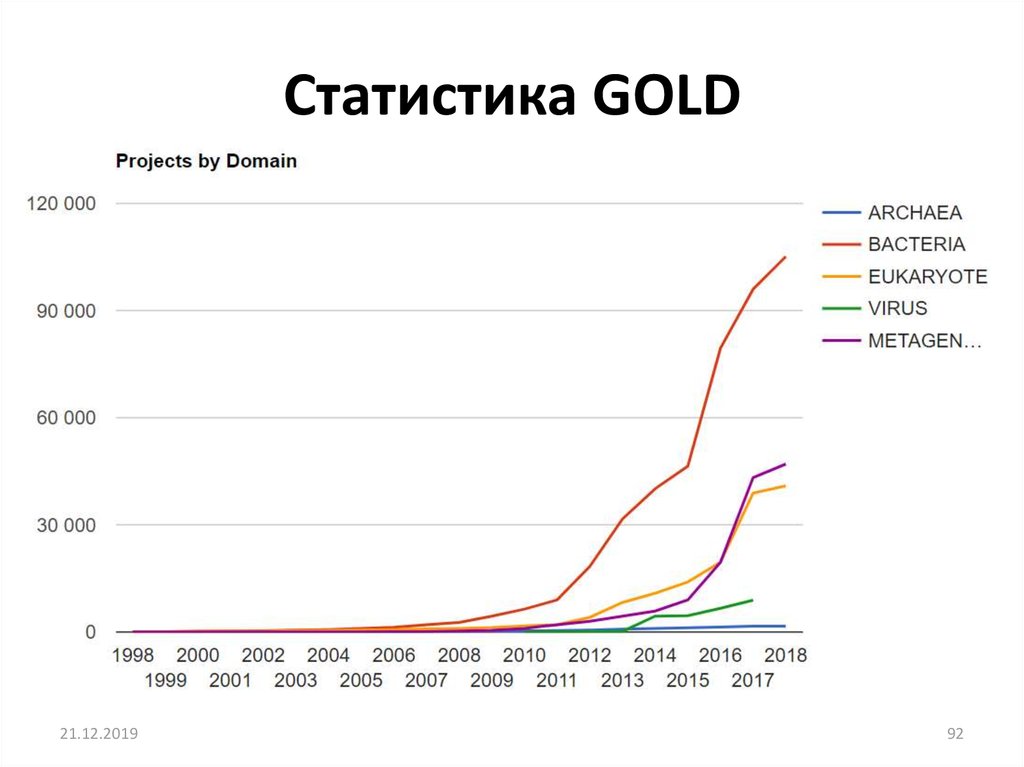
92. Статистика GOLD
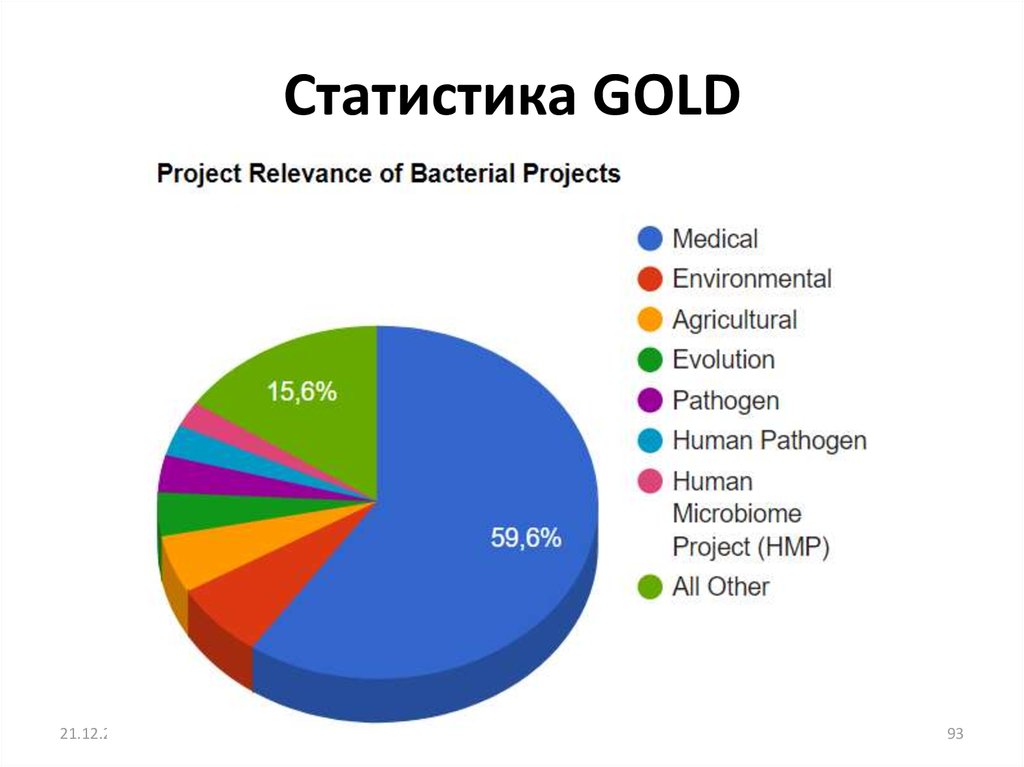
21.12.201993
93. Статистика GOLD
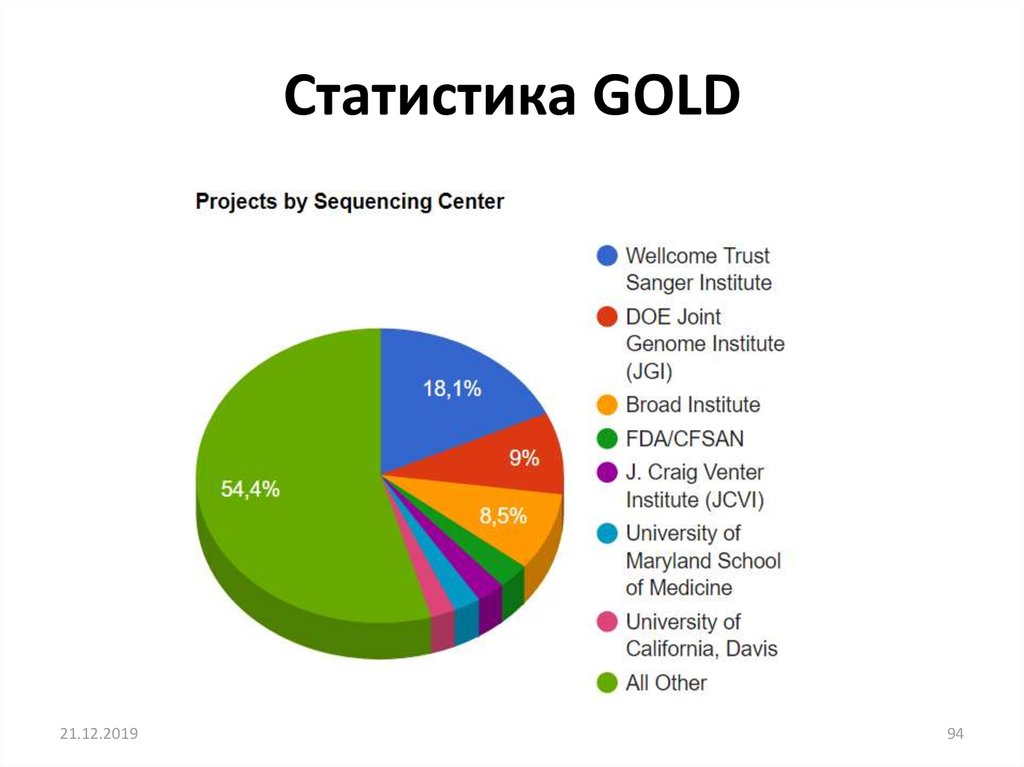
21.12.201994
94. Статистика GOLD
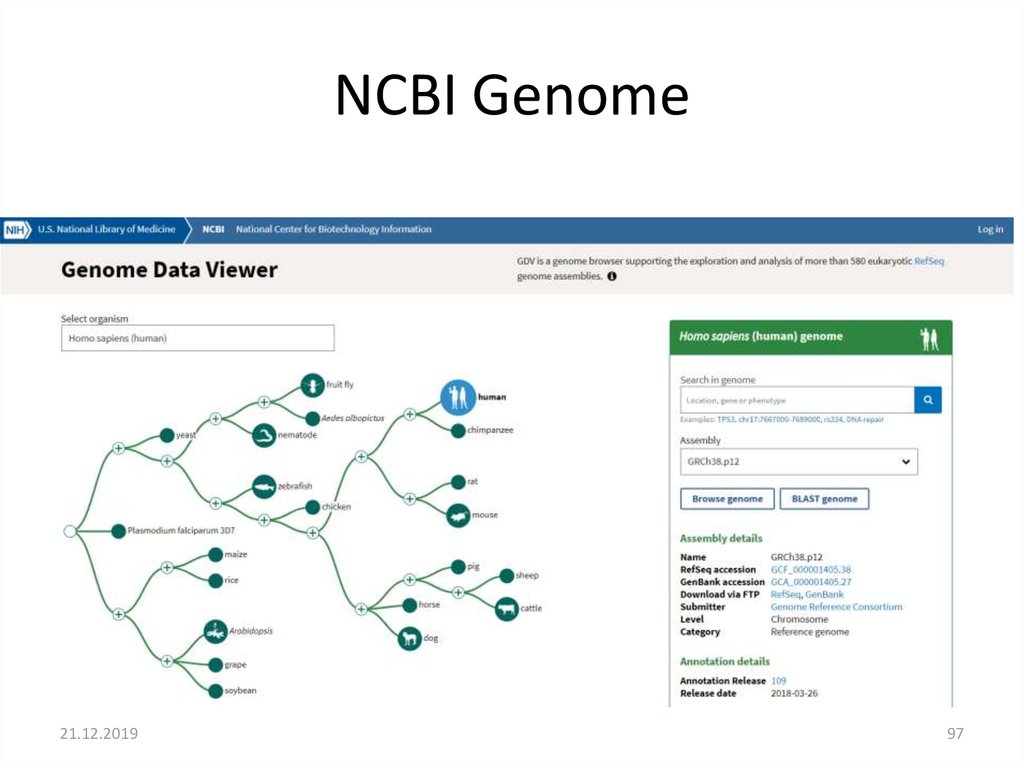
NCBI Genome21.12.2019
95
95. NCBI Genome
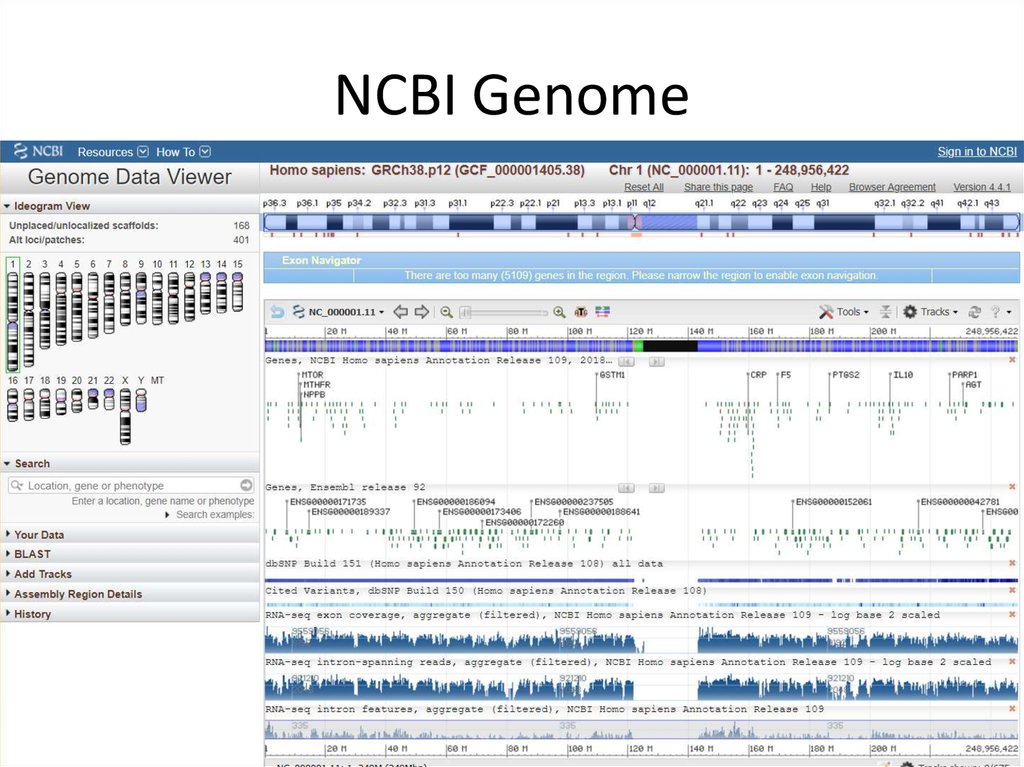
21.12.201996
96. NCBI Genome
21.12.201997
97. NCBI Genome
21.12.201998
98. NCBI Genome
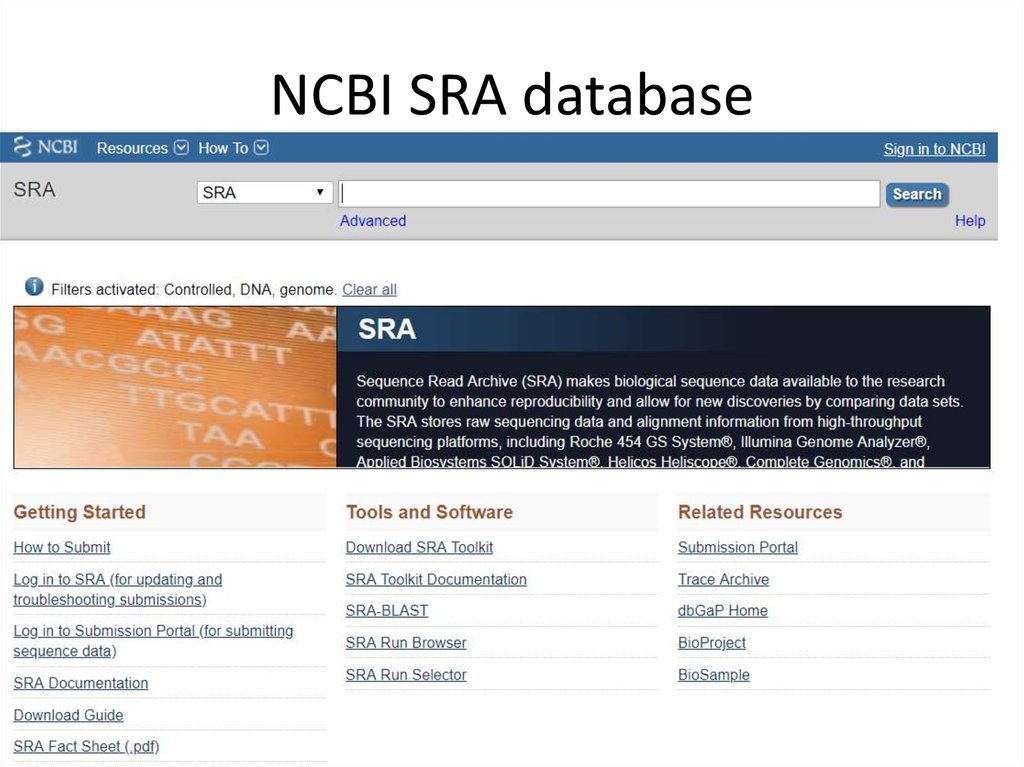
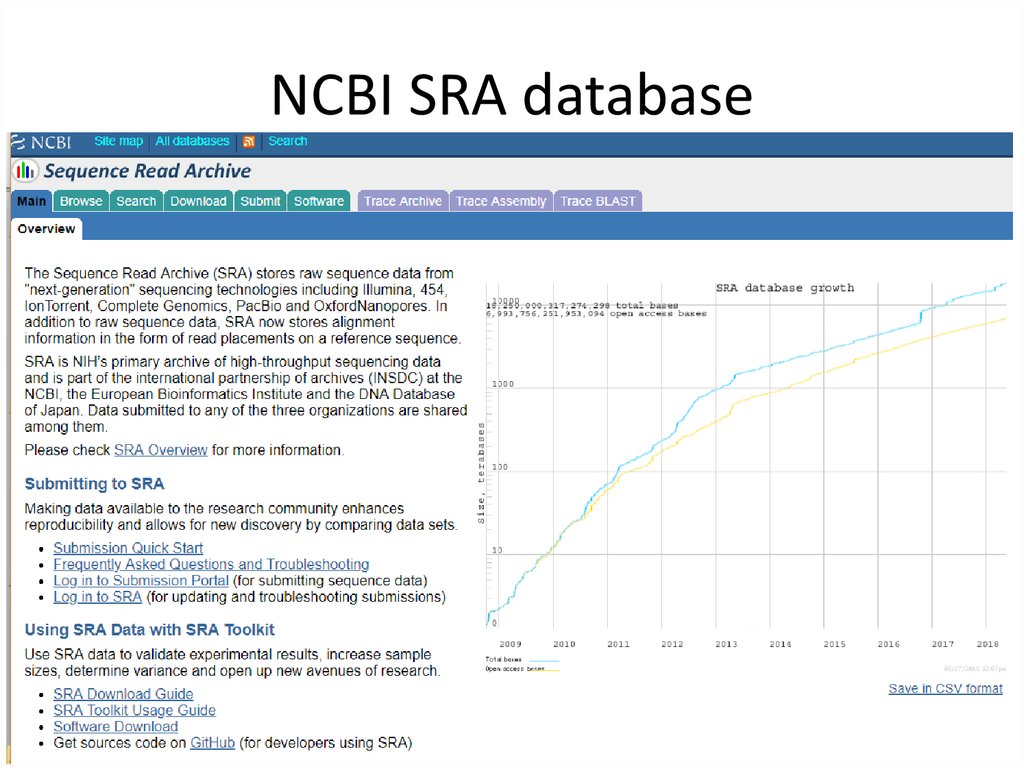
NCBI SRA database21.12.2019
99
99. NCBI SRA database
21.12.2019100















































 biology
biology informatics
informatics








