Similar presentations:
NGS приложения RNA-Seq. Этапы подготовки образца стабилизация РНК
1.
Next-generation sequencingRNA-Seq
RNA-Seq “wet”
Схема эксперимента, этапы подготовки образца:
стабилизация РНК, выделение РНК, приготовление
библиотеки, секвенирование.
«Агентство Химэксперт»
2.
Руководство к действиюПо мотивам
Principles of transcriptome analysis and
gene expression quantification: an RNAseq tutorial by Jochen B.W. Wolf
3.
«Мокрая» лабораторияСбор образцов
Контаминация
Выделение РНК и оценка качества
Истощение по рРНК
или полиА-обогащение
4.
«Мокрая» лабораторияСинтез кДНК
Создание библиотеки ДНК
Фрагментация ДНК/получение ампликонов
Лигирование адапторов
Амплификация библиотеки
Нормализация библиотеки
5.
«Мокрая» лабораторияСтратегия секвенирования
Ion PGM/Ion Proton
Пулирование образцов
Покрытие
Секвенирование
Подготовка матрицы
Секвенирование
6.
БиоинформатикаВычислительные ресурсы
Навыки программирования
Форматы файлов
Контроль качества
7.
БиоинформатикаРабота с транскриптомом
Variant calling
Количественная оценка экспрессии
Картирование
Определение генов
8.
СтатистикаНормализация
Экспрессия генов
Альтернативный сплайсинг
Функции генов и их взаимодействие
9.
Применение NGS для исследования РНК1. Уровень экспрессии генов
1. Транскриптом
2. Таргетные исследования
2. Альтернативный сплайсинг
3. Малые РНК (miRNA, siRNA, snoRNA, snRNA, tRNA, piRNA)
4. Антисмысловые и некодирующие РНК
5. Транскриптом одной клетки
6. Транслируемые РНК (РНК-белковые взаимодействия)
7. Двуцепочечные РНК и вторичные структуры на РНК
10.
RNA-seqНекорректное название изучения РНК с помощью
высокопроизводительного секвенирования, т.к.
секвенируется не РНК, а кДНК
NGS высокопроизводительное секвенирование. П/ред Д.Ребрикова. М.: Бином, 2014.
11.
RNA-seqПринцип RNA-Seq заключается в глубоком
секвенировании кДНК с добавленными адаптерами
на обоих концах кДНК
Wang Z., Gerstein M., Snyder M. RNA-Seq: a revolutionary tool for transcriptomics // Nat Rev Genet. - 2009. - Vol. 10, No. 1. - P. 57-63.
Sánchez-Pla A., Reverter F., Ruíz de Villa M.C., Comabella M. Transcriptomics: mRNA and alternative splicing// J Neuroimmunol. - 2012. - Vol. 248, No.
1-2. - P. 23-31.
12.
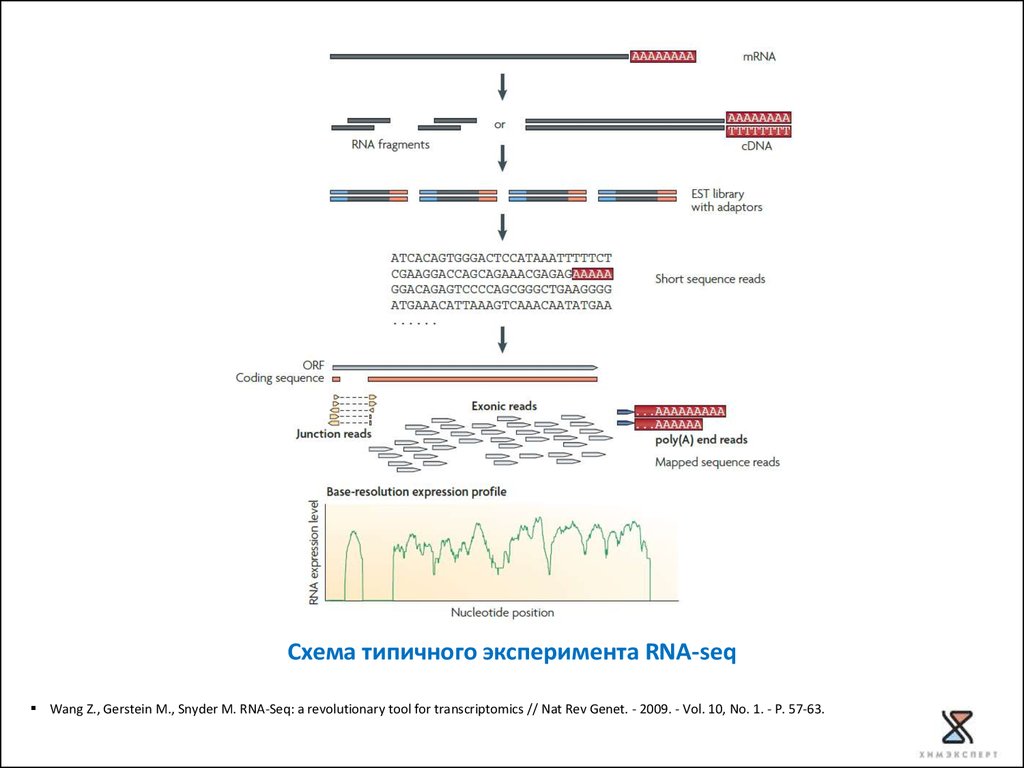
Схема типичного эксперимента RNA-seqWang Z., Gerstein M., Snyder M. RNA-Seq: a revolutionary tool for transcriptomics // Nat Rev Genet. - 2009. - Vol. 10, No. 1. - P. 57-63.
13.
NGShttp://gcat.davidson.edu/phast/
14.
RNA-Seq для анализа РНКШаг 1: Стабилизация РНК образце
Шаг 2: Выделение РНК из образца
Шаг 3: Приготовление библиотеки
Шаг 4: Секвенирование
Шаг 5: Анализ данных
Life Technologies—Sample to RNA-Seq. 2012
15.
Шаг 1: Стабилизация РНК16.
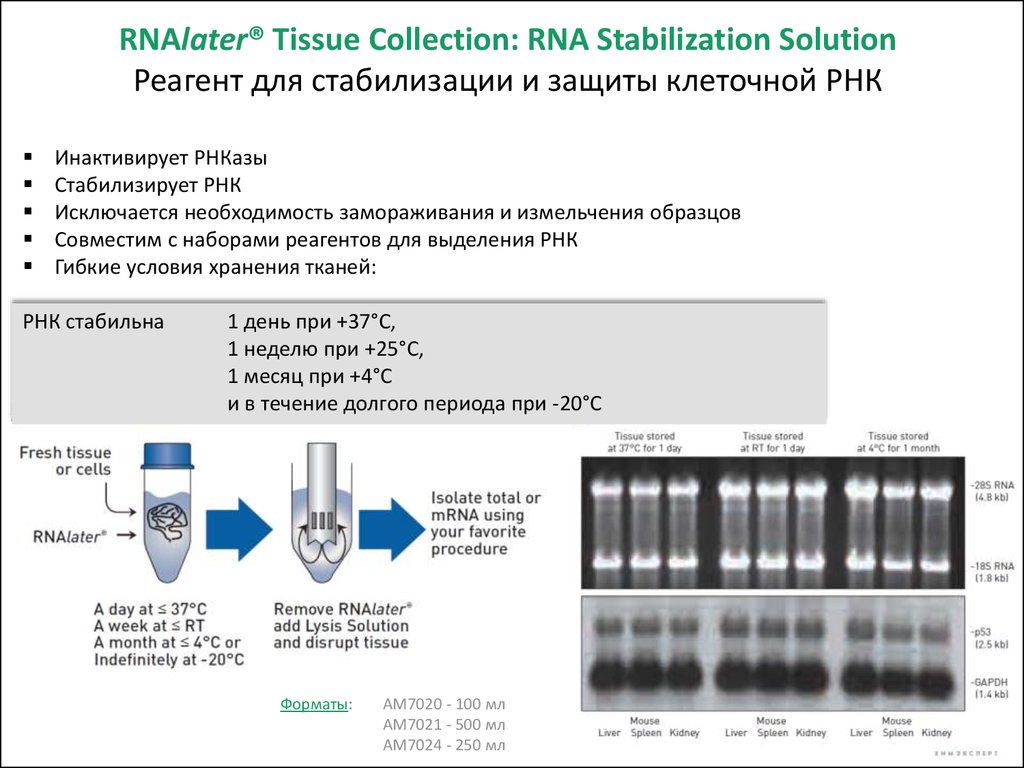
RNAlater® Tissue Collection: RNA Stabilization SolutionРеагент для стабилизации и защиты клеточной РНК
Инактивирует РНКазы
Стабилизирует РНК
Исключается необходимость замораживания и измельчения образцов
Совместим с наборами реагентов для выделения РНК
Гибкие условия хранения тканей:
РНК стабильна
1 день при +37°C,
1 неделю при +25°C,
1 месяц при +4°C
и в течение долгого периода при -20°C
Форматы:
AM7020 - 100 мл
AM7021 - 500 мл
AM7024 - 250 мл
17.
Шаг 2: Выделение РНК18.
Основные наборы для выделения РНКНаборы для выделения РНК из широкого ряда образцов
PureLink® RNA Mini Kit
mirVana™ miRNA
Isolation Kit
Dynabeads® mRNA
DIRECT™ Micro Kit
20 мин
30 мин
15 мин
вся РНК
- включая рРНК
10-100 мг ткани
До 5 x 107 клеток*
микроРНК
- малые и большие молекулы РНК
- включая рРНК
До 100 мг ткани
До 5 x 107 клеток
*Количество исходного материала
Кат.ном.:
12183018A
AM1560
610-21
только мРНК
- PureLink® RNA Mini Kit
- mirVana™ miRNA Isolation Kit
- Dynabeads® mRNA DIRECT™ Micro Kit
До 5 мг ткани
До 1 × 104 клеток
До 500 нг очищенной
тотальной РНК
19.
Набор для выделения мРНКDynabeads® mRNA DIRECT™ Micro Kit
Dynabeads® Oligo (dT)25 – суперпарамагнитные
частицы с олиго(dT)-последовательностью,
ковалентно прикрепленной на поверхности
Суперпарамагнетизм — форма магнетизма, проявляющаяся у
ферромагнитных и ферримагнитных частиц.
В обычном состоянии не проявляют магнитные свойства, но при
воздействии магнитного поля они становятся намагниченными
Метод валидирован для создания
транскриптомных библиотек для RNA-Seq
при помощи Ion Total RNA-Seq Kit v2
20.
Специализированные наборы для выделения РНКНаборы для выделения РНК оптимизированные
Парафинизированные образцы
MagMAX™ FFPE Total Nucleic Acid Isolation Kit
RecoverAll™ Total Nucleic Acid Isolation Kit
for FFPE
Кровь
MagMAX™ for Stabilized Blood Tubes RNA Isolation Kit
Вирусы
PureLink® Viral RNA/DNA Mini Kit
21.
Прокариоты:80% рРНК
15% тРНК
4-5% мРНК и некодРНК
Эукариоты:
Человек 1-5% мРНК
22.
Обогащение РНКНабор RiboMinus™ Eukaryote Kit v2 для удаления рРНК
Мишени:
28S, 18S, 5.8S, 5S субъединицы цитоплазматической рРНК
12S и 16S субъединицы митохондриальной рРНК
• 1 час
• магнитные частицы
• 1-5 мкг или 100 нг – 1 мкг
Данные NGS для универсальной
референсной РНК человека до и
после удаления рРНК
23.
Обогащение РНКНабор RiboMinus™ Eukaryote Kit v2 для удаления рРНК
мРНК
рРНК
магнитная
частица со
стрептавидином
биотинилированный
олигонуклеотид-приманка
Взаимодействие биотинилированного зонда с рРНК
Удаление рибосомальной РНК за счет взаимодействия биотин-стрептавидин
24.
Зонды RiboMinus™ для гибридизации с рРНКRiboMinus™ для гибридизации с высококонсервативными участками
5S, 5.8S, 18S и 28S рРНК,
состоит из смеси олигонуклеотидов (содержащих по 3 LNA™ мономера*),
включающей по 2 специфичных зонда к каждому классу рРНК.
*LNA (locked nucleic acid) - «закрытая» нуклеиновая кислота
25.
26.
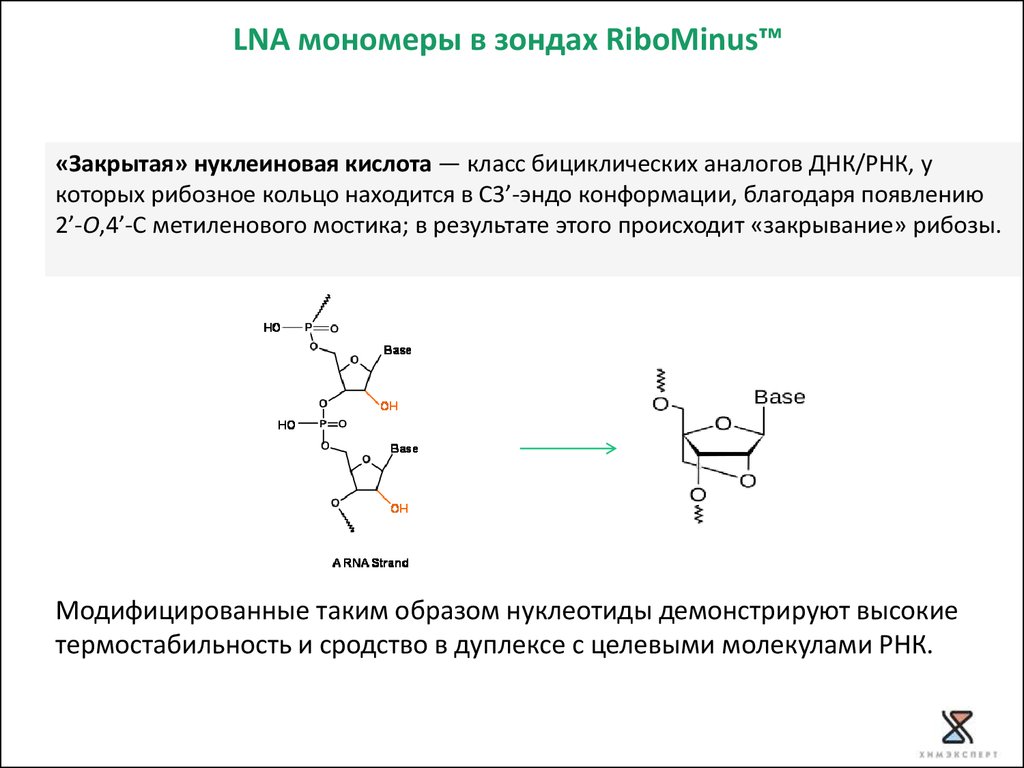
LNA мономеры в зондах RiboMinus™«Закрытая» нуклеиновая кислота — класс бициклических аналогов ДНК/РНК, у
которых рибозное кольцо находится в C3’-эндо конформации, благодаря появлению
2’-O,4’-C метиленового мостика; в результате этого происходит «закрывание» рибозы.
Модифицированные таким образом нуклеотиды демонстрируют высокие
термостабильность и сродство в дуплексе с целевыми молекулами РНК.
27.
Рабочий процессRiboMinus™ Eukaryote System v2 Workflow
5 мин 37⁰C – гибридизация с
частицами со стрептавидином
2.
1.
10 мин 70⁰C – денатурация
20 мин 37⁰C – гибридизация с
биотинилированными зондами
Отбор супернатанта из образца в
магнитном поле (без рРНК)
3.
Кат.ном.:
A15020
A15026
- RiboMinus™ Eukaryote Kit v2
- RiboMinus™ Eukaryote System v2
4.
30 мин – концентрирование РНК на
магнитных частицах Nucleic Acid Binding Beads
28.
КонтрольThe External RNA Controls Consortium (ERCC)
Консорциум по контрольной внешней РНК (ERCC) - спецгруппа из академических,
частных и общественных организаций.
ERCC - набор контрольной РНК для оценки качества экспрессии генов на
различных платформах:
количественная ОТ-ПЦР,
микрочипы,
NGS технологии.
29.
КонтрольERCC RNA Spike-In Control Mixes
92 варианта полиаденилированных транскриптов
Размер транскриптов 250-2000 нукл.
«мимикрирует» под естественную
эукариотическую мРНК
Используется для оценки точности измерений
дифференциальной экспрессии генов
Состав:
• 10 мкл ExFold Spike-In Mix 1
• 10 мкл ExFold Spike-In Mix 2
• 1.75мл вода без нуклеаз
Transcript molar ratios in ERCC RNA Spike-In Control Mixes. The transcripts
in Spike-In mix 1 and Spike-In mix 2 are present at defined mix 1:mix 2
molar concentration ratios, described by 4 subgroups. Each subgroup
contains 23 transcripts spanning a 106-fold concentration range, with
approximately the same transcript size distribution and GC content. The
controls are ideal for next-generation sequencing experiments.
Кат.ном.:
4456739
- ERCC RNA Spike-In Control Mixes
30.
Шаг 3: Приготовлениебиблиотеки
31.
Набор Ion Total RNA-Seq Kit V2.0Единый набор для получения
ориентированной библиотеки
кДНК для любых видов РНК
Библиотека за 6 часов
Может использоваться для анализа как
коротких РНК, так и полного набора
транскриптов в клетке (транскриптома).
Рекомендации:
1–500 нг поли(А) РНК,
10–500 нг тотальной РНК без рРНК
Тотальная РНК высокого качества (RIN >7)
Ion Total RNA-Seq Kit v2 Publication Number 4476286 Revision E © 2013 Life Technologies Corporation
32.
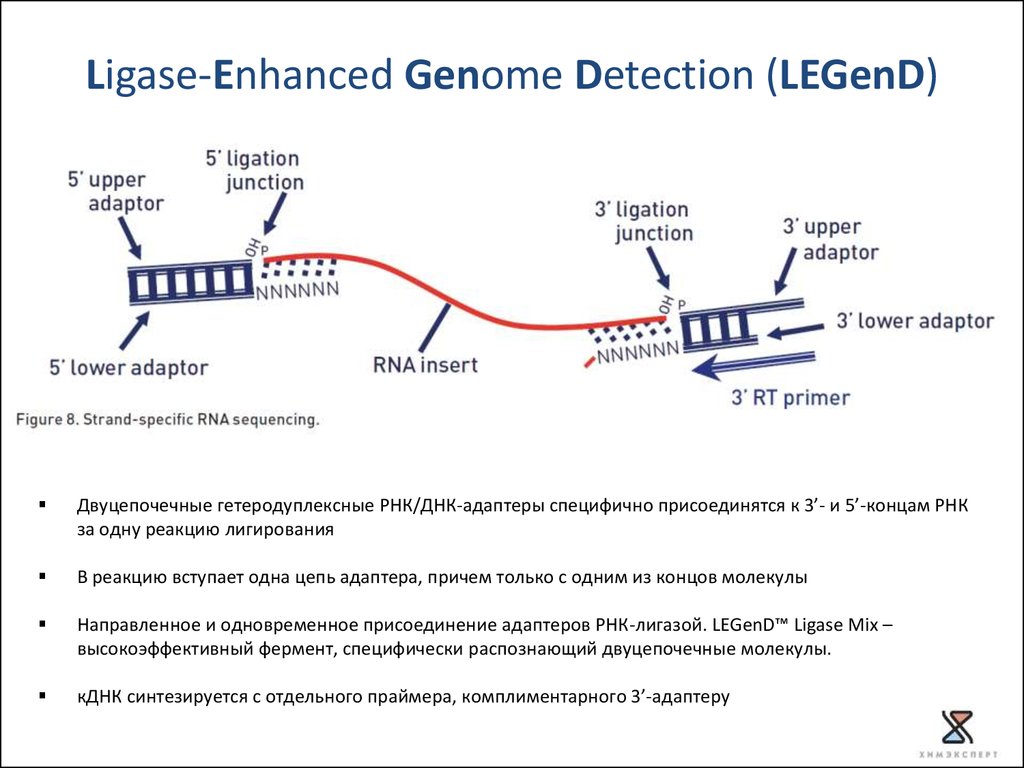
Ligase-Enhanced Genome Detection (LEGenD)Двуцепочечные гетеродуплексные РНК/ДНК-адаптеры специфично присоединятся к 3’- и 5’-концам РНК
за одну реакцию лигирования
В реакцию вступает одна цепь адаптера, причем только с одним из концов молекулы
Направленное и одновременное присоединение адаптеров РНК-лигазой. LEGenD™ Ligase Mix –
высокоэффективный фермент, специфически распознающий двуцепочечные молекулы.
кДНК синтезируется с отдельного праймера, комплиментарного 3’-адаптеру
33.
Единый набор для получения ориентированнойбиблиотеки кДНК для любых видов РНК
5’ RNA
adaptor
5’ ligation
junction
3’ ligation
junction
3’ DNA
adaptor
P
P
5’ ‘guide’
adaptor
RNA fragment or
Short RNA 3’-amine
Strand Oriented cDNA
siRNA
12,14,16 nts
3’ ‘guide’ adaptor
(3’ blocked)
3’ RT primer
34.
Oligonucleotides and methodsfor the preparation of RNA libraries
Oligonucleotides and methods for the preparation of rna libraries US 20140128291 A1
35.
Ion Total RNA-Seq Kit v2: Рабочий процессPreparation of whole transcriptome RNA
Preparation of small RNA
25-500 ng rRNA-depleted total RNA or 1-500 ng poly(A) RNA or
100 ng total RNA
Магнитные
частицы
Obtain total RNA then determine the quality
Enrich small RNA
Fragment the RNA
Clean up the RNA
Магнитные
частицы
Quantitate small RNA sample and determine input amount
SOLiDPGM
™ amplified
™ Создание
library
библиотеки
construction
Гибридизация и лигирование адаптеров с РНК
Обратная транскрипция
< 5 часов
Магнитные
частицы
Магнитные
частицы
Очистка кДНК
Амплификация кДНК
(добавление баркодов)
Магнитные
частицы
Магнитные
частицы
Очистка амплифицированной ДНК
Оценка выхода и размеров амплифицированной ДНК
IonSphere™ particle preparation and Ion PGM™sequencing
< 6 часов
36.
Секвенирование малых РНКМалые РНК – короткие некодирующие молекулы РНК
tRNA - транспортировка аминокислот к месту синтеза белка.
snRNA - класс РНК, которые встречаются в ядре эукариотических клеток. Они транскрибируются РНКполимеразой II или РНК-полимеразой III и участвуют в важных процессах, таких как сплайсинг (удаление
интронов из незрелой мРНК), регуляции факторов транскрипции (7SK РНК) или РНК-полимеразы (B2
РНК) и поддержании целостности теломер.
от 73 до 93
нуклеотидов
snoRNA - малые ядрышковые РНК считаются подгруппой малых ядерных РНК. Участвуют в
химических модификациях (метилировании и псевдоуридилировании) рибосомных РНК, а также тРНК и
малых ядерных РНК.
miRNA - малые некодирующие молекулы РНК длиной обнаруженные у растений, животных и
некоторых вирусов, принимающие участие в транскрипционной и посттранскипционной регуляции
экспрессии генов путем РНК-интерференции.
siRNA - малые интерферирующие РНК или короткие интерферирующие РНК, класс двухцепочечных
РНК, Принимают участие в процессах РНК-интерференции, понижая экспрессию специфических генов.
piRNA - наиболее крупный класс малых некодирующих РНК, экспрессируемых в клетках животных;
они обнаружены в комплексах с белками семейства Piwi, за что и получили своё название. piРНК
обычно длиннее микроРНК и малых интерферирующих РНК и имеют , кроме того, в отличие от
микроРНК, они не так консервативны.
18-25 нуклеотидов
(в среднем 22)
20-25 нуклеотидов
26—32 нуклеотида
37.
Создание библиотеки малых РНКГибридизация и лигирование адаптеров с РНК
• Ion Adaptor Mix v2
• Hybridization Solution
• 2X Ligation Buffer
• Ligation Enzyme Mix
• 3 µL of small RNA sample
(1–100 ng of miRNA in ≤1 µg of enriched small RNA)
Обратная транскрипция
• Nuclease-Free Water
• 10X RT Buffer
• 2.5 mM dNTP Mix
• Ion RT Primer v2
• 10X SuperScript® III Enzyme Mix
38.
Фермент для обратной транскрипцииSuperScript III
(50°С оптимум температуры)
Чем выше термостабильность ревертазы,
тем выше эффективность обратной транскрипции
(связано с денатурацией вторичных структур РНК при
повышении температуры)
+ сниженная RNase H активность
(SuperScript® III RT was engineered to contain mutations that
knock out the RNase H activity)
39.
Создание библиотеки малых РНКОчистка и size-select кДНК на магнитных частицах
1) Первое связывание: на частицах задерживаются большие молекулы кДНК (tRNA и rRNA).
2) Второе связывание: целевые продукты кДНК (miRNA и другие маляе РНК) при повышении
концентрации EtOH из надосадочной жидкости повторно связываются с частицами.
3) Элюция целевых продуктов кДНК водой Nuclease-Free (37°C).
• Wash Solution Concentrate
• Binding Solution Concentrate
• Nucleic Acid Binding Beads
• Processing Plate
• Nuclease-Free Water
Кат.ном.:
AM10027
AM10050
- Magnetic stand for 96-well plates (Ambion®)
- Magnetic stand for 96-well plates (Ambion®)
40.
Создание библиотеки малых РНКАмплификация кДНК
Компоненты Ion Total RNA-Seq Kit v2 для библиотек без баркода:
• Ion 5′ PCR Primer v2
• Ion 3′ PCR Primer v2
• Platinum® PCR SuperMix High Fidelity
Компоненты Ion Xpress™ RNA-Seq Barcode 1–16 Kit для баркодированных библиотек:
• Ion Xpress™ RNA-Seq Barcode BC 01 – BC 16
• Ion Xpress™ RNA 3′ Barcode Primer
Platinum® PCR SuperMix High Fidelity – высокоточная полимераза
Кат.ном.:
4475485
- Ion Xpress™ RNA-Seq Barcode 1-16 Kit
41.
Создание библиотеки малых РНКОчистка и size-select амплифицированной ДНК на магнитных
частицах
1) Первое связывание: на частицах задерживаются большие молекулы кДНК (tRNA и rRNA).
2) Второе связывание: целевые продукты кДНК (miRNA и другие малые РНК) при повышении
концентрации EtOH из надосадочной жидкости повторно связываются с частицами.
3) Элюция целевых продуктов кДНК водой Nuclease-Free (37°C).
• Wash Solution Concentrate
• Binding Solution Concentrate
• Nucleic Acid Binding Beads
• Processing Plate
• Nuclease-Free Water
Кат.ном.:
AM10027
AM10050
- Magnetic stand for 96-well plates (Ambion®)
- Magnetic stand for 96-well plates (Ambion®)
42.
Создание библиотеки малых РНКОценка выхода и размеров амплифицированной ДНК
a. Задать размерные границы
продуктов ПЦР:
50–300 bp (все продукты после лигирования)
86–106 bp (для небаркод. библиотек) или
94–114 bp (для баркодированных библиотек)
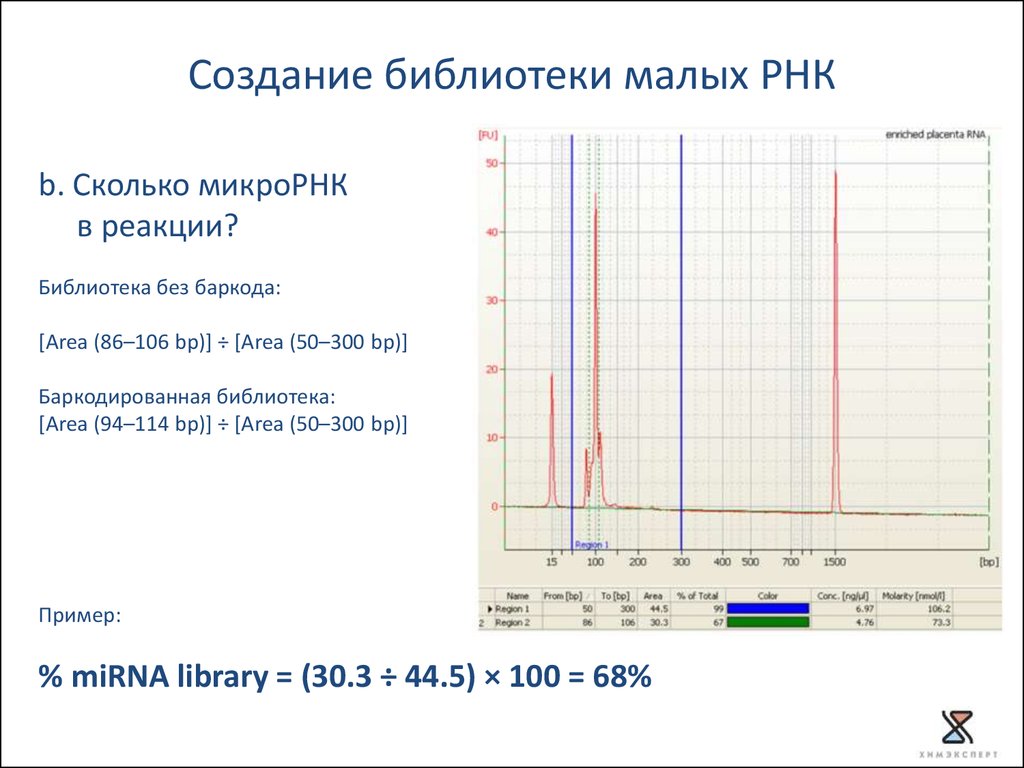
b. Сколько микроРНК в реакции?
c. Концентрация кДНК?
Agilent® 2100 Bioanalyzer® instrument with the Small RNA Kit chip
43.
Создание библиотеки малых РНКb. Сколько микроРНК
в реакции?
Библиотека без баркода:
[Area (86–106 bp)] ÷ [Area (50–300 bp)]
Баркодированная библиотека:
[Area (94–114 bp)] ÷ [Area (50–300 bp)]
Пример:
% miRNA library = (30.3 ÷ 44.5) × 100 = 68%
44.
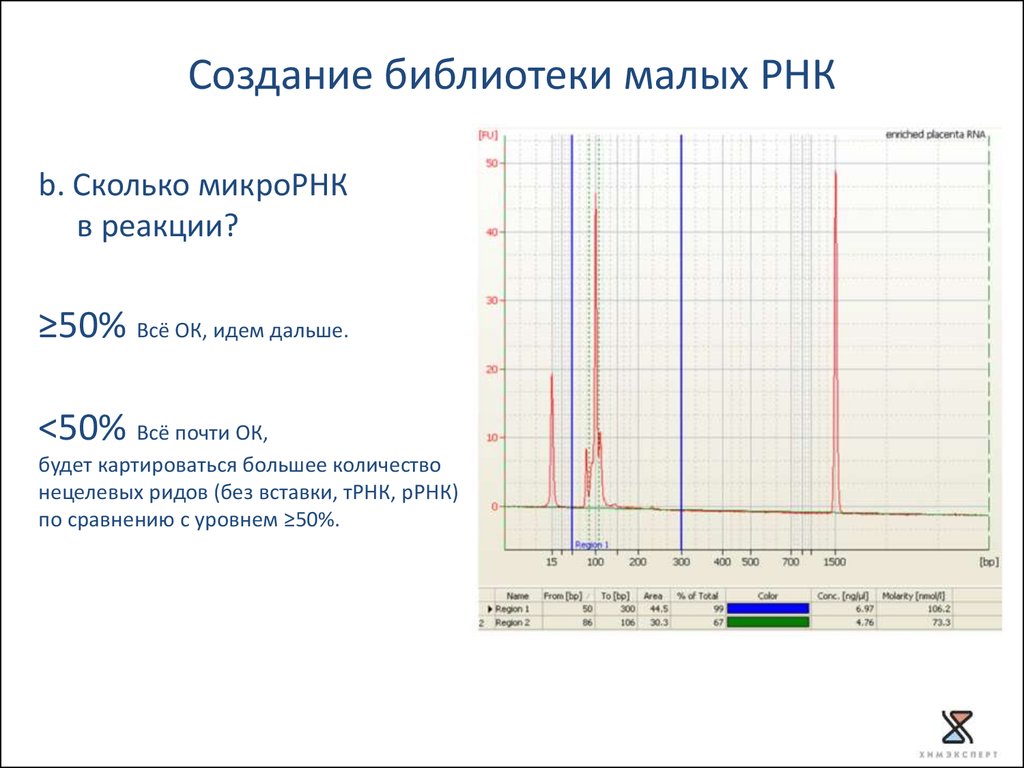
Создание библиотеки малых РНКb. Сколько микроРНК
в реакции?
≥50% Всё ОК, идем дальше.
<50% Всё почти ОК,
будет картироваться большее количество
нецелевых ридов (без вставки, тРНК, рРНК)
по сравнению с уровнем ≥50%.
45.
Создание библиотеки малых РНКРазведение библиотек
• Ion PI™ Template OT2 200 kit (обновления!)
Конечная концентрация: 11 pM.
Пример:
– The library concentration is 2200 pM.
– The library dilution is 2200 pM/11 pM = 200.
– Therefore, 1 µL of library mixed with 199 µL of Low TE (1:200 dilution) yields approximately 11 pM.
• Ion OneTouch™ 200 Template v2 DL
Конечная концентрация: 14 pM.
Пример:
– The library concentration is 2200 pM.
– The library dilution is 2200 pM/14 pM = 157.1
– Therefore, 1 µL of library mixed with 156.1 µL of Low TE (1:157.1 dilution) yields approximately 14 pM.
46.
Шаг 4: Секвенирование47.
Выбор стратегии обогащения зависит от вопроса,на который вы ищете ответ
Стратегия обогащения
Как проводить
Необходимое
кол-во прочтений
Критерии выбора, преимущества
«Глубокоий» полный
транскриптом
Удаление рРНК
80-200M
Detection of low abundance transcripts
Полный транскриптом
Удаление рРНК
30-40M
Enables detection of fusion transcripts, novel
events, alternative splicing
Detect ncRNA
Секвенирование мРНК
Секвенирование коротких
РНК и микроРНК
Отбор поли(А)
фракции
Отбор микроРНК
фракции
15-25M
1-2M (для экспрессии)
2-5M (для обнаружения
новых видов)
Gene level detection & gene expression
quantitation
Similar level of information that is found in
microarray research
Gene level detection & gene expression
quantitation
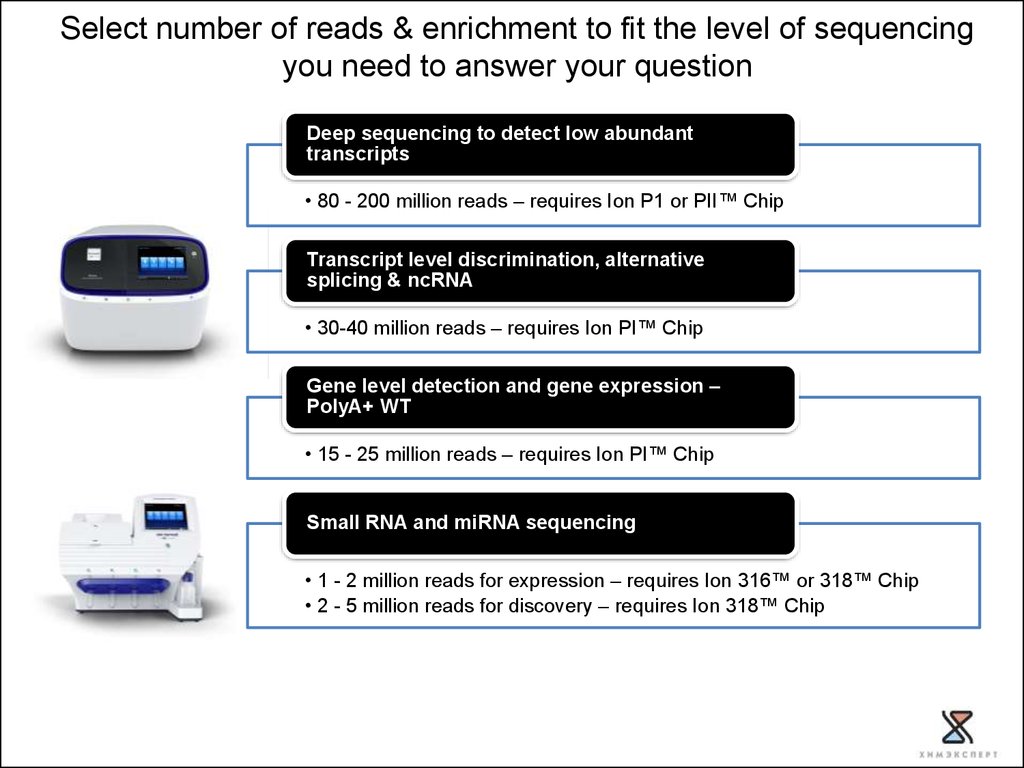
48. Select number of reads & enrichment to fit the level of sequencing you need to answer your question
Select number of reads & enrichment to fit the level of sequencingyou need to answer your question
Deep sequencing to detect low abundant
transcripts
• 80 - 200 million reads – requires Ion P1 or PII™ Chip
Transcript level discrimination, alternative
splicing & ncRNA
• 30-40 million reads – requires Ion PI™ Chip
Gene level detection and gene expression –
PolyA+ WT
• 15 - 25 million reads – requires Ion PI™ Chip
Small RNA and miRNA sequencing
• 1 - 2 million reads for expression – requires Ion 316™ or 318™ Chip
• 2 - 5 million reads for discovery – requires Ion 318™ Chip
49.
Схемы обогащения при дизайне исследованияRNA-Seq
rRNA depletion
• remove ribosomal RNA
to enrich for mRNA,
lincRNA
polyA selection
• select only
polyadenylated RNA’s
(“genes”)
Size/modification
selection
• select for miRNA’s,
nascent transcripts
(GRO-Seq)
RNA-protein
complex selection
• Ribosome associated
(TRAP-Seq), RNAbinding proteins (HITSCLIP)
50.
Технология Ion AmpliSeq™ RNA1. Обратная транскрипция
2. Ген-специфическая ПЦР
3. Лигирование ION-адаптеров
Доступно 96 баркодов
51.
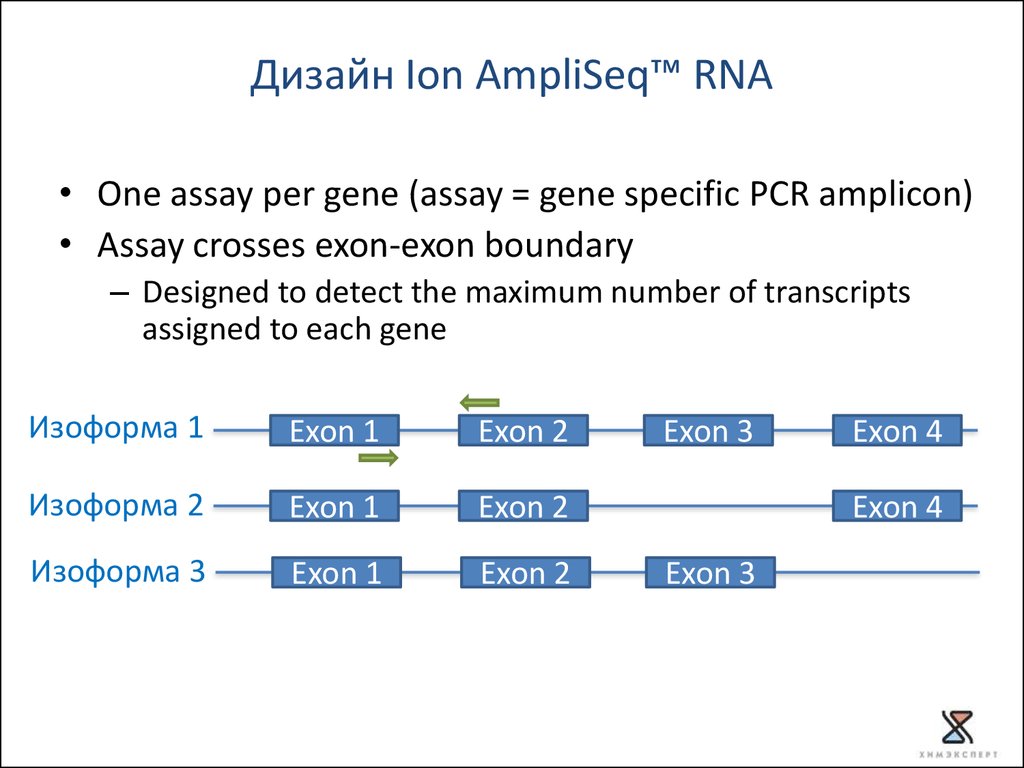
Дизайн Ion AmpliSeq™ RNA• One assay per gene (assay = gene specific PCR amplicon)
• Assay crosses exon-exon boundary
– Designed to detect the maximum number of transcripts
assigned to each gene
Изоформа 1
Exon 1
Exon 2
Изоформа 2
Exon 1
Exon 2
Изоформа 3
Exon 1
Exon 2
Exon 3
Exon 4
Exon 4
Exon 3
52.
Рабочий процесс Ion AmpliSeq™ RNAОбратная транскрипция (~30 мин)
Амплификация выбранных участков при помощи
готовых/пользовательских панелей (~90 мин)
Обрезание праймеров и фосфорилирование (~40 мин)
Лигирование адаптеров (~40 мин)
Очистка на магнитных частицах (~35 мин)
1 round cleanup
Амплификация библиотеки (~10 мин)
2nd PCR
2 round cleanup
Library quantitation
Очистка на магнитных частицах (~20 мин)
Quantitation kit (~45-60 мин)
или BioAnalyzer (~35 мин)
Всего ~5 ½ часов
53.
Ion AmpliSeq™ RNA Fusion Transcript DetectionFusion transcript
5’ Gene
3’ Gene
S. Magdaleno et al. Using Ion AmpliSeq™ RNA to Detect Fusion Transcripts in Human Cancer Samples. © 2013 Life Technologies
54.
Ion AmpliSeq™ RNA design strategy fordetection of fusion transcripts
WT RNAseq
Only a fraction of the reads cross through the
fusion breakpoint
55.
Ion AmpliSeq™ RNA design strategy fordetection of fusion transcripts
Ion AmpliSeq™ RNA strategy for fusion transcripts
Focus reads around breakpoint
Use optimized design strategy for all fusion transcripts into single
custom panel
56.
Ion AmpliSeq™ RNA design strategy fordetection of fusion transcripts
Ion AmpliSeq™ RNA
All reads map to designed breakpoint between fusion gene partners if it is present in
sample.
Allows focused concentration of reads on areas of interest for enhanced sensitivity of
detection of the fusion transcript.
57.
Новые панели для анализа экспрессии:AmpliSeq™ RNA Panels
RNA Cancer Panel
RNA Apoptosis Panel
• 50 генов
• Соответствует генам панели
AmpliSeq Cancer Hotspot v2
• 267 генов
• Валидированные тест-системами
TaqMan®
RNA Stem Cell Panel
RNA Custom Panels
• 243 генов
• Факторы дифференцировки и
плюрипотентности
• До 300 генов на ваш выбор
• Дизайн с 1 пулом
58.
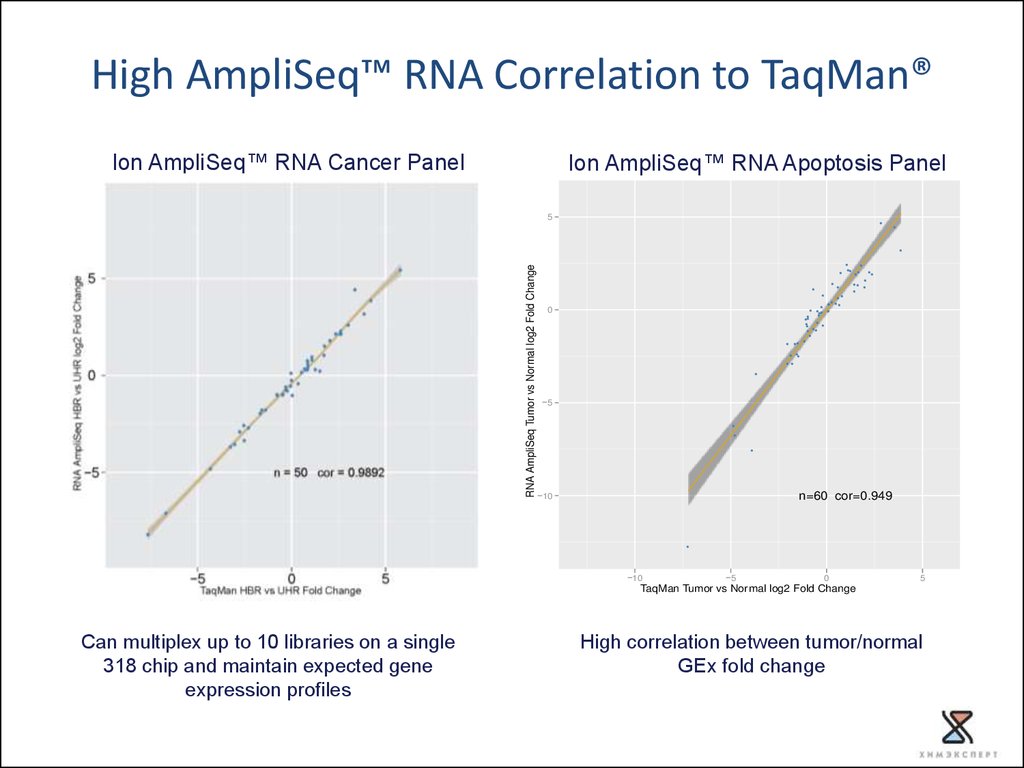
High AmpliSeq™ RNA Correlation to TaqMan®Ion AmpliSeq™ RNA Cancer Panel
Ion AmpliSeq™ RNA Apoptosis Panel
5
RNA AmpliSeq Tumor vs Normal log2 Fold Change
● ● ●
●
●
● ●●
● ●●
●
● ●
● ●
0
●
●
● ●
●
−5
−10
n=60 cor=0.949
−10
−5
0
5
TaqMan Tumor vs Normal log2 Fold Change
Can multiplex up to 10 libraries on a single
318 chip and maintain expected gene
expression profiles
High correlation between tumor/normal
GEx fold change
59.
Ion AmpliSeq™ Transcriptome Human GeneExpression Research Panel
Панель рассчитана на выявление
18 574 кодирующих генов и
2 228 некодирующих генов
на основании аннотации генома человека UCSC hg19
20 802 ампликона (41 604 праймера)
средний размер рида 150 п.о.
1 пробирка.
Кат.ном.:
A26325
A26326
A26327
4471250
Ion AmpliSeq™ Transcriptome Human Gene Expression Kit* 24 rxns
Ion AmpliSeq™ Transcriptome Human Gene Expression Kit* 96 rxns
Ion AmpliSeq™ Transcriptome Human Gene Expression Kit* 384 rxns
Ion Xpress™ Barcode Adapters 1-16 Kit 1 kit
60.
Ion AmpliSeq™ Transcriptome Human GeneExpression Research Panel
Рекомендация: секвенируйте 8 образцов на чипе Proton PI™ Chip.
Измерение кратного изменения экспрессии гена в образцах
Требует 10 нг FFPE общей РНК (истощение по рРНК или полиА+ обогащение не
требуется)
Создание библиотек при помощи Ion AmpliSeq™ Kit Plus и баркодированных
адаптеров Ion Xpress™ Barcode Adapters
Совместим с системами пробоподготовки Ion Chef ™ и Ion OneTouch™ Systems и
секвенаторами Ion Proton™ и Ion PGM™
61.
Ion AmpliSeq™ TranscriptomeImproved gene-level transcript
detection using the
Ion AmpliSeq™
Transcriptome Human Gene
Expression Kit
and the Ion Proton™ System. A
Venn diagram
demonstrating the
concordance of genes
identifi ed by MAQC
microarray data and the
Ion AmpliSeq™ Transcriptome
Human Gene
Expression Kit.
62.
Ion AmpliSeq™ TranscriptomeDetection of significantly
differentially
expressed genes (DEGs) by
using the
Ion AmpliSeq™ Transcriptome
Human Gene
Expression Kit. The Venn
diagram shows the
concordance of significant
DEGs between MAQC
microarray and Ion AmpliSeq™
Transcriptome
Human Gene Expression Kit
data.
63.
Шаг 5: Анализ данных64.
65.
1. Малые РНК (miRNA, snoRNA, piRNA,snRNA, tRNA и др.)
Целевые молекулы для
библиотеки
Используемый подход
Короткая фракция
кДНК
Использование 5’-концевого
фосфата.
Достаточно коротких прочтений
(50-100 п.н.)
66.
2. Уровень экспрессии геновЦелевые молекулы для
библиотеки
Используемый подход
ПолиА-мРНК
Обогащение полиА-фракции.
Часто достаточно коротких
прочтений (50-100 п.н.) с 3’-конца
67.
3. Альтернативный сплайсингЦелевые молекулы для
библиотеки
Используемый подход
Экзон-интронные
стыки
Необходимы длинные прочтения
(более 300 п.н.)
68.
4. Транскриптом одной клеткиЦелевые молекулы для
библиотеки
Используемый подход
ПолиА-мРНК
Необходима предварительная
амплификация библиотеки кДНК
69.
5. Антисмысловые и некодирующие РНКЦелевые молекулы для
библиотеки
Используемый подход
Антисмысловые и
некодирующие РНК
Необходимо создание 5’-3’
ориентированной библиотеки
кДНК.
70.
6. Транслируемые РНКЦелевые молекулы для
библиотеки
Используемый подход
РНК на рибосомах
Ферментативное разрушение РНК
(кроме участков внутри рибосом)
71.
7. Двуцепочечные РНК и вторичныеструктуры на РНК
Целевые молекулы для
библиотеки
Используемый подход
Двуцепочечные
участки РНК
Обработка ацилирующими
агентами






























































 biology
biology informatics
informatics








