Similar presentations:
Репарация и рекомбинация
1.
Московский государственный университет имени М.В. ЛомоносоваОсновы молекулярной
биологии
Репарация и рекомбинация
2.
Причины появления повреждений в ДНК• Ошибки репликации
• Репликация «через повреждения» с использованием
полимераз, отличающихся низкой точностью копирования
• Повреждения ДНК за счет внешнего воздействия
(экзогенные факторы):
облучение
повреждение химическими веществами
(например, алкилирование)
• Повреждения ДНК из-за эндогенных (внутренних)
факторов
гидролиз (депуринизация, дезаминирование)
2 2
3.
Виды репарации1.
Прямая репарация
2.
BER – Base excision repair, репарация вырезанием основания
3.
NER – Nucleotides excision repair, репарация вырезанием
нуклеотидов
4.
MMR – Mismatch репарация, репарация неспаренных
нуклеотидов
5.
SOS-ответ
6.
Репарация при помощи рекомбинации
7.
Репарация двухцепочечных разрывов ДНК
3
4.
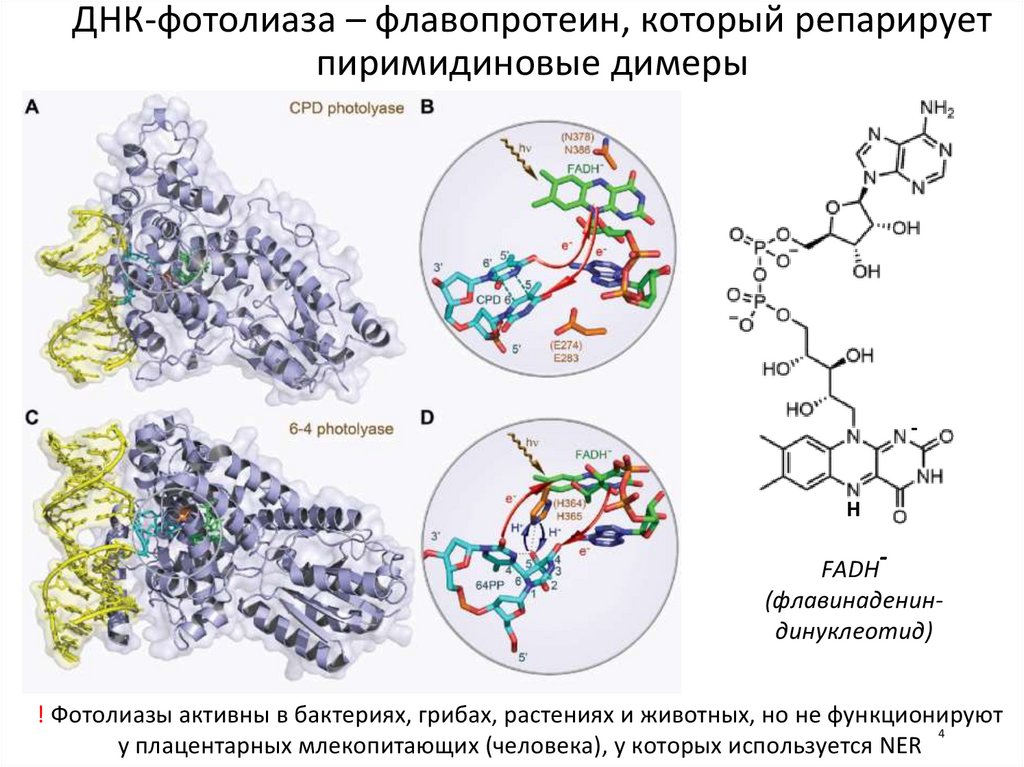
ДНК-фотолиаза – флавопротеин, который репарируетпиримидиновые димеры
-
H
FADH(флавинадениндинуклеотид)
! Фотолиазы активны в бактериях, грибах, растениях и животных, но не функционируют
4
у плацентарных млекопитающих (человека), у которых используется NER
5.
Прямая репарацияТиминовые (пиримидиновые) димеры могут образовываться между двумя
соседними основаниями под воздействием ультрафиолетового излучения
(например, от солнечного света)
cyclobutane pyrimidine
80%
dimer (CPD)
20%
pyrimidine-pyrimidone (6-4)
photoproduct (6-4PP)
6.
Механизм действия ДНК-фотолиазыФотоантенна
MTHF
или
8-HDF
MTHF*
или
8-HDF*
6
7.
Прямая репарация:MGMT деметилирует m6G и m4T
(O6 -MG)-DNA-метилтрансфераза (MGMT или AGT– у высших эукариот, Ada и Ogt – E.coli)
7
переносит метильный остаток с гуанина на остаток цистеина в своём активном сайте.
8.
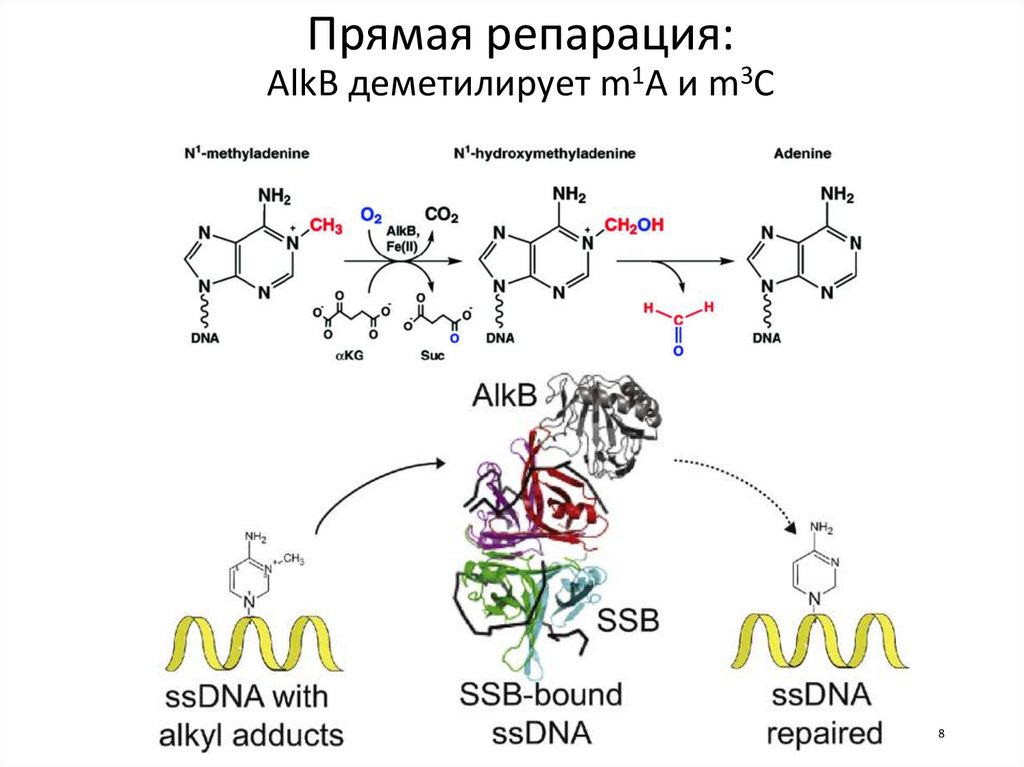
Прямая репарация:AlkB деметилирует m1A и m3C
8
9.
Base excision repair (BER): ДНК-гликозилазы1.Дезаминированные основания
(С - > U; G - > ксантин; A - > гипоксантин)
Урацил-ДНКгликозилаза
ксантин
гипоксантин
1.Окисленные основания (o8G, FapyG, FapyA)
8-оксогуанин
FapyG (2,6-диамино-4-гидрокси-5формамидопиримидин)
1.Алкилированные основания (m3A, m7G)
3-метиладенин
7-метилгуанин
9
10.
BER у бактерий10
11.
BER у эукариотShort patch (1 nt)
Long patch (2-10 nt)
11
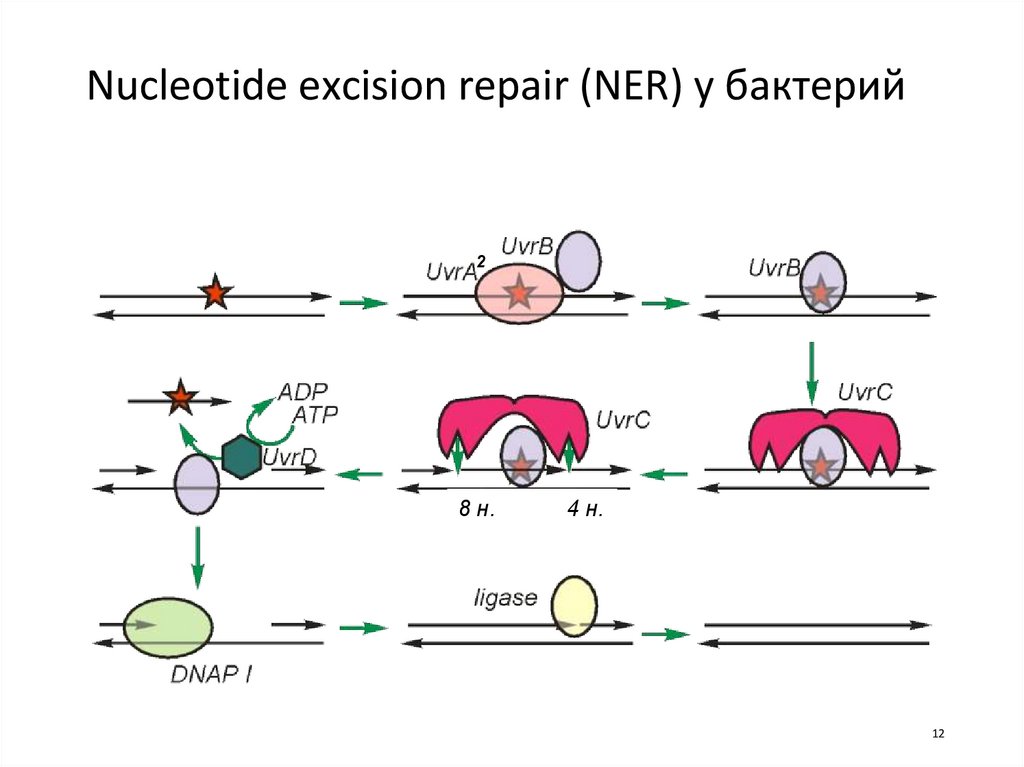
12.
Nucleotide excision repair (NER) у бактерий2
8 н.
4 н.
12
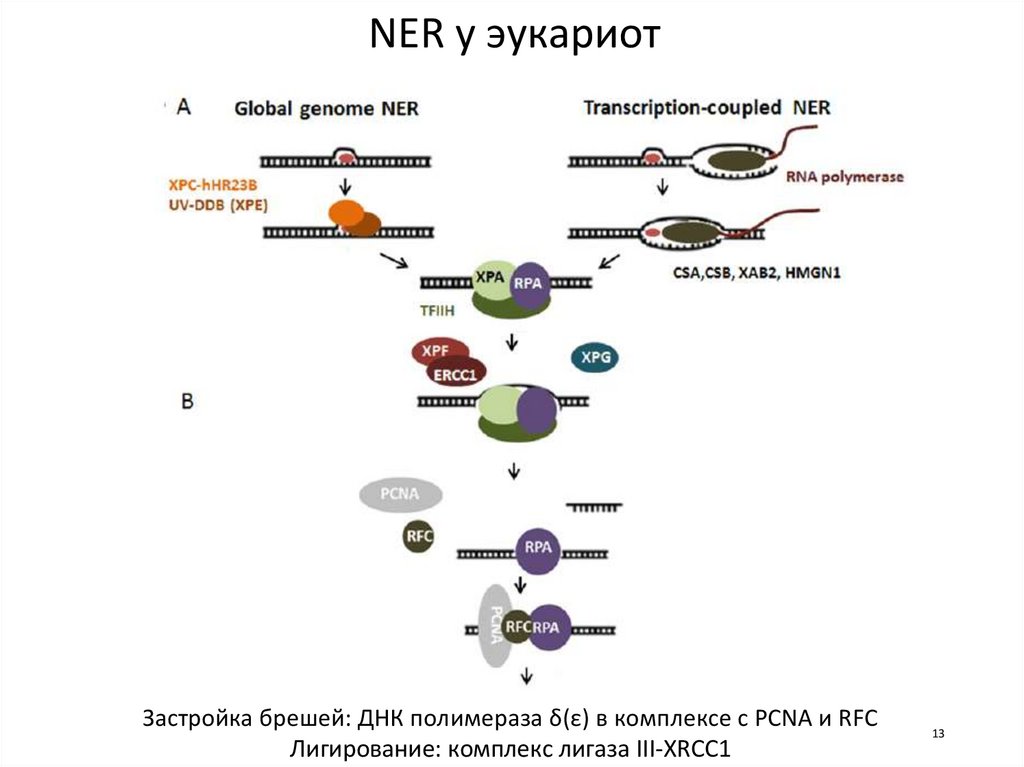
13.
NER у эукариотЗастройка брешей: ДНК полимераза δ(ε) в комплексе с PCNA и RFC
Лигирование: комплекс лигаза III-XRCC1
13
14.
Репарация неспаренных нуклеотидов(mismatch) у грамм-отрицательных бактерий
=SSB
14
15.
Mismatch репарация у эукариот15
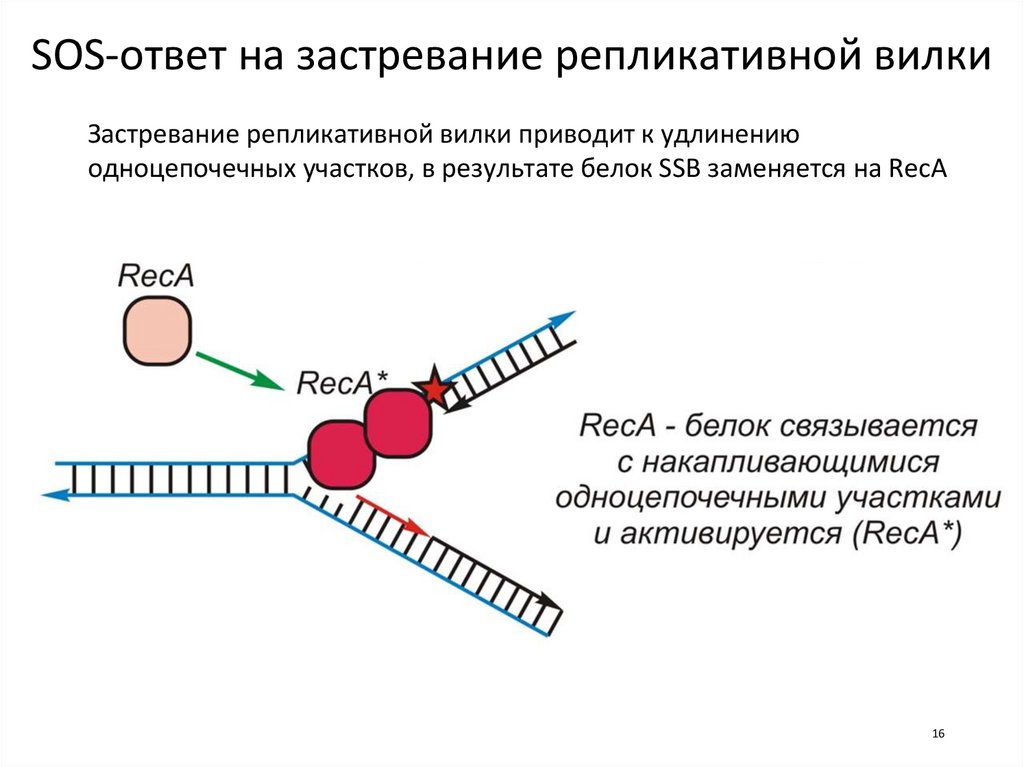
16.
SOS-ответ на застревание репликативной вилкиЗастревание репликативной вилки приводит к удлинению
одноцепочечных участков, в результате белок SSB заменяется на RecA
16
17.
SOS-ответ17
18.
SOS-ответ18
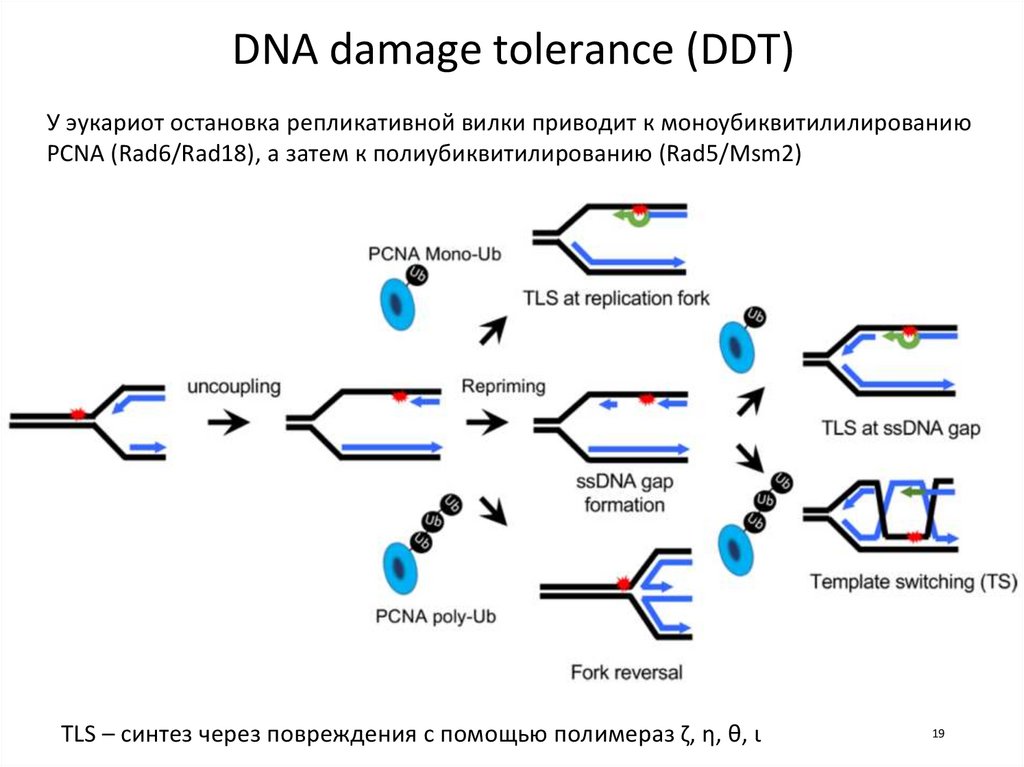
19.
DNA damage tolerance (DDT)У эукариот остановка репликативной вилки приводит к моноубиквитилилированию
PCNA (Rad6/Rad18), а затем к полиубиквитилированию (Rad5/Msm2)
TLS – синтез через повреждения с помощью полимераз ζ, η, θ, ι
19
20.
Двухцепочечные разрывы ДНК! Бактерии почти всегда используют гомологичную рекомбинацию
20
21.
Негомологичное соединение концов (NHEJ) у эукариотУ E.coli такой системы нет, у некоторых других бактерий (B.subtilis, M.tuberculosis)
есть аналогичная система, для которой необходимы только гомодимер Ku и лигаза D
21
22.
Виды рекомбинации22
23.
Гомологичная рекомбинация23
24.
Гомологичная рекомбинация убактерий
24
25.
Гомологичная рекомбинация убактерий
25
26.
Гомологичная рекомбинация26
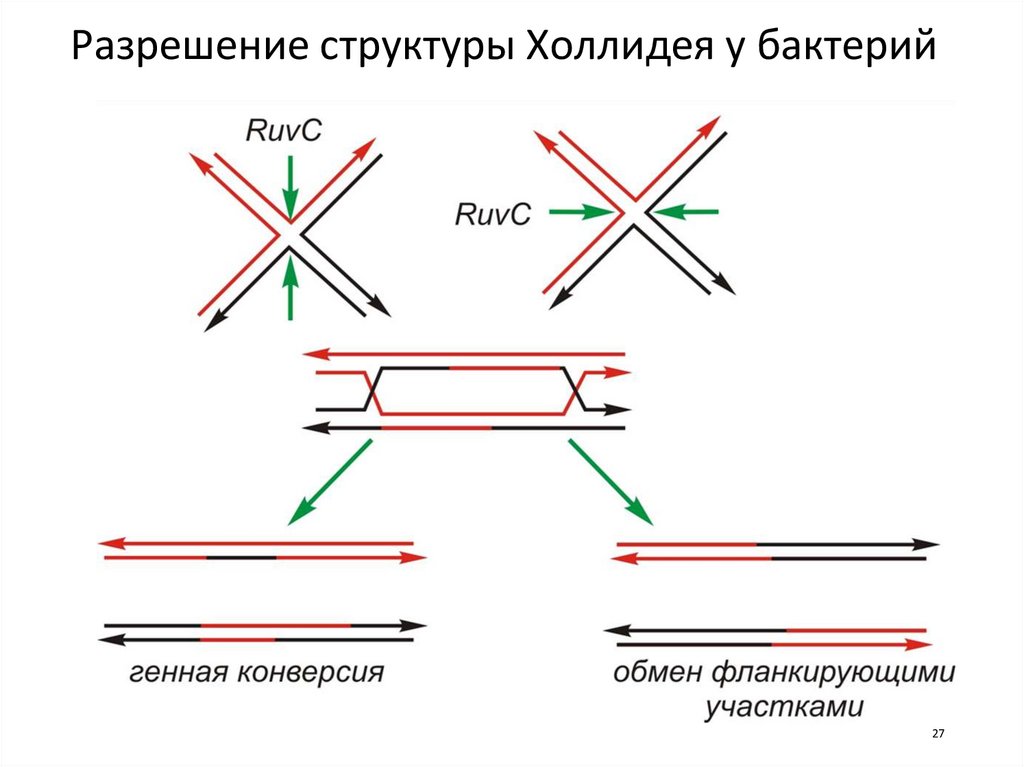
27.
Разрешение структуры Холлидея у бактерий27
28.
Симметрия структуры Холлидея ивзаимодействующие с ней белки бактерий
28
29.
Гомологичная рекомбинация у эукариот29
30.
Гомологичная рекомбинация у эукариотMRN
CtIP
WRN
MutLγ, Exo1, Sgs1
30
31.
V(D)J-рекомбинацияVariable Diversity Joining
31
32.
V(D)J-рекомбинация: лёгкая цепь32
33.
V(D)J-рекомбинация: тяжёлая цепь33
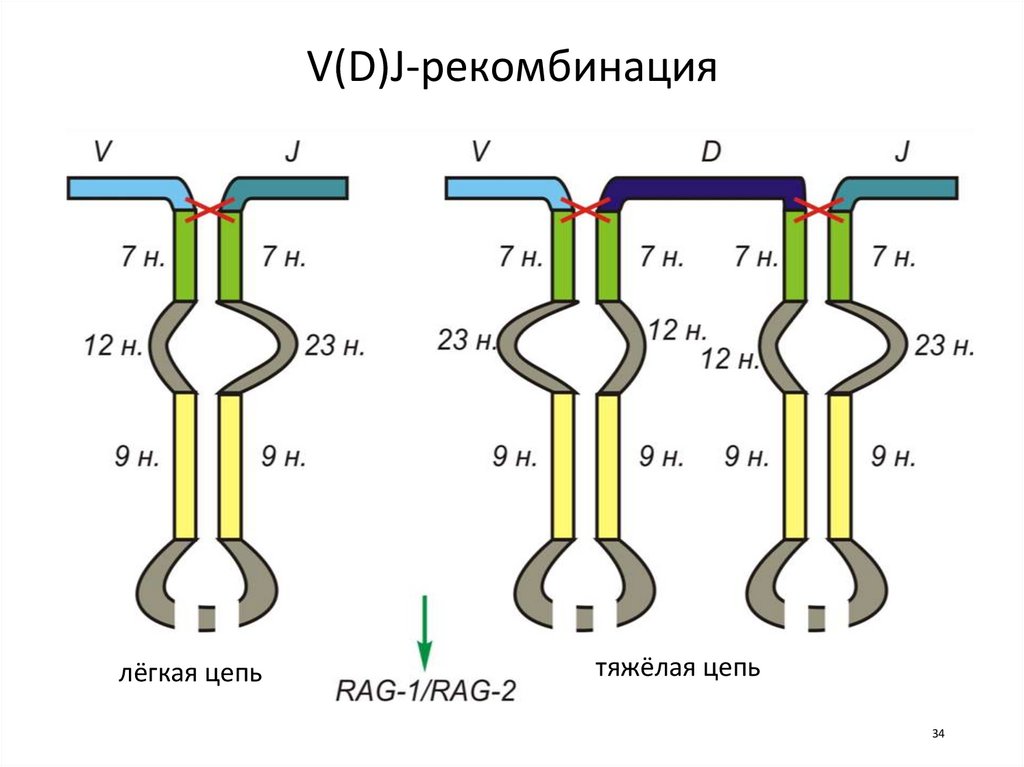
34.
V(D)J-рекомбинациялёгкая цепь
тяжёлая цепь
34
35.
V(D)J-рекомбинацияКu70 и Ku80, Artemis, XRCC4, лигаза IV – соединяют V, D, J
сегменты (негомологичное соединение концов)
35



























 biology
biology








