Similar presentations:
Исследование повторяющихся последовательностей в Aegilops ventricosa
1. Исследование повторяющихся последовательностей в Aegilops ventricosa
ФГБОУ ВО РГАУ - МСХА имени К.А. ТимирязеваКафедра селекции и семеноводства
Выполнила – Минькова Яна Вадимовна
Научный руководитель – Дивашук Михаил Георгиевич, кандидат биологических
наук, доцент кафедры генетики, селекции и семеноводства ФГБОУ ВО «Российский
государственный аграрный университет – МСХА им. К.А. Тимирязева»
2. Актуальность
3. Цели и задачи:
Цель: Получение обобщенной совокупной информации околичественном и качественном составе повторяющихся
последовательностей ДНК на примере Aegilops ventricosa.
Задачи:
1. Первичная обработка сиквенсов 4 образцов Aegilops tauschii и 1
образца Aegilops ventricosa после проведения полногеномного
секвенирования на платформе Illumina.
2. Кластерный анализ с помощью пайплайна RepeatExplorer2.
3. Отбор и анализ полученных кластеров.
4. Методика проведения исследований
Выделение ДНК СТАВ-методомСеквенирование с помощью NGS (Illumina NextSeq) пяти образцов Aegilops:
Aegilops Tauschii T21.1 (KK1);
Aegilops Tauschii T36.1 (KK2);
Aegilops Tauschii 44.1(KK3);
Aegilops Tauschii T22.1 (KK4);
Aegilops ventricosa T38.1 (KK5)
5.
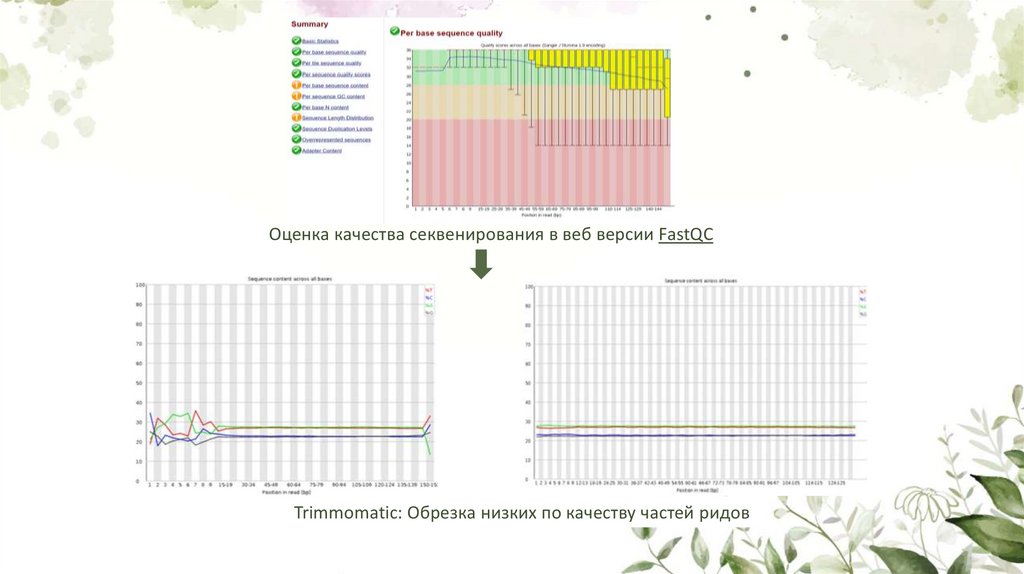
Оценка качества секвенирования в веб версии FastQCTrimmomatic: Обрезка низких по качеству частей ридов
6.
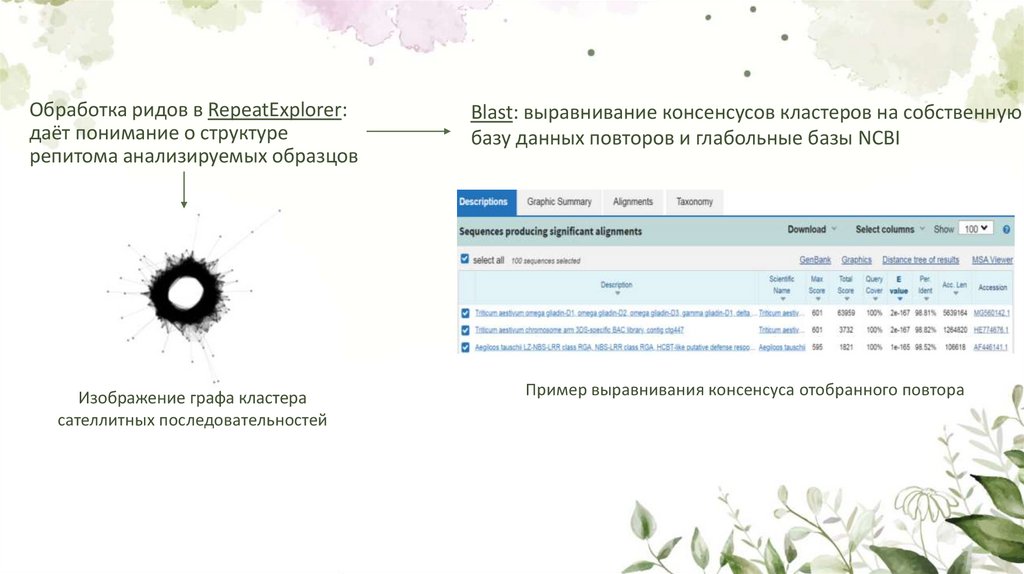
Обработка ридов в RepeatExplorer:даёт понимание о структуре
репитома анализируемых образцов
Blast: выравнивание консенсусов кластеров на собственную
базу данных повторов и глабольные базы NCBI
Изображение графа кластера
сателлитных последовательностей
Пример выравнивания консенсуса отобранного повтора
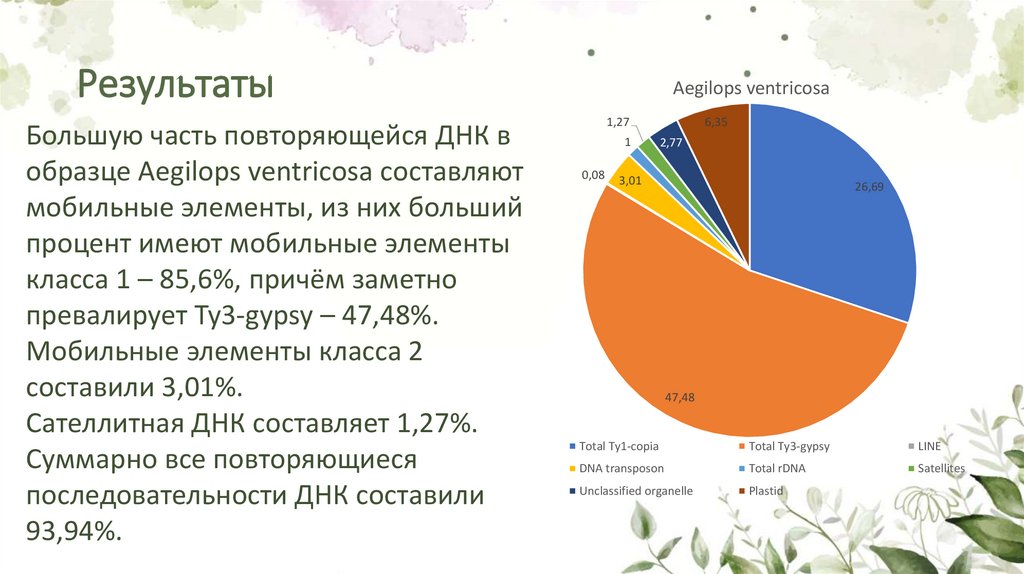
7. Результаты
Большую часть повторяющейся ДНК вобразце Aegilops ventricosa составляют
мобильные элементы, из них больший
процент имеют мобильные элементы
класса 1 – 85,6%, причём заметно
превалирует Ty3-gypsy – 47,48%.
Мобильные элементы класса 2
составили 3,01%.
Сателлитная ДНК составляет 1,27%.
Суммарно все повторяющиеся
последовательности ДНК составили
93,94%.
Aegilops ventricosa
1,27
1
0,08
6,35
2,77
3,01
26,69
47,48
Total Ty1-copia
Total Ty3-gypsy
LINE
DNA transposon
Total rDNA
Satellites
Unclassified organelle
Plastid
8. Результаты
9. Результаты
Повтор kk5kk1cl16910. Выводы
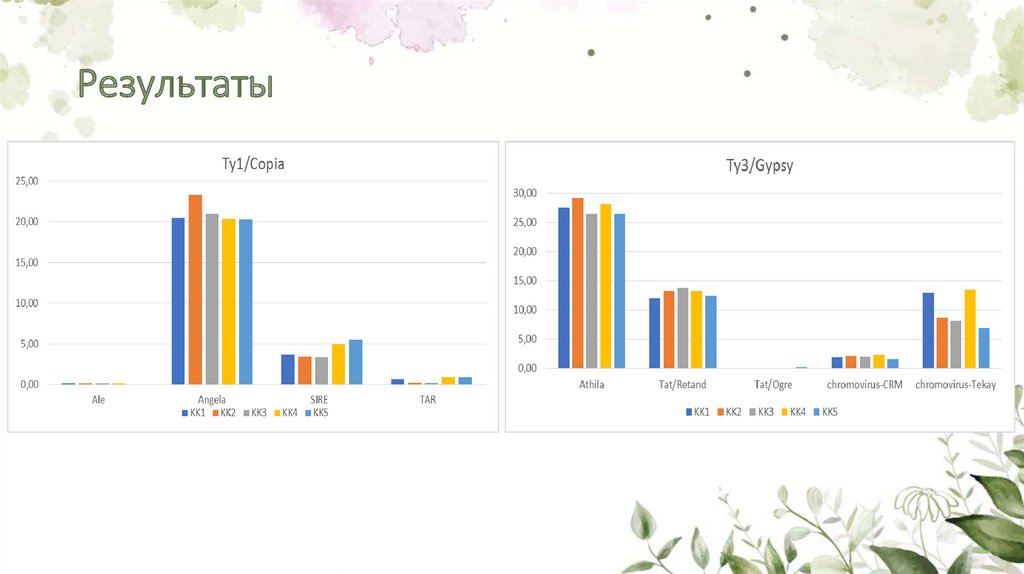
1. В результате биоинформатического анализа с помощью пайплайнаRepeatExplorer сиквенсов 4 образцов Aegilops tauschii и 1 образца Aegilops
ventricosa было установлено, что копийность LTR-ретротранспозонов Ty3-gypsy
у Aegilops tauschii составляет 53,83%, у Aegilops ventricosa 47,48%; копийность
LTR-ретротранспозонов Ty1-copia составляет 25,73% и 26,69% соответсвенно
2. После проведения биоинформатического анализа было установлено, что
среди LTR-ретротранспозонов Ty3-gypsy наибольшую копийность имеет
семейство Athilla (в образцах Aegilops tauschii – 27,85%, в Aegilops ventricosa –
26,43%); среди Ty1-copia наибольшую копийность показало семейство Angela
(в образцах Aegilops tauschii 21,29%, в Aegilops ventricosa – 20,29%)
11. Выводы
3. С помощью биоинформатических методов было установлено три повтора,гипотетически более специфичные для генома Aegilops ventricosa, поскольку
количество ридов находится в соотношении более 80% от ventricosa и менее 20% от
tauschii. Эта пропорция сохраняется вне зависимости от образца Aegilops tauschii.
Один из них (kk5kk1cl169) имеет гомологичные повторы из базы NCBI найденные в
мягкой пшенице
4. В результате биоинформатического анализа была отмечена полиморфность
последовательности повтора kk5kk1cl164, в результате повторных запусков с
образцом kk2, kk3 и kk4, однако идентичность последовательности мономера
повтора в запуске с образцом kk3 гораздо нижеВозможно говорить о различиях в
последовательности данного повтора в зависимости от образца, что может быть
использовано в дальнейших исследованиях в зависимости от поставленных задач.