Similar presentations:
Масс-спектрометрия в протеомных исследованиях. Часть 2: Приложения
1.
МАСС-СПЕКТРОМЕТРИЯВ ПРОТЕОМНЫХ ИССЛЕДОВАНИЯХ
Часть 2: Приложения
2. ПРОТЕОМИКА – набор высокотехнологичных методов установления состава сложных смесей белков
1)2)
3)
4)
определение количества того или иного белка в образце;
идентификация белка;
уточнение первичной структуры;
определение пост-трансляционных модификаций.
2D-электрофорез
Специфический гидролиз
MALDI-MS
Специфический гидролиз
Хроматография
ESI-MS-MS, микросеквенирование
Белковые чипы
3. Масс-спектрометрия целых белков
В базе данныхNCBI nr более 2 Для идентификации белка по его молекулярной массе
необходима точность ее измерения < 2ppm
500 000 записей
Точность недостижима при использовании времяпролетной массспектрометрии, ионных ловушек
Такая точность доступна при использовании масс-спектрометрии ИЦР
Изучаемый белок как правило отличен от имеющегося в базе данных
Пример: при анализе всех цитозольных белков бактерии Rhodobacter
с использованием масс-спектрометрии ИЦР идентифицировано около 10%
белков, пики которых наблюдались в спектре. Массы остальных белков
были отличны от рассчитанных по гену из-за наличия пост-трансляционных
модификаций.
4. Профилирование белков
Подложки с разными типами поверхности:обращенная фаза, анионообменная,
катионообменная.
Биомаркер рака яичников
10000
12000
14000
20
КОНТРОЛЬНАЯ
СЫВОРОТКА
15
Для идентификации белков отличий
необходимо их выделение.
10
20
10000
12000
14000
РАК МАТКИ
15
Детектируются, как правило, хорошо
представленные белки.
10
5
17.5
10000
15
12000
14000
12000
12000
14000
14000
РАК ЯИЧНИКОВ
12.5
10
7.5
10000
10000
В данном случае –
сывороточный амилоид А1
5. Стратегия анализа: 2DE + MALDI-MS
2D электрофорез белковКомпьютерный анализ изображений гелей
Вырезание и трипсинолиз белков в
фрагментах геля
Экстракция пептидов и получение
MALDI масс-спектра суммарного
гидролизата
Поиск в базе данных
измеренных масс пептидов
(пептидный фингерпринт)
Post Source Decay-MS, TOF-TOF
фрагментация отдельных пептидов
Поиск в базе данных
измеренных масс фрагментов
6. 2D-Электрофорез
Первое направление – изоэлектрическая фокусировкаВторое направление – разделение по массам в полиакриламидном геле
Белки, растворенные в неионном детергенте,
вносятся в гель с фиксированным градиентом рН
Эффективное разделение
по массам белков 10-200 кДа
Электорофорез по Лэмли:
Добавляется додецилсульфат Na:
сильный детергент
вносит отрицательный заряд
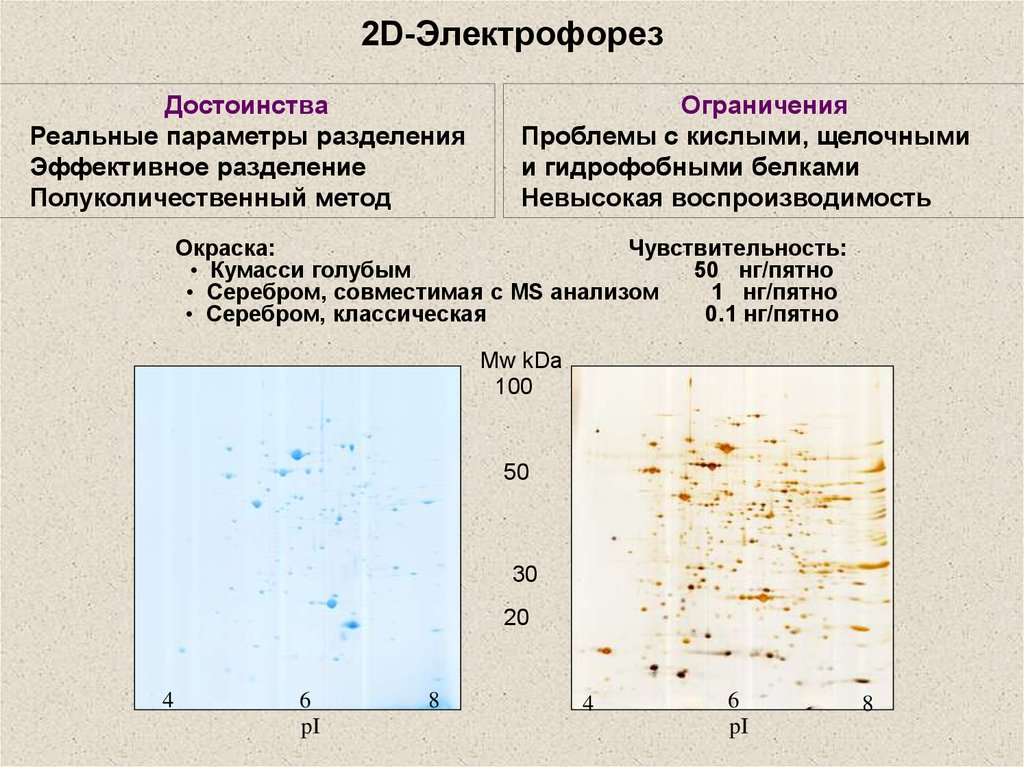
7. 2D-Электрофорез
ДостоинстваРеальные параметры разделения
Эффективное разделение
Полуколичественный метод
Ограничения
Проблемы с кислыми, щелочными
и гидрофобными белками
Невысокая воспроизводимость
Окраска:
Чувствительность:
• Кумасси голубым
50 нг/пятно
• Серебром, совместимая с MS анализом
1 нг/пятно
• Серебром, классическая
0.1 нг/пятно
Mw kDa
100
50
30
20
4
6
pI
8
4
6
pI
8
8. 2D-Электрофорез
Окраска DIGE Cy3/Cy5Электрофорез 2-х предварительно
окрашенных белков
Mycoplasma gallisepticum
проводится на одном геле.
Зеленым (Cy3) – белки из
контрольных клеток,
Красным (Cy5) – белки из
обработанных клеток.
Достоинства: Очень чувствителен,
прекрасное сопоставление
пятен, очень точен и удобен для
оценки экспрессии белка
Недостатки: Недолговечность
флуоресценции (сутки),
требует дорогих сканеров,
непригоден для вырезания
пятен без дополнительной
окраски серебром
9. Схема проведения идентификации белка по его пептидному фингерпринту
878.5x10 4
1.5
1849.7
1125.7
1490.7
1546.8
902.5
Вырезание отдельных белковых пятен
(>20 нг/мм2)
Трипсинолиз белков в фрагментах геля
Получение масс-спектра
Intens. [a.u.]
Схема проведения идентификации белка
по его пептидному фингерпринту
2869.3
2735.1
2290.0
2005.7
2097.0
2147.8
2201.1
1794.7
1273.7
1198.7
976.4
0.5
1612.7
1096.6
687.4
1929.9
1.0
0.0
750
1000
1250
1500
1750
2000
2250
2500
2750
3000
m /z
выбор базы данных
ограничение поиска по видовой принадлежности
учет возможных модификаций пептидов
точность измеренных масс
список масс пептидных фрагментов
количество кандидатов
www.matrixscience.com
10. Немного статистики:
В базе данныхNCBI nr более 2
500 000 записей
Среднее количество пептидов разной длины,
образующихся в результате полного гидролиза
Триптические пептиды 1000-2000Да
точность (%)
0.1
0.01
0.001
кол-во кандидатов
>1000
100-1000
5-500
Распределение моноизотопных масс пептидов
«уникальные»
11. Критерии достоверности поиска белков в базах данных: peptide fingerprint
1.2.
3.
4.
5.
6.
7.
количество «совпавших» пептидов, в предположении одного белка
количество «совпавших» пептидов, в предположении нескольких белков
распределение «совпавших» пептидов по точности
«уникальность» пептидов
перекрывание «совпавшими» пептидами последовательности белка
паттерны протеолиза
oтклонение массы белка от предполагаемой и т. д.
база данных
измеренные m/z
Приоритет критериев
2, 4, 7
программа Profound
Score
Приоритет критериев
1, 5, 6
программа Mascot
12. Идентификация белка заменяется нахождением его ближайшего гомолога
Изучаемый белок обычно отличен от имеющегося в базе данныхмассы
20 кандидатов
случайное
совпадение
достоверный
поиск
вероятность
20 кандидатов в порядке убывания достоверности поиска
1.
2.
3.
4.
20.
N
Масса Вероятность
Описание
gi|8486123 27875 88 reading frame [Influenza A virus]
gi|4996872 26451 74 M1 [influenza A virus (A/Taiwan/96/1769)]
gi|324260 27872 73 M1 subunit [Influenza A virus]
gi|75116
27866 73 M1 - influenza A virus (strain A/WSN/33)
------------gi|6048806 27218 58 M1 [Influenza A virus (A/Duck/Hong Kong/698/79 (H5N3))]
Sequence Coverage: 35%
IVPSGPLKAE IAQRLEDVFA
FTLTVPSERG LQRRRFVQNA
EIALSYSAGA LASCMGLIYN
QMVTTTNPLI RHENRMVLAS
QAMRTIGTHH SSSAGLKDDL
GKNTDLEVLM
LNGNGDPNNM
RMGTVTTEVA
TTAKAMEQMA
LENLQAYQKR
EWLKTRPILS
DKAVKLYRKL
FGLVCATCEQ
GSSEQAAEAM
MGVQMQRFK
PLTKGILGFV
KREITFHGAK
IADSQHRSHR
DIASQARQMV
13. Cпектр гидролизата смеси белков (1DE)
AccessionMass Score
1. gi|4249653 53914 154
2. gi|10834998 57381 149
3. gi|6470141 57353 138
4. gi|6166183 60776 134
5. gi|6325467 60794 123
6. gi|13643376 60921 113
7. gi|284102
60462 111
8. gi|5705937 55015 73
9. gi|226295
55481 73
10. gi|219571
56182 72
11. gi|13699818 56515 72
……………………
18. gi|4758794 776492 64
Description
CYTOCHROME P450 2E1
----//-------//---DIMETHYLANILINE MONOOXYGENASE
----//-------//-------//---CYTOCHROME P-450 IIC
----//-------//-------//---NEBULIN
14. 2DE + MALDI-MS - результаты
Mycoplasma gallisepticum strain RMW
Protein coding genes - 726
200
150
100
70
Белковых пятен
> 400
Проанализировано
>150
50
30
Индивидуальных белков
Mycoplasma gallisepticum 105
20
10
pI
4.5
5
5.5
6
6.5
7
7.5
8
15. Примеры анализа 2DE карт
А70
Примеры анализа 2DE карт
Б
2DE карты клеточных белков
двух штаммов Helicobacter pylori
Всего видно на геле ~ 500 белковых пятен
Видимых различий ~ 200
Идентифицировано ~ 150 разных белков
Из них белки отличия ~ 100
40
20
pI 4
6
8
pI 4
А
70
6
8
Б
2DE карты белков штамма клеток E.coli (А)
и штамма-продуцента треонина (Б)
Всего видно на геле ~1200 белковых пятен
Видимых различий ~ 50
Идентифицировано ~ 30 белков отличия
40
20
pI 4
6
8
pI 4
А
6
8
2DЕ карты белков клеточной линии
аденокарциномы молочной железы MCF7:
контрольной (А) и устойчивой к Hoechst33342 (Б)
Б
70
Всего видно на геле ~ 2500 белковых пятен
Видимых различий ~ 30
Идентифицировано ~ 10 белков отличия
40
20
pI 4
6
8
pI 4
6
8
16.
Стратегия анализа: 1D - LC + ESI-MS/MS1D электрофорез белков
Вырезание и трипсинолиз белков в полосах геля
Экстракция и хроматографическое
разделение пептидов
Intens.
x10 4
50.
+MS, 47.4-48.0mi n #2800-#2818
2+
710.6
8
Получение масс-спектров
пептидов и их
фрагментации
6
4
533.6
2
1+
445.4
2+
293.3
1+
391.6
610.3
205.3
972.9
1603.0
1034.0
0
200
400
600
800
1000
1200
1400
1600
m/z
Intens. 50.
x10 4
+MS2(710.6), 47.5-48.0min #2801-#2819
1+
791.4
1.0
0.8
0.6
Объединение данных
и анализ
1+
629.3
0.4
1+
943.5
0.2
1+
477.2
1238.7
890.5
530.3
1139.6
1003.6
0.0
400
600
800
1000
1200
m/z
17. Способы фрагментации пептидов
LID – фрагментация, индуцированная лазеромПептид поглощает энергию лазера, захваченная энергия перераспределяется по
молекуле, разрушается ближайшая слабая связь.
Основной тип образовавшихся пептидных фрагментов - b-ионы и y-ионы.
Может происходить отщепление модификаций.
CID – фрагментация, индуцированная столкновениями
Пептид приобретает энергию от столкновений, захваченная энергия
перераспределяется по молекуле, разрушается ближайшая слабая связь.
Основной тип образовавшихся пептидных фрагментов - b-ионы и y-ионы.
Дополнительно образуются a-ионы и x-ионы.
Часто происходит отщепление модификаций.
ECD – фрагментация, индуцированная захватом медленных электронов
Точный механизм неизвестен. Пептид захватывает медленные электроны на азот
пептидной связи, происходит мгновенный разрыв пептидной цепи с образованием
z- ионов.
Не происходит отщепления модификаций.
18. Интерпретация спектров распада пептидов
Intens. 50.x10 4
+MS2(710.6), 47.5-48.0min #2801-#2819
1+
791.4
TIGTHPSSSAGLK
1.0
b
102.0
215.1
272.2
373.2
510.3
607.3
694.4
781.4
868.4
939.5
996.5
1109.6
1237.7
0.8
0.6
1+
629.3
0.4
1+
943.5
[MH]2+
0.2
1+
477.2
1238.7
890.5
530.3
1139.6
1003.6
0.0
400
600
800
1000
1200
m/z
1
2
3
4
5
6
7
8
9
10
11
12
13
Res.
T
I
G
T
H
P
S
S
S
A
G
L
K
y
13
12
11
10
9
8
7
6
5
4
3
2
1
1154.6
1041.5
984.5
883.5
746.4
649.4
562.3
475.3
388.3
317.2
260.2
147.1
19. Критерии достоверности поиска белков в базах данных: peptide fingerprint + MS/MS
база данныхизмеренные m/z
Score
1.
Отбор из базы данных всех кандидатов,
содержащих триптические пептиды указанных m/z
2.
«Примерка» спектров фрагментации (MS/MS) :
количество «совпавших» фрагментов пептидов
«уникальность» спектров фрагментации пептидов
3.
Score белка = S Score пептидов
Score пептидов
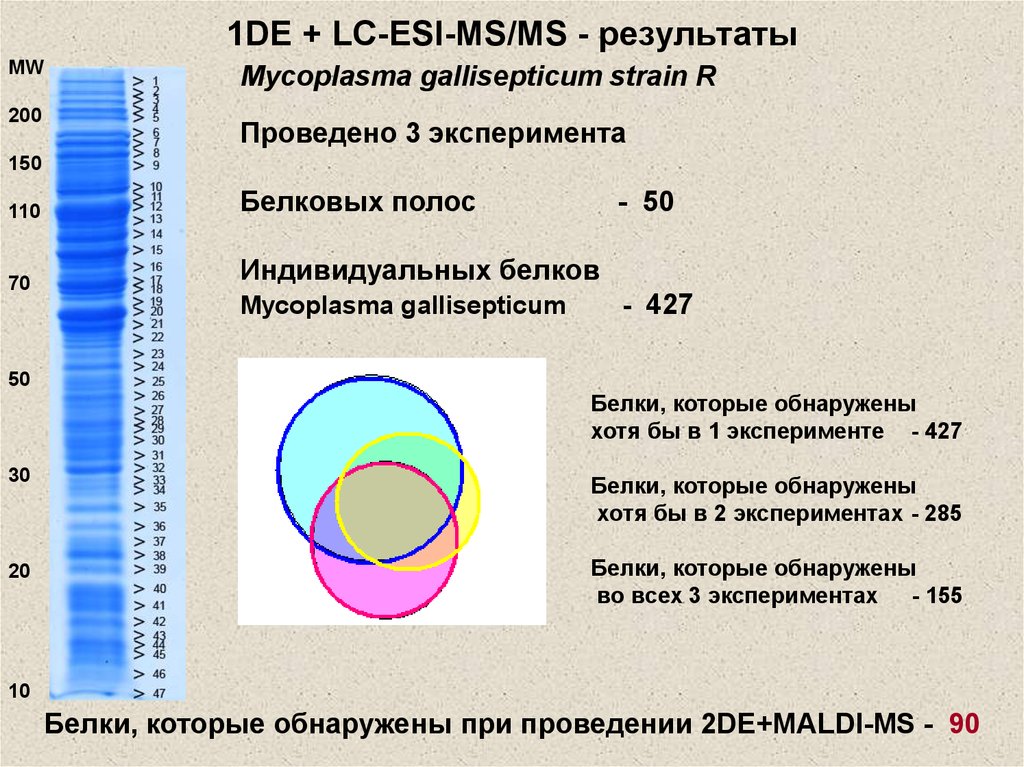
20. 1DE + LC-ESI-MS/MS - результаты
MWMycoplasma gallisepticum strain R
200
Проведено 3 эксперимента
150
110
Белковых полос
70
Индивидуальных белков
Mycoplasma gallisepticum
50
Эксп.1
30
Эксп.3
- 427
Белки, которые обнаружены
хотя бы в 1 эксперименте - 427
Белки, которые обнаружены
хотя бы в 2 экспериментах - 285
155 ?
20
- 50
Эксп.2
Белки, которые обнаружены
во всех 3 экспериментах - 155
10
Белки, которые обнаружены при проведении 2DE+MALDI-MS - 90
21. СОПОСТАВЛЕНИЕ РЕЗУЛЬТАТОВ: 2DE + MALDI-MS и 1DE + LC-ESI-MS/MS
№ NCBIgi|31541381
gi|31541566
gi|31541571
gi|31541607
gi|31541608
mW
score
85818 119
100472 130
120325 273
130197 205
133826 168
Protein
№ NCBI
conserved hypothetical lipoprotein gi|31541381
conserved hypothetical lipoprotein gi|31541566
HepA/SNF
gi|31541753
conserved hypothetical lipoprotein gi|31541571
conserved hypothetical lipoprotein gi|31541607
lipoprotein
gi|45453612
lipoprotein
gi|45453638
lipoprotein
gi|33327196
conserved hypothetical lipoprotein gi|31541608
mW
score
85818 127
100472 817
132618 341
120325 2152
130197 1781
18284
313
18339
362
25889
436
133826 1008
conserved
conserved
conserved
conserved
conserved
Protein
hypothetical lipoprotein
hypothetical lipoprotein
hypothetical lipoprotein
hypothetical lipoprotein
hypothetical lipoprotein
22. Стратегия анализа: LC + ESI-MS/MS
Специфический гидролизсуммарного белка + мечение ICAT
Стратегия анализа:
LC + ESI-MS/MS
Выделение цистеинсодержащих пептидов
(афинное связывание с авидином)
Хроматографическое
разделение пептидов + MS/MS
Пример: ICAT-метки
Достоинства:
Анализируются все меченные белки
Анализ содержания одного белка в
контроле и опыте
Ограничения:
Не видно взаимных количеств
разных белков в образце
Артефакты в интерпретации MS/MS
23. Интерпретация спектров распада пептидов (LID +CID)
Сиквенирование de-novo:•определение пар пиков, в сумме дающих массу родительского иона
•по наличию соседних пиков +/-28, +/-15 определение типа ионов
•по разнице масс соседних однотипных ионов построение
возможных последовательностей
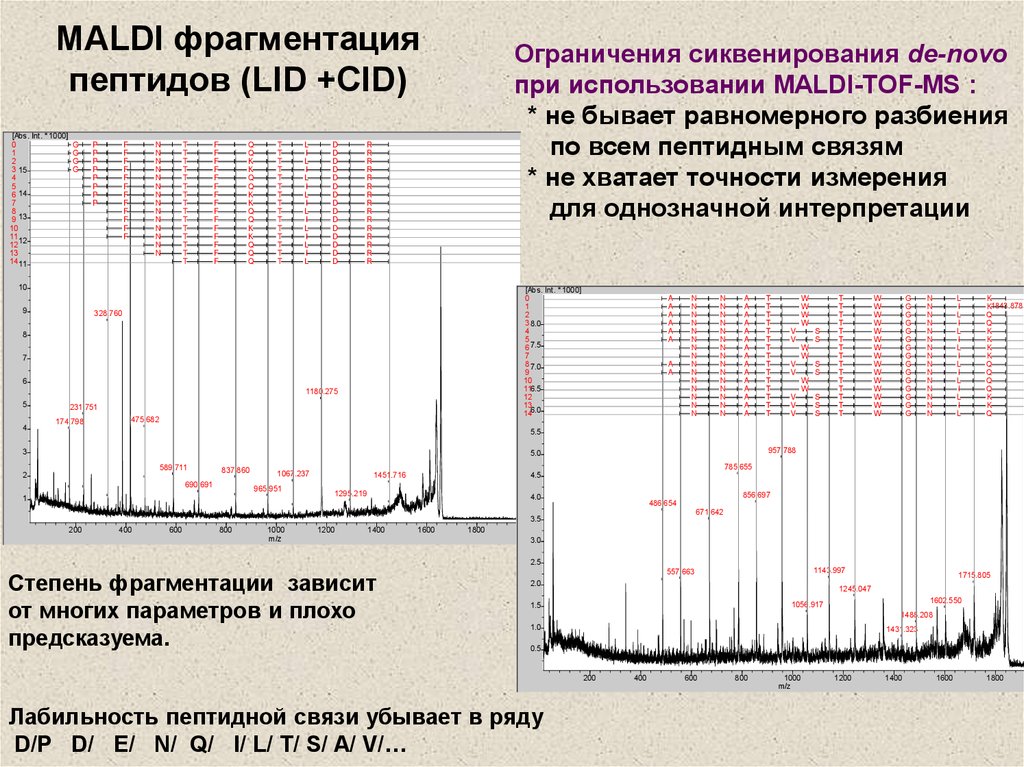
24. MALDI фрагментация пептидов (LID +CID)
[Abs. Int. * 1000]0
1
2
3 15
4
5
6 14
7
8
9 13
10
11 12
12
13
14 11
G
G
G
G
P
P
P
P
P
P
P
P
F
F
F
F
F
F
F
F
F
F
F
F
N
N
N
N
N
N
N
N
N
N
N
N
N
N
T
T
T
T
T
T
T
T
T
T
T
T
T
T
T
F
F
F
F
F
F
F
F
F
F
F
F
F
F
F
Q
Q
K
K
Q
Q
K
K
Q
Q
K
K
Q
Q
Q
T
T
T
T
T
T
T
T
T
T
T
T
T
T
T
L
I
L
I
L
I
L
I
L
I
L
I
L
I
L
D
D
D
D
D
D
D
D
D
D
D
D
D
D
D
Ограничения cиквенирования de-novo
при использовании MALDI-TOF-MS :
* не бывает равномерного разбиения
по всем пептидным связям
* не хватает точности измерения
для однозначной интерпретации
R
R
R
R
R
R
R
R
R
R
R
R
R
R
R
10
9
[Abs. Int. * 1000]
0
1
2
3 8.0
4
5
6 7.5
7
8 7.0
9
10
116.5
12
13
146.0
328.760
8
7
6
1180.275
5
4
231.751
174.798
475.682
A
A
A
A
A
A
A
A
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
A
A
A
A
A
A
A
A
A
A
A
A
A
A
A
T
T
T
T
T
T
T
T
T
T
T
T
T
T
T
W
W
W
W
V
V
S
S
W
W
V
V
S
S
W
W
V
V
V
S
S
S
T
T
T
T
T
T
T
T
T
T
T
T
T
T
T
W
W
W
W
W
W
W
W
W
W
W
W
W
W
W
G
G
G
G
G
G
G
G
G
G
G
G
G
G
G
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
L
I
L
I
L
I
L
I
L
I
L
I
L
I
L
K
K1843.878
Q
Q
K
K
K
K
Q
Q
Q
Q
K
K
Q
5.5
3
957.788
5.0
589.711
2
837.860
690.691
785.655
1067.237
965.951
1
1451.716
4.5
1295.219
856.697
4.0
486.654
671.642
3.5
200
400
600
800
1000
m/z
1200
1400
1600
1800
3.0
2.5
Степень фрагментации зависит
от многих параметров и плохо
предсказуема.
1143.997
557.663
2.0
1715.805
1245.047
1602.550
1056.917
1.5
1488.208
1.0
1431.323
0.5
200
Лабильность пептидной связи убывает в ряду
D/P D/ E/ N/ Q/ l/ L/ T/ S/ A/ V/…
400
600
800
1000
m/z
1200
1400
1600
1800
25. Определение пост-трансляционных модификаций белков
Модификации in vitro:Модификации белков после их разделения в ПААГ
Т9
Cys + I ацетамид
Т6
Cys + акриламид
Т13
Т13-14
Met окислен
Т11
цистеины алкилированы йодоацетамидом (в геле)
Модификации in vivo:
Изучение S-S мостов
Лизоцим
26. Стабильность различных модификаций белков в условиях получения масс-спектров (LID + CID)
Сахарный остаток локализованна N-концевом серине ?
Связи менее устойчивые,
чем пептидная:
фосфоэфирная
гликозидная
дисульфидная (иногда)
Такова степень фосфорилирования ?
Pi
N-ac
О-гликозилированный пептид SAPASTTQPIGSTTSTTTK
сахарный остаток галактоза (верх) либо фукоза (низ)
Фрагмент спектра трипсинолиза природного (верх)
и генно-инженерного (низ) белка
27. Стабильность различных модификаций белков в условиях получения масс-спектров (LID + CID)
Связи сопоставимые поустойчивости с пептидной:
тиоэфирная
любая амидная N-ацетилирование
дисульфидная (иногда)
Пептид, модифицированный тремя пальмитатами
(тиоэфирная связь)
Пептид N- ацетилирован, а цистеин модифицирован
янтарным ангидридом (тиоэфирная связь)
28. Сравнение подходов
при определении белкового состава сложной многокомпонентной смесиMS целых
белков
LC- ESI- 2DE -MALDI- 1DE-MALDIMS/MS
TOF- MS
TOF-MS
Определение количества одного белка в разных образцах
++
++ - +++++
+++
+
Определение количеств разных белков в одном образце
++
+
+++
+
++++
+++++
+++
+
+++
+
Идентификация сильно представленных в образце белков
Идентификация мало представленных в образце белков
Идентификация мембранных белков
Определение возможных модификаций белка
+++
+
Стратегия определения модификаций:
+++
++
Выделение белка
Гидролиз специфическими протеазами соотнесение значений масс пиков в спектре с первичной структурой.
Определение пептидов отличных по массе от рассчитанной –
гипотезы о природе модификации
Индивидуальный подход к получению структурной информации из спектров фрагментации –
выбор типа фрагментации, использование ферментов для дальнейшего расщепления
пептида, удаления модификации, химические подходы...
29. Литература:
* Говорун В.М., Арчаков А.И. Протеомные технологии в современной биомедицинской науке.Biochemistry (Mosc). 2002 Oct;67(10):1109-23.
* Lottspeich F. Proteome Analysis: A Pathway to the Functional Analysis of Proteins.
Angew Chem Int Ed Engl. 1999 Sep;38(17):2476-2492
* M.Mann, R.C.Hendrickson, A.Pandey, Analysis of proteins and proteomes by mass spectrometry,
Annu. Rev. Biochem., 70, 437-473 (2001).
* O.N.Jensen, M.Wilm, A.Shevchenko, M.Mann, Sample preparation methods for mass spectrometric peptide mapping
directly from 2-DE gels, Methods Mol. Biol., 112, 513-530 (1999).
* B.Kuster, T.N.Krogh, E.Mortz, D.J.Harvey, Glycosylation analysis of gel-separated proteins, Proteomics, 1, 350-361 (2001).
* Aebersold R, Goodlett DR Mass spectrometry in proteomics Chem Rev 2001 Feb;101(2):269-95
* Nordhoff E, Egelhofer V, Giavalisco P, Eickhoff H, Horn M, Przewieslik T, Theiss D, Schneider U, Lehrach H, Gobom J.
Large-gel two-dimensional electrophoresis-matrix assisted laser desorption/ionization-time flight-mass spectrometry:
an analytical challenge for studying complex protein mixtures. Electrophoresis. 2001 Aug;22(14):2844-55
* Roepstorff P MALDI-TOF mass spectrometry in protein chemistry.. EXS 2000;88:81-97
* Gygi SP, Aebersold R. Using mass-spectrometry for quantitative proteomics Proteomics: A Trands Guide 2000, 31-36
* Andersen JS, Mann M Functional genomics by mass spectrometry. FEBS Lett. 2000 Aug 25;480(1):25-31
* Kussmann M, Roepstorff P Sample preparation techniques for peptides and proteins analyzed by MALDI-MS.
Methods Mol Biol. 2000;146:405-24
* Chaurand P, Luetzenkirchen F, Spengler B. Peptide and protein identification by matrix-assisted laser desorption
ionization (MALDI) and MALDI-post-source decay time-of-flight mass spectrometry.
J Am Soc Mass Spectrom. 1999 Feb;10(2):91-103
* Spengler, B, Post-source decay analysis in matrix-assisted laser desorption/ionization mass spectrometry of
biomolecules. J. Mass Spectrom., 32(10) 1019-36 (1997).
* Jonscher, KR and Yates, JR, 3rd, The quadrupole ion trap mass spectrometer--a small solution to a big challenge.
Anal Biochem, 244(1) 1-15 (1997).
* Qin, J and Chait, BT, Identification and characterization of posttranslational modifications of proteins by MALDI
ion trap mass spectrometry. Anal Chem, 69(19) 4002-9 (1997).
* Papayannopoulos, IA, The interpretation of collision-induced dissociation tandem mass spectra of peptides.
Mass Spectrom. Rev., 14(1) 49-73 (1995).
* Hillenkamp, F, Karas, M, Beavis, RC and Chait, BT, Matrix-assisted laser desorption/ionization mass spectrometry
of biopolymers. Anal. Chem., 63(24) 1193A-203A (1991).
* Fenn, JB, Mann, M, Meng, CK, Wong, SF and Whitehouse, CM, Electrospray ionization-principles and practice.
Mass Spectrom. Rev., 9(1) 37-70 (1990)


























 physics
physics








