Similar presentations:
Разработка программного обеспечения для анализа масс-спектрометрической информации
1.
Разработка программногообеспечения для анализа
масс-спектрометрической
информации
Авторы:
Трофименков Иван Васильевич,
Махнач Никита Владимирович, студенты 3 курса физического факультета
Научные руководители:
-Коваленко Антон Николаевич,
старший научный сотрудник НИИ ЯП БГУ
-Коваленко Елена Иосифовна,
доцент физического факультета БГУ
кандидат биологических наук, доцент
МИНСК,2021
2.
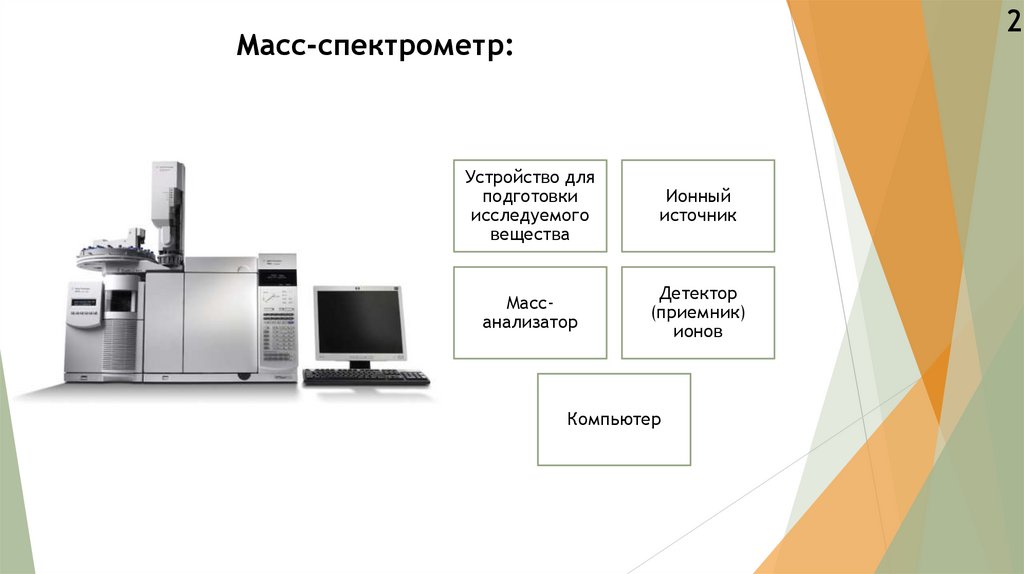
2Масс-спектрометр:
Устройство для
подготовки
исследуемого
вещества
Ионный
источник
Массанализатор
Детектор
(приемник)
ионов
Компьютер
3.
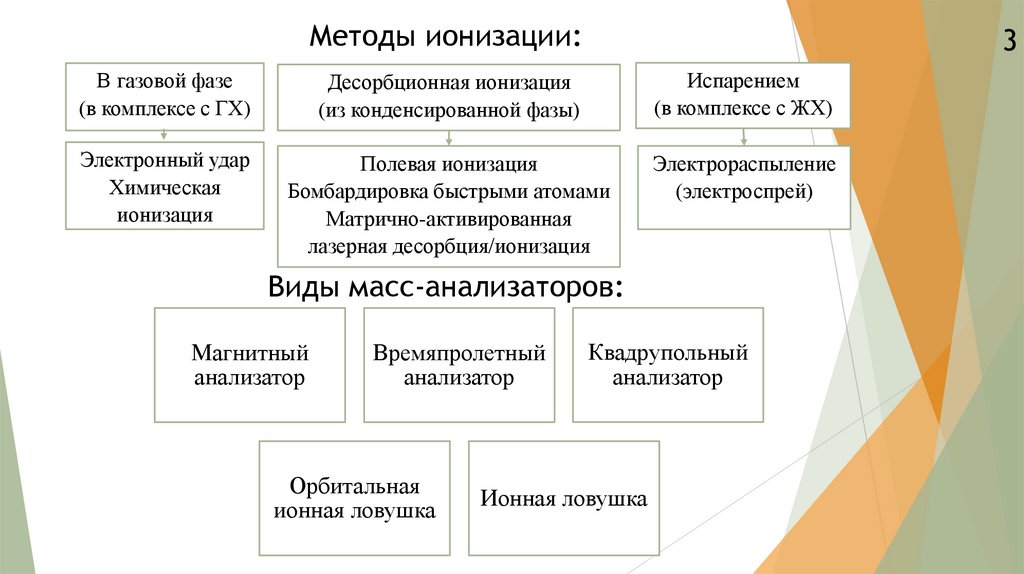
Методы ионизации:3
В газовой фазе
(в комплексе с ГХ)
Десорбционная ионизация
(из конденсированной фазы)
Испарением
(в комплексе с ЖХ)
Электронный удар
Химическая
ионизация
Полевая ионизация
Бомбардировка быстрыми атомами
Матрично-активированная
лазерная десорбция/ионизация
Электрораспыление
(электроспрей)
Виды масс-анализаторов:
Магнитный
анализатор
Времяпролетный
анализатор
Орбитальная
ионная ловушка
Квадрупольный
анализатор
Ионная ловушка
4.
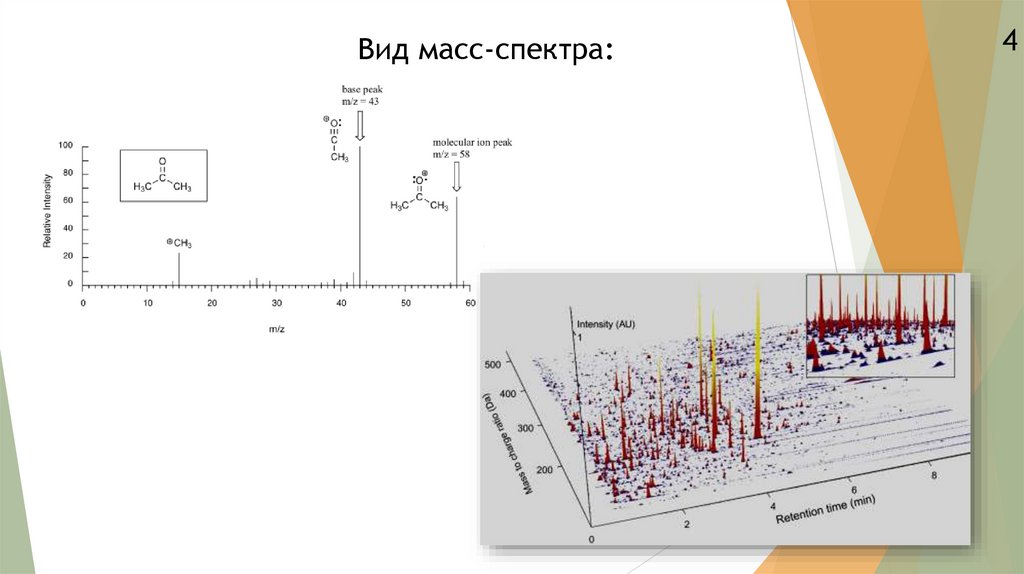
Вид масс-спектра:4
5.
Цель работы:1) Разработать универсальный кросс-платформенный программный
интерфейс к базам данных для масс-спектрометрической идентификации.
2) Обеспечить совместимость с разработанным интерфейсом систему
регистрации хранения и обработки спектрометрической информации
UniChrom-97.
3) Реализовать данный интерфейс в заменяемом программном модуле
(DLL) для использования базы данных NIST MS 2020
4) Реализовать данный интерфейс в заменяемом программном модуле
(DLL) для использования SQL-ориентированной базы данных.
5
6.
Общий алгоритм поиска неизвестноговещества в базе данных (БД):
1. Получить масс-спектр (скан) в указанной
временной точке.
2. Вычитание шума
3. Создать контекст поиска.
4. Передать спектр в выбранный контекст в модуле
поиска
5. Вызвать метод поиска кандидатов
6
7.
76. Выполнить подготовку БД.
7. Предварительный поиск кандидатов в БД.
8. Полный поиск кандидатов в БД.
9. Сопоставить масс-спектры кандидатов с
информацией по веществам.
10.Список масс-спектров.
11.Возвратить код выхода
12.Представление в табличном виде
8.
Реализация модуля поиска в базахданных при помощи NIST :
Программное
обеспечение
Версия Назначение
NIST Lib
2020
Библиотеки масс-спектров и структуры NIST2020
Free Pascal
Compiler
3.2
Кросс-платформный компилятор ObjectPascal
совместимый с Delphi Pascal
Lazarus IDE
2.0
Интегрированная среда разработки для Free
Pascal Compiler (FPC), написана на FPC
8
9.
Этапы поиска в системе NISTПодготовка БД: обеспечивается вызовом функции
nistms_search
Передача путей: mainlib, replib, userlib
Предварительный (скрининговый) поиск.
Поиск кандидатов: sim_num, rev_sim_num, hit_prob.
Загрузка масс-спектров для каждого из кандидатов:
TSpDesc1, get_spectrum
9
10.
Реализация модуля поиска в базахданных при помощи SQL :
Программное
обеспечение
Версия
Назначение
Firebird
3.0
SQL сервер
Free Pascal Compiler 3.2
Кросс-платформный компилятор ObjectPascal
совместимый с Delphi Pascal
Lazarus IDE
2.0
Интегрированная среда разработки для Free Pascal
Compiler (FPC), написана на FPC
UniChrom
5.1
Программа для визуализации масс-спектра и работы
с ним
10
11.
Виды таблиц:Таблица БД
Назначение
COMP
Информация о компоненте (веществе). Записи уникальны, так
как даже для одинаковой брутто-формулы (FORMULA)
структура вещества может быть различна.
MSLIB
Информация о спектре. Для одного вещества может быть
несколько разных спектров, полученных при разных
условиях. Содержит ссылку на вещество COMP_ID в таблице
COMP
SPECTRA
Пики всех спектров. Содержит ссылку SP_ID на спектр в
таблице MSLIB
SYNON
Все наименования вещества. Содержит ссылку COMP_ID на
таблицу MSLIB
SPECTRA_TMP
Пики неизвестного спектра. Идентична по структуре таблице
SPECTRA.
11
12.
Выполнение поиска:Первая этап поиска:
with sptop as (
select distinct sp.sp_id from spectra sp inner join
(select first 5 skip 0 mz, intens from spectra_tmp order by intens desc)
xx
on trunc(sp.MZ*100)=trunc(xx.mz*100) and abs(sp.INTENS-xx.intens)<10)
Критерий предварительной фильтрации определяется как:
(select first 5 skip 0 mz, intens from spectra_tmp order by intens desc) xx
Осуществляется выборка из библиотеки SPECTRA spl только пиков,
принадлежащих спектрам фильтрованным запросом предварительной
выборки sptop
select ....
from spectra spl
inner join sptop on sptop.sp_id = spl.SP_ID
12
13.
Расчёт агрегатных значений:count cnt
Количество пиков в группе
-- Stein
Сумма корня из произведений интенсивности
sum(spl.mz*sqrt(spl.INTENS*spu.INTEN библиотечного и неизвестного пиков с фактором mz
S)) xnom,
Сумма интенсивностей библиотечных пиков с
sum(spl.MZ*spl.INTENS) ilib,
фактором mz
sum(spu.MZ*spu.INTENS) iunk,
Сумма интенсивностей неизвестного спектра с
фактором mz
-- Pearson
sum(spl.INTENS) mx,
sum(spl.INTENS*spl.INTENS) mx2,
sum(spu.INTENS) my,
sum(spu.INTENS*spu.INTENS) my2,
sum(spl.INTENS*spu.INTENS) mxy,
Сумма интенсивностей библиотечного mx и
неизвестного my,
Сумма квадратов интенсивности mx2, my2
И сумма кросс-коэффициента mxy
sum(power(spl.intens-spu.intens,2))
diff2
Сумма квадратов разности интенсивностей
библиотечного и неизвестного пика совпадающих по
m/z
13
14.
После расчёта агрегатных значений по группе пиков спектравыполняется финальное вычисление скалярных величин,
характеризующих подобие библиотечного и неизвестного спектра.
select
x.*,
xnom/sqrt(ilib*iunk) st,
(mxy/(sqrt(mx2)*sqrt(my2))) sc,
(cnt*mxy - mx *my) / sqrt((cnt*mx2 - mx*mx)*(cnt*my2 - my*my)) pearson
from x
st - Коэффициент подобия по S.Stein
sc - Косинус угла спектральной контрастности (скалярное произведение
векторов)
pearson - Коэффициент корреляции спектров Пирсона.
14
15.
Сопоставление масс-спектров полученныхкандидатов с информацией о веществах:
Массивы
Идентификатор спектра
c.formula
Брутто-формула из таблицы COMP
ss.sname
Имя компонента из таблица SYNON
c.mw
Молекулярная масса из таблицы COMP
mss.st
Коэффициент подобия по S.Stein
mss.sc
Коэффициент спектральной контрастности (косинус угла
между спектральными векторами)
mss.pearson Коэффициент корреляции Пирсона между неизвестным и
библиотечным спектром.
15
16.
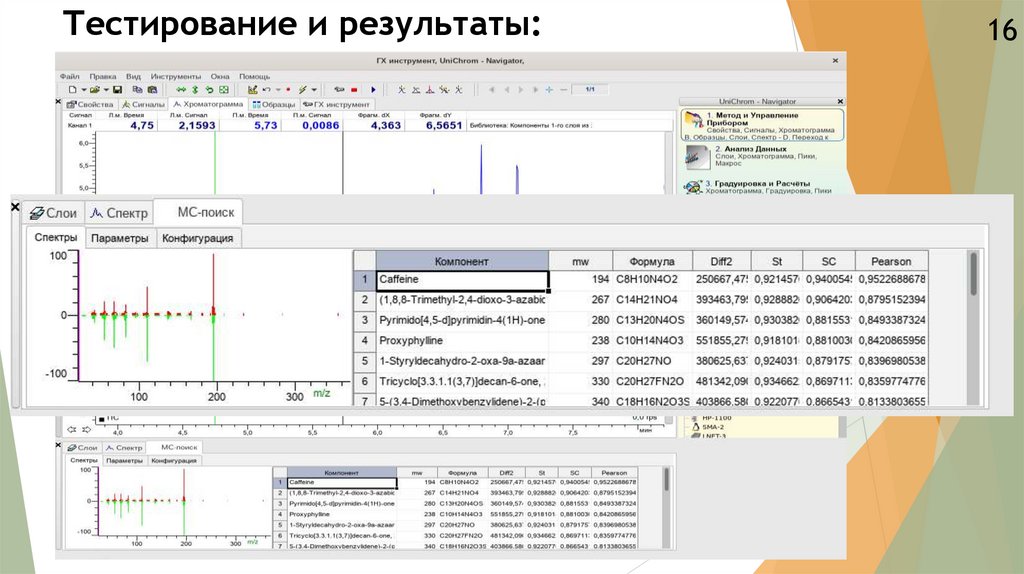
Тестирование и результаты:16
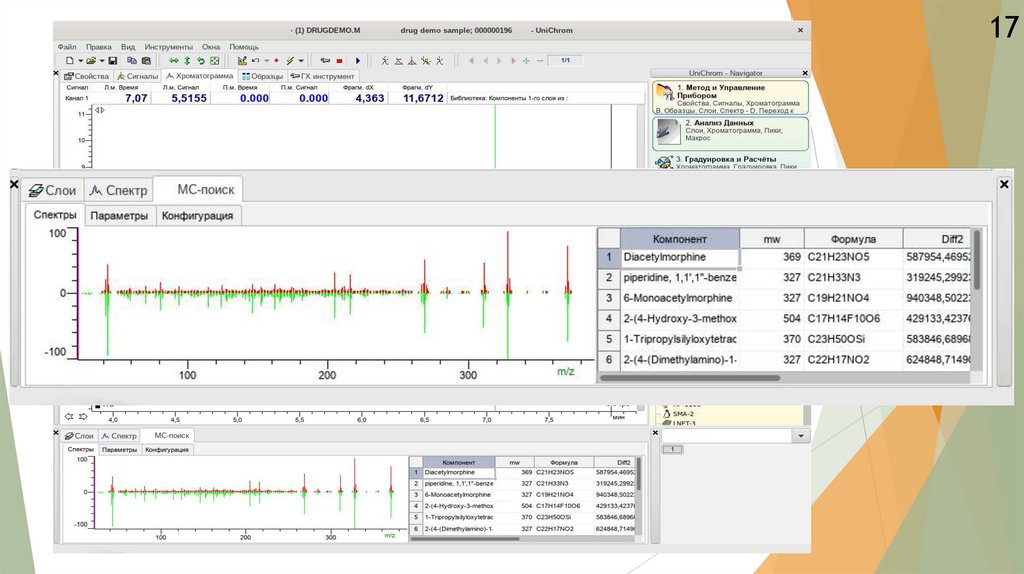
17.
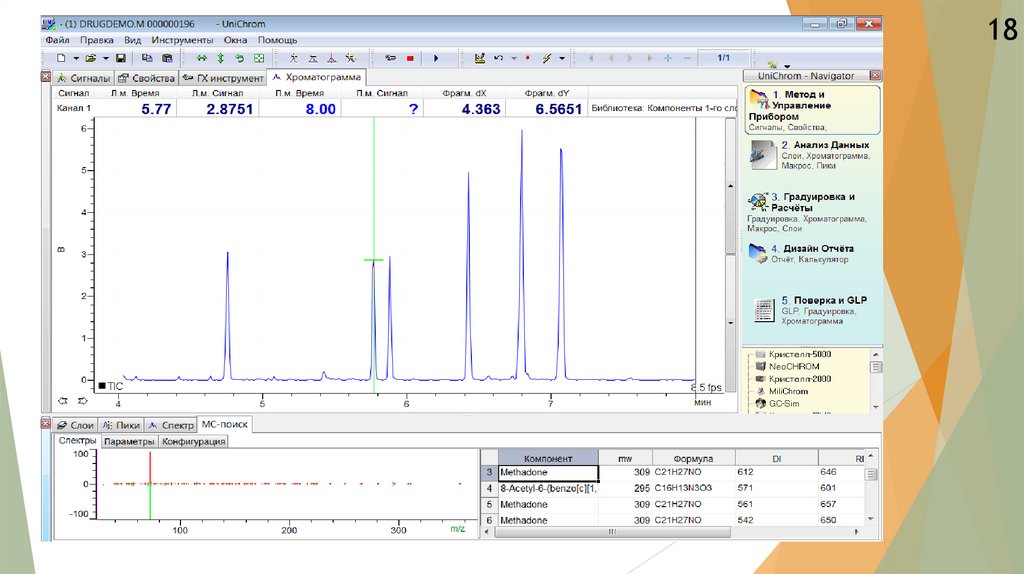
1718.
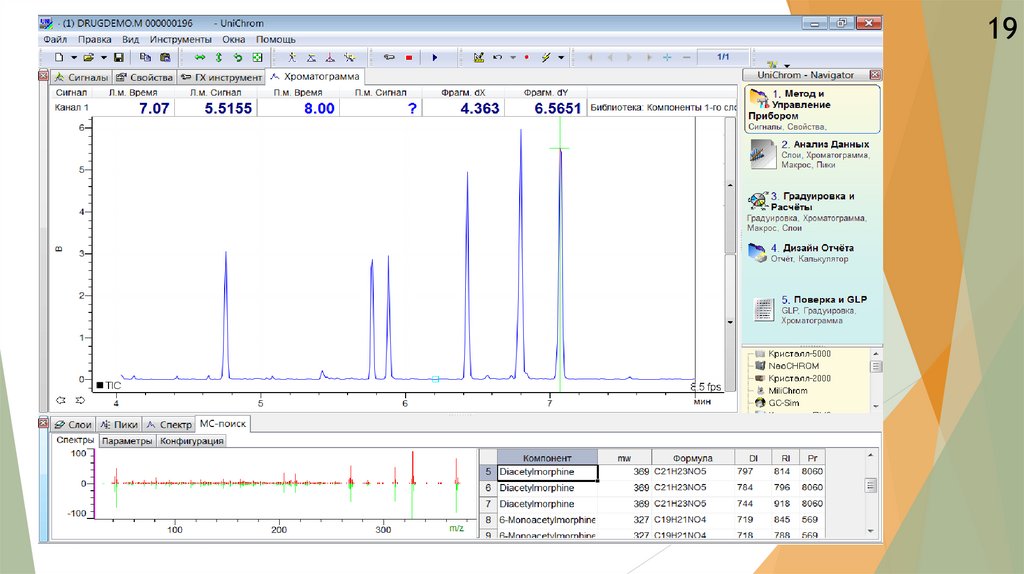
1819.
1920.
20Заключение
Разработан универсальный кросс-платформенный программный
интерфейс к базам данных для масс-спектрометрической
идентификации и протестирован с UniChrome-97 под ОС Linux
Debian GNU/Linux 10.9 и Windows 7/Windows 10 64bit.
Модуль поиска может быть использован с другими программными
продуктами и другими базами данных (в том числе с Webинтерфейсом).














 programming
programming software
software








