Similar presentations:
Основы молекулярной биологии. Структура и взаимодействие макромолекул
1.
Московский государственный университет имени М.В. ЛомоносоваОсновы молекулярной биологии
Структура и взаимодействие
макромолекул
2.
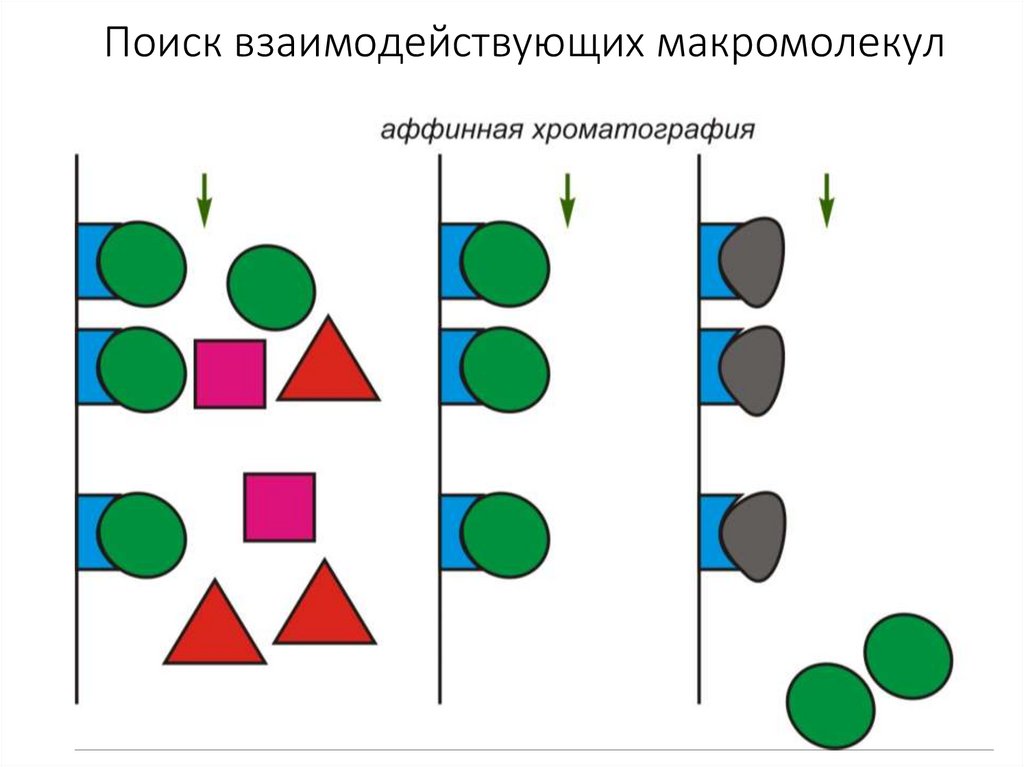
Поиск взаимодействующих макромолекул2
3.
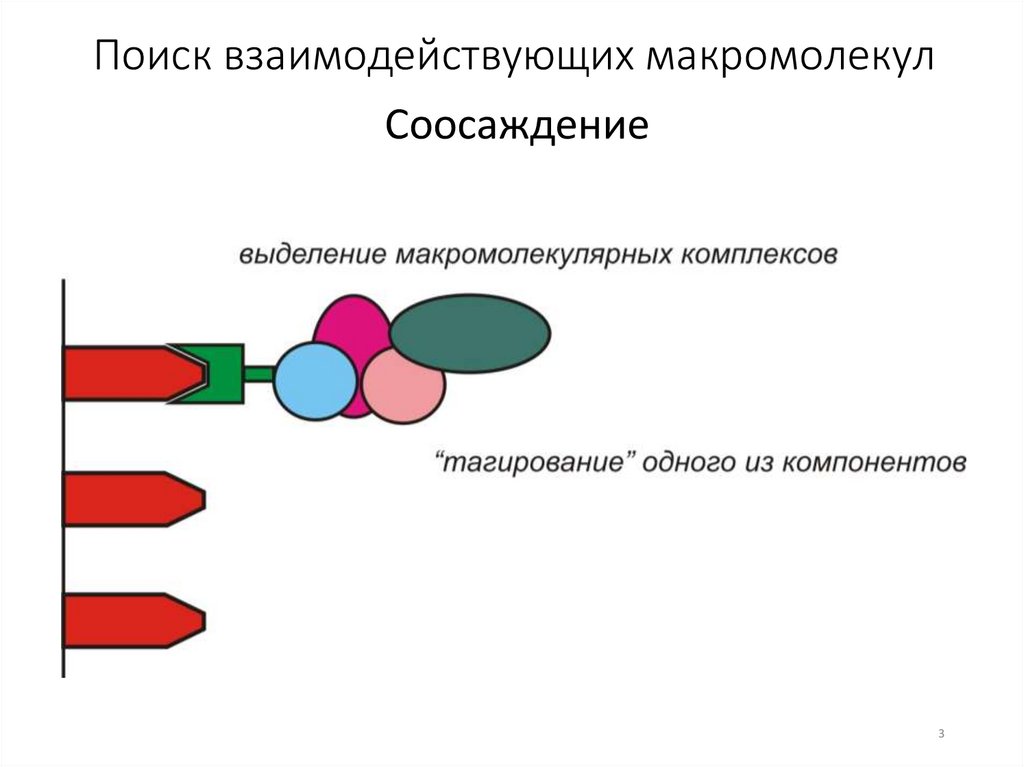
Поиск взаимодействующих макромолекулСоосаждение
3
4.
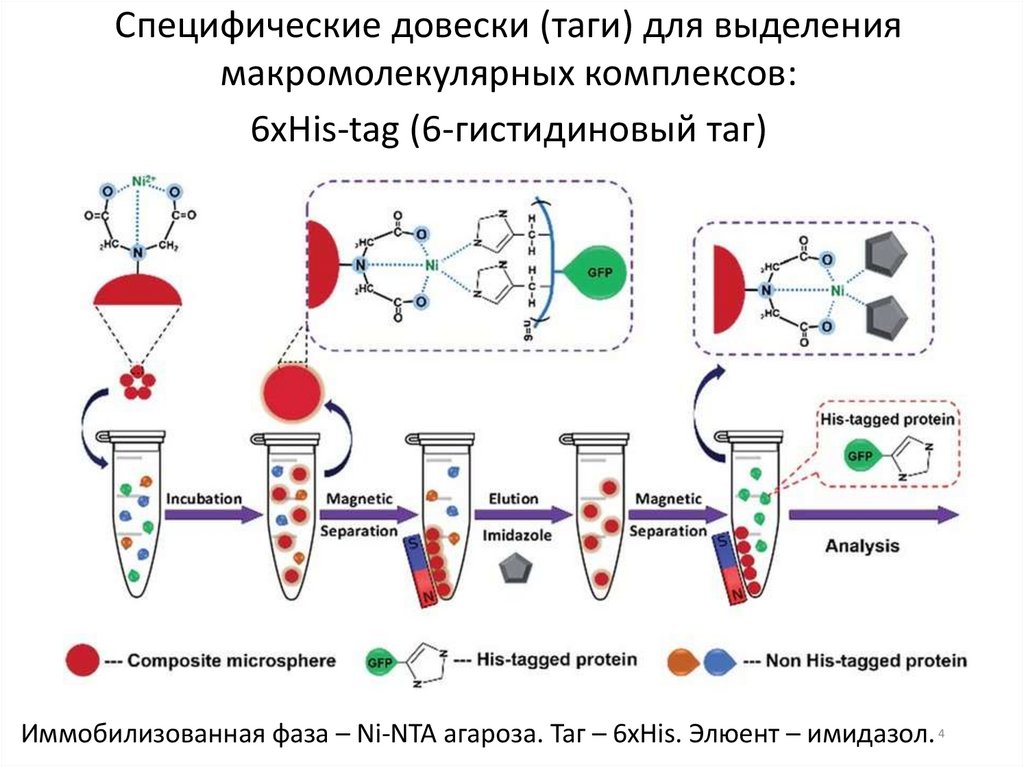
Специфические довески (таги) для выделениямакромолекулярных комплексов:
6xHis-tag (6-гистидиновый таг)
Иммобилизованная фаза – Ni-NTA агароза. Таг – 6хHis. Элюент – имидазол. 4
5.
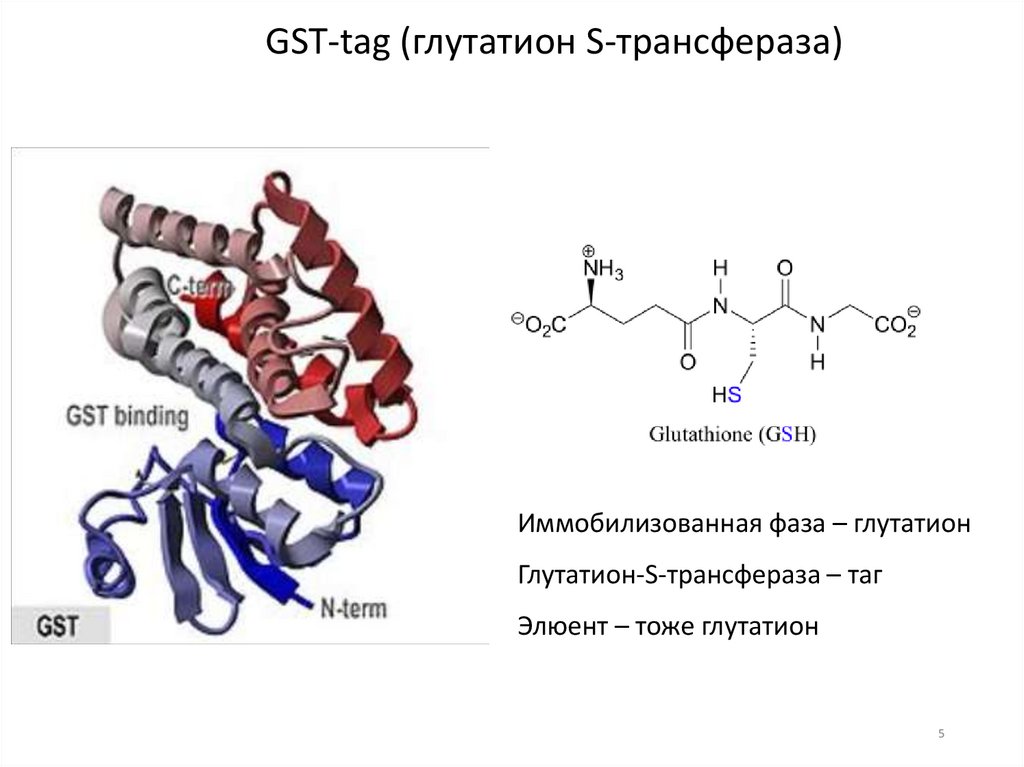
GST-tag (глутатион S-трансфераза)Иммобилизованная фаза – глутатион
Глутатион-S-трансфераза – таг
Элюент – тоже глутатион
5
6.
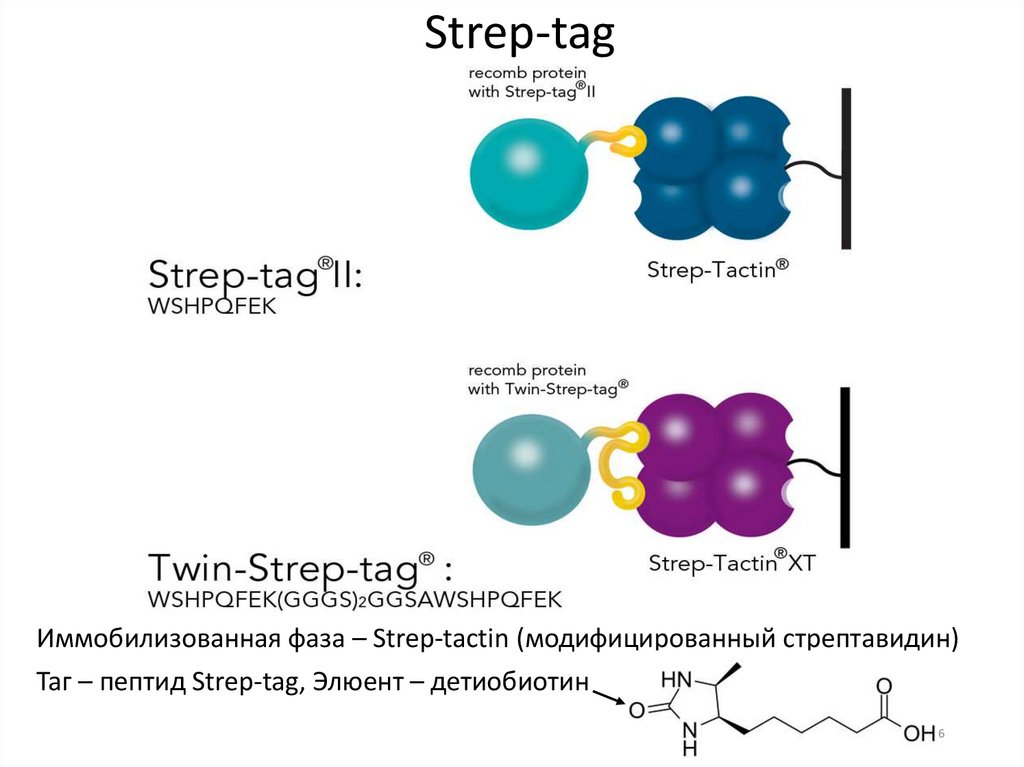
Strep-tagИммобилизованная фаза – Strep-tactin (модифицированный стрептавидин)
Таг – пептид Strep-tag, Элюент – детиобиотин
6
7.
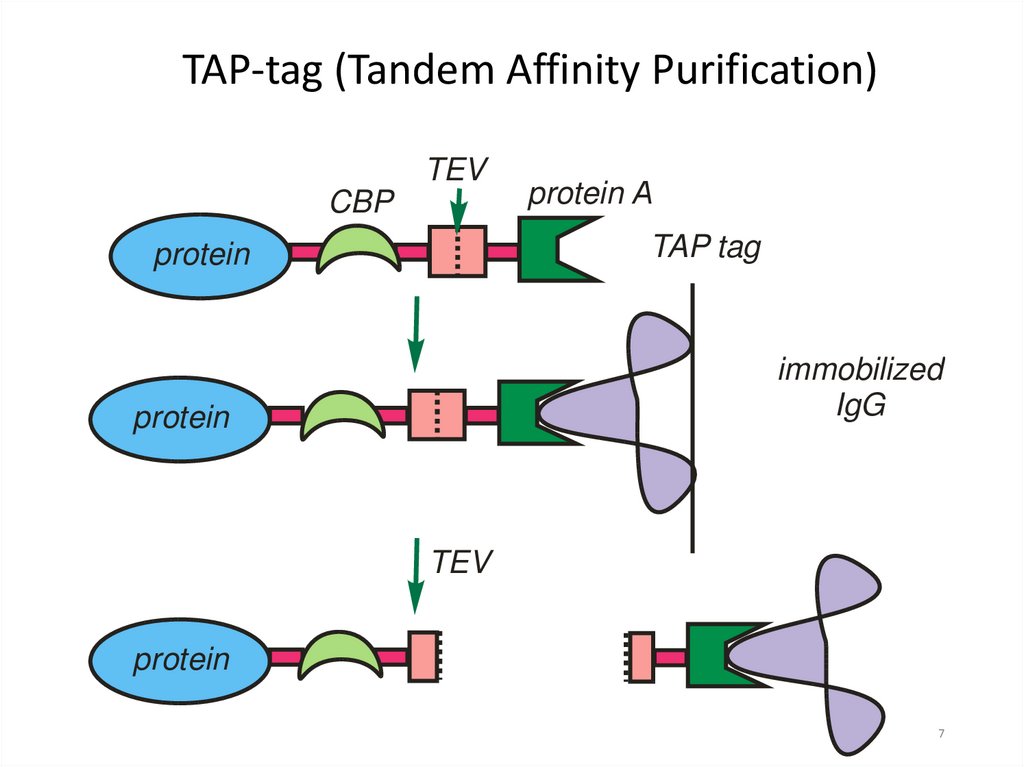
TAP-tag (Tandem Affinity Purification)TEV
CBP
protein А
TAP tag
protein
immobilized
IgG
protein
TEV
protein
7
8.
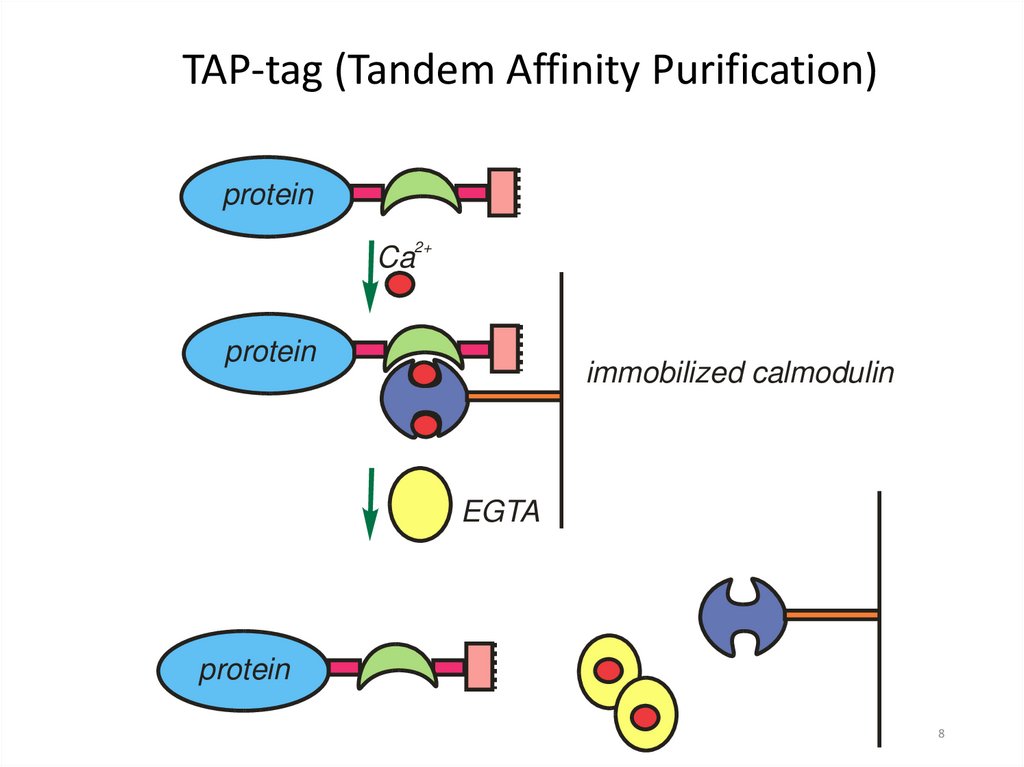
TAP-tag (Tandem Affinity Purification)protein
2+
Ca
protein
immobilized calmodulin
EGTA
protein
8
9.
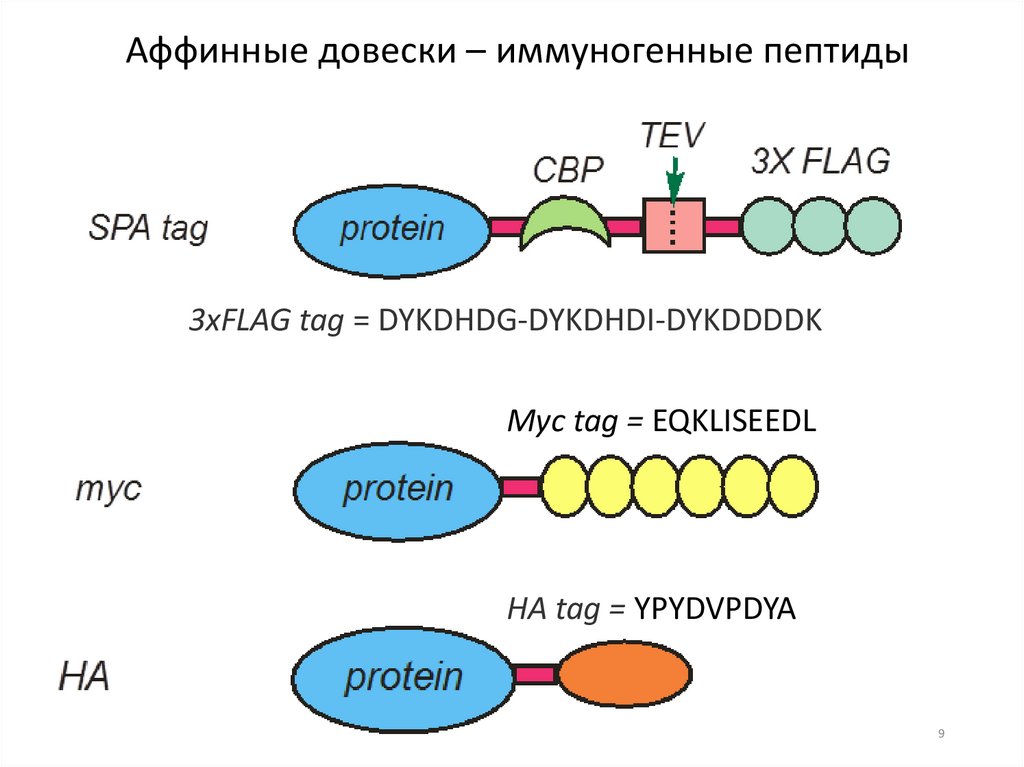
Аффинные довески – иммуногенные пептиды3хFLAG tag = DYKDHDG-DYKDHDI-DYKDDDDK
Myc tag = EQKLISEEDL
HA tag = YPYDVPDYA
9
10.
MBP-tag (мальтоза-связывающий белок)11.
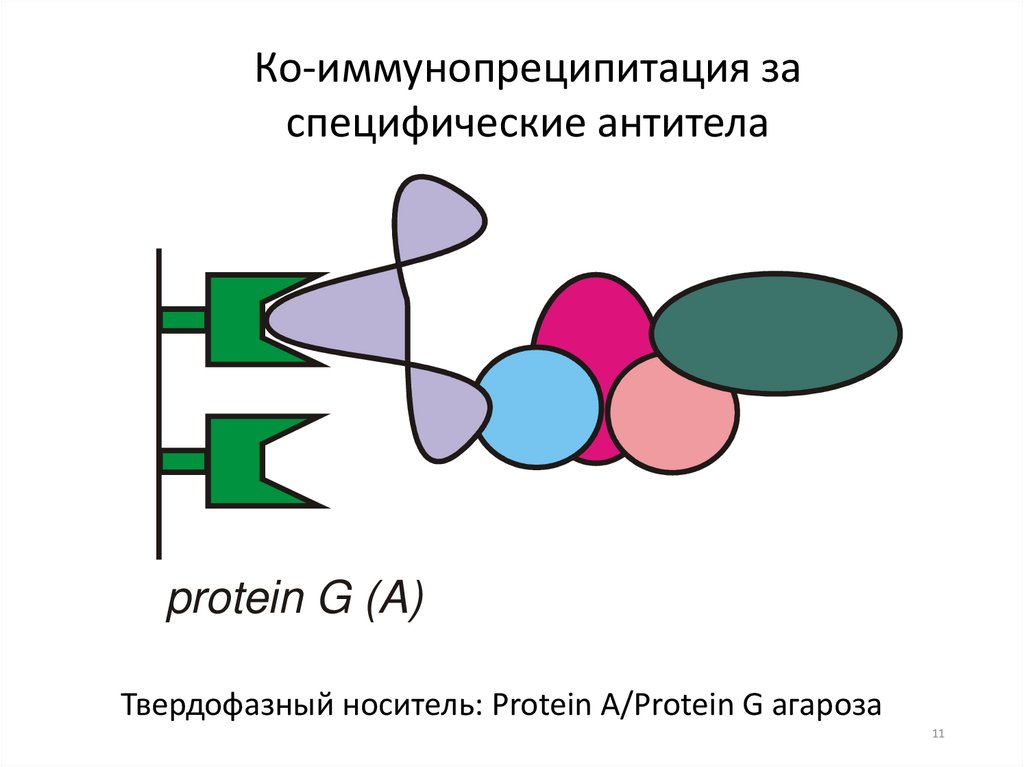
Ко-иммунопреципитация заспецифические антитела
protein G (A)
Твердофазный носитель: Protein A/Protein G агароза
11
12.
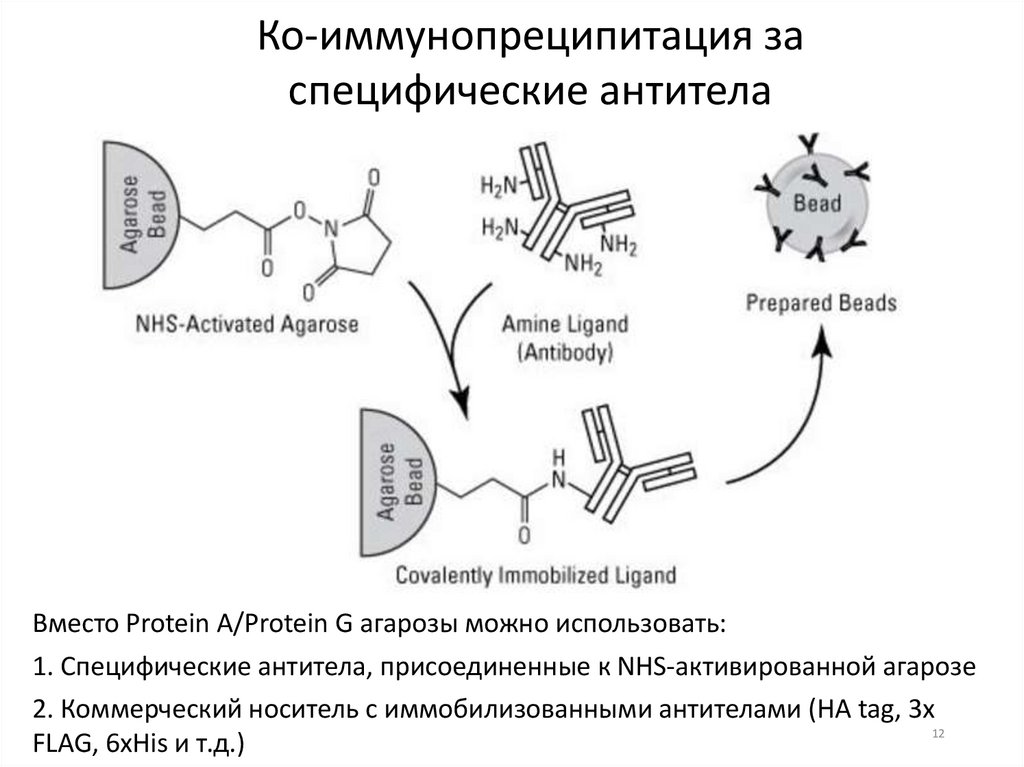
Ко-иммунопреципитация заспецифические антитела
Вместо Protein A/Protein G агарозы можно использовать:
1. Специфические антитела, присоединенные к NHS-активированной агарозе
2. Коммерческий носитель с иммобилизованными антителами (HA tag, 3x
12
FLAG, 6xHis и т.д.)
13.
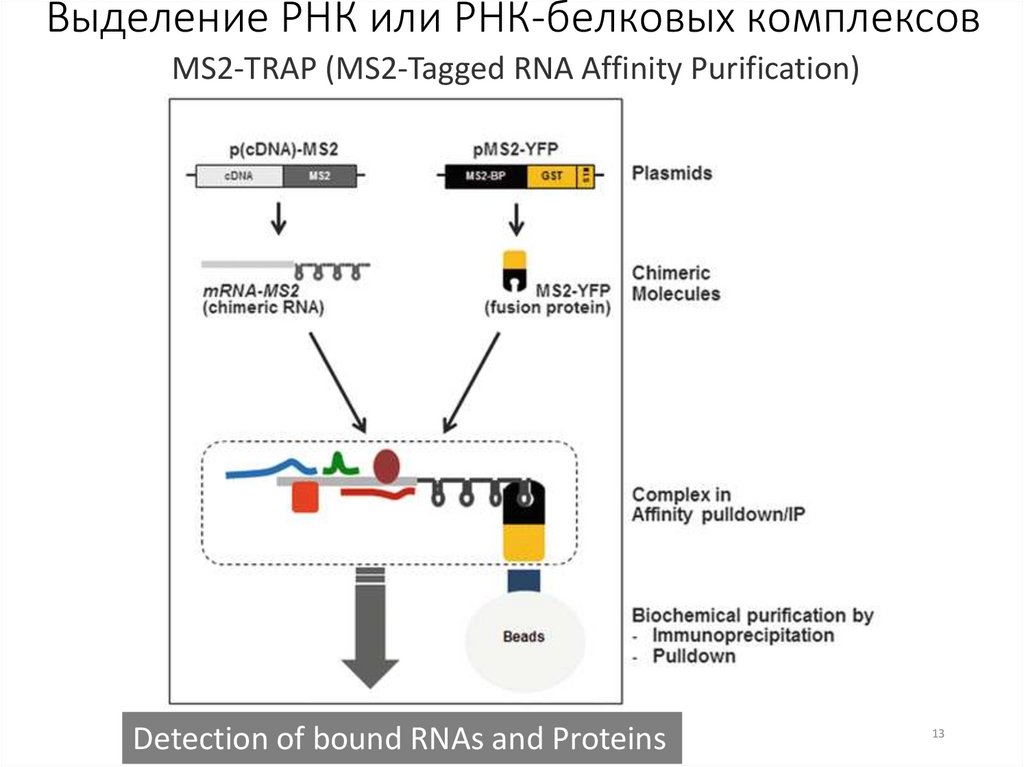
Выделение РНК или РНК-белковых комплексовMS2-TRAP (MS2-Tagged RNA Affinity Purification)
Detection of bound RNAs and Proteins
13
14.
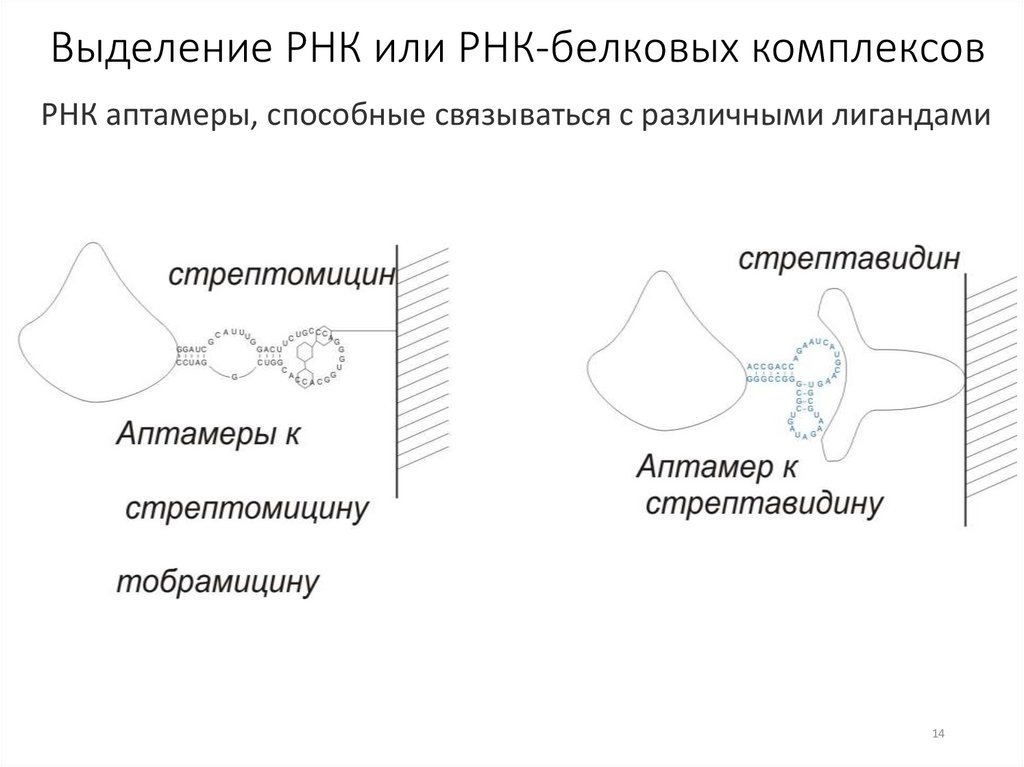
Выделение РНК или РНК-белковых комплексовРНК аптамеры, способные связываться с различными лигандами
14
15.
Как детектировать соосажденные белки и РНК?1. Если вы предполагаете, какие белки могут образовывать
комплекс:
А). Гель-электрофорез смеси белков по Лэммли
Б). Иммуноблоттинг
В). Детекция с помощью нескольких антител
2. Если вы не знаете, какие белки образуют комплекс с вашим:
А). Гель электрофорез смеси белков по Леммли или 2D
Б). Вырезаем отдельные полосы
В). Трипсинолиз белков прямо в геле
Г). Масс-спектрометрический анализ пептидов и детекция белков
3. РНК подвергают обратной транскрипции, а затем проводят
количественную ПЦР (qPCR) или секвенирование РНК (RNA-seq)
15
16.
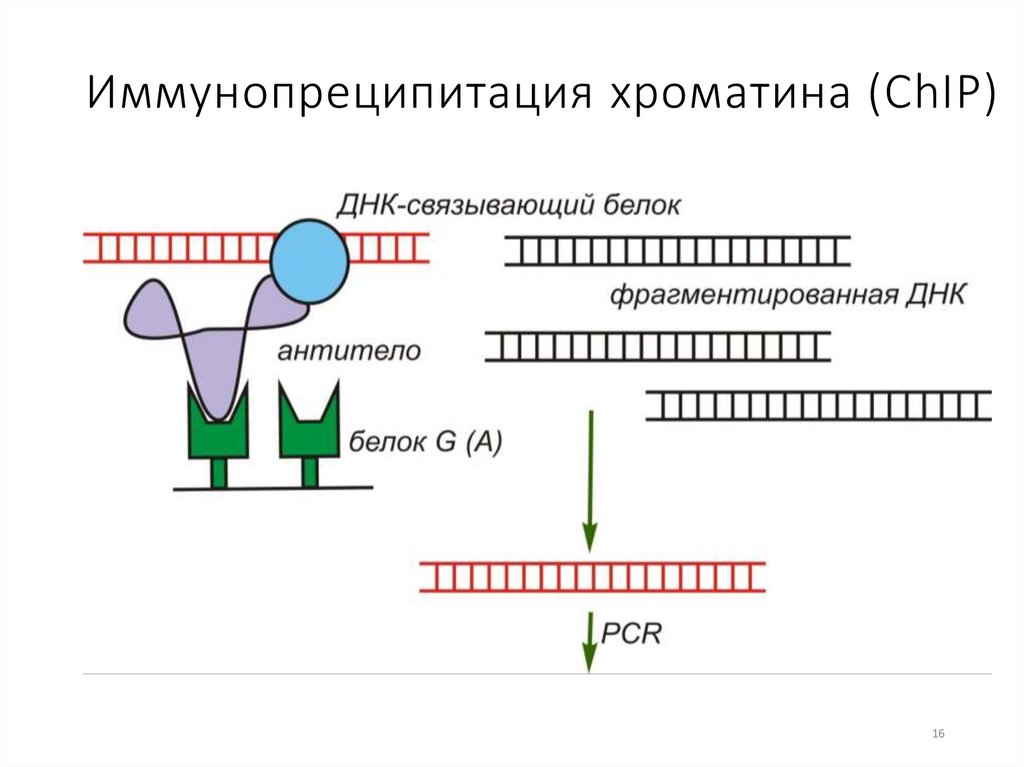
Иммунопреципитация хроматина (ChIP)16
17.
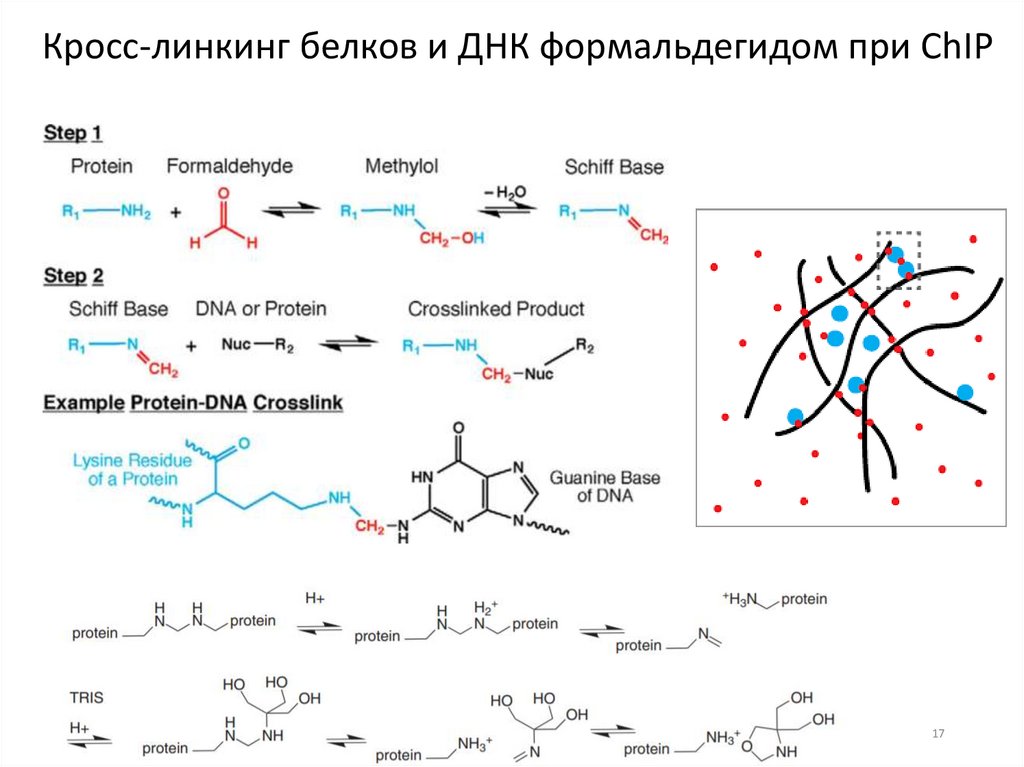
Кросс-линкинг белков и ДНК формальдегидом при ChIP17
18.
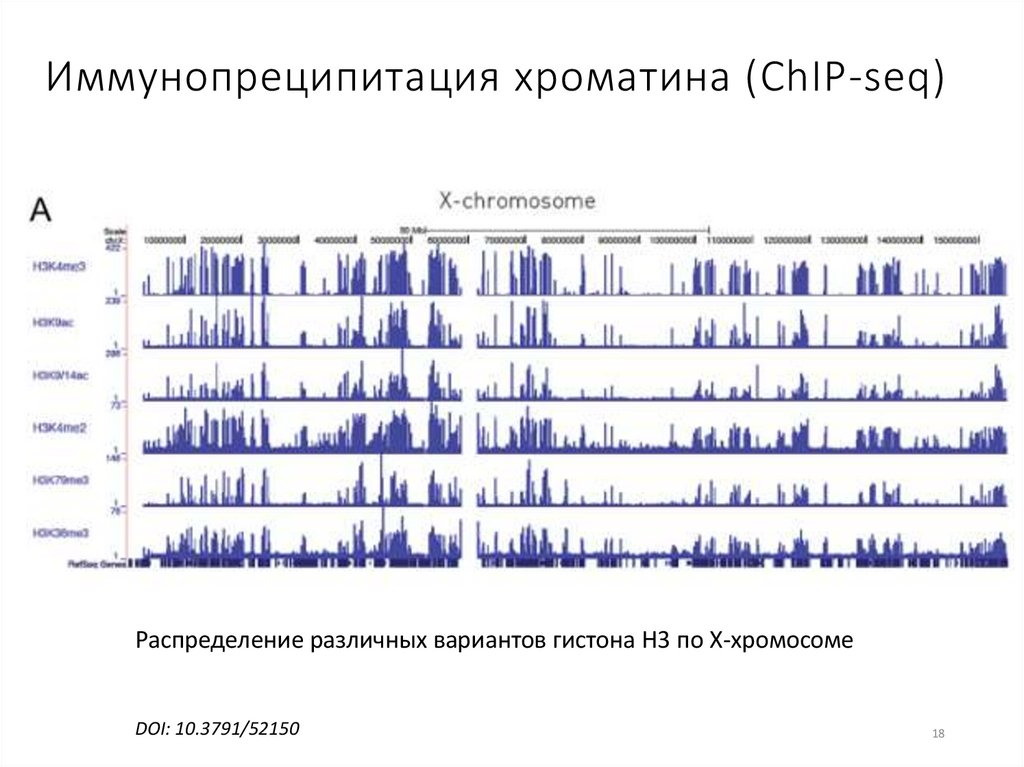
Иммунопреципитация хроматина (ChIP-seq)Распределение различных вариантов гистона H3 по Х-хромосоме
DOI: 10.3791/52150
18
19.
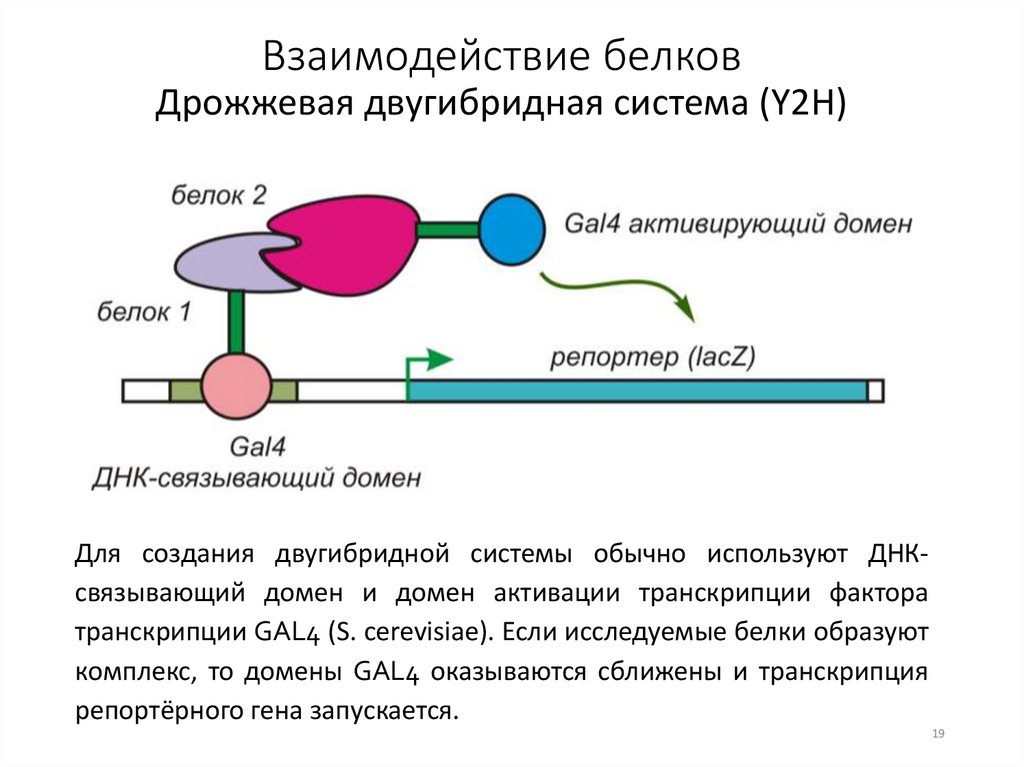
Взаимодействие белковДрожжевая двугибридная система (Y2H)
Для создания двугибридной системы обычно используют ДНКсвязывающий домен и домен активации транскрипции фактора
транскрипции GAL4 (S. cerevisiae). Если исследуемые белки образуют
комплекс, то домены GAL4 оказываются сближены и транскрипция
репортёрного гена запускается.
19
20.
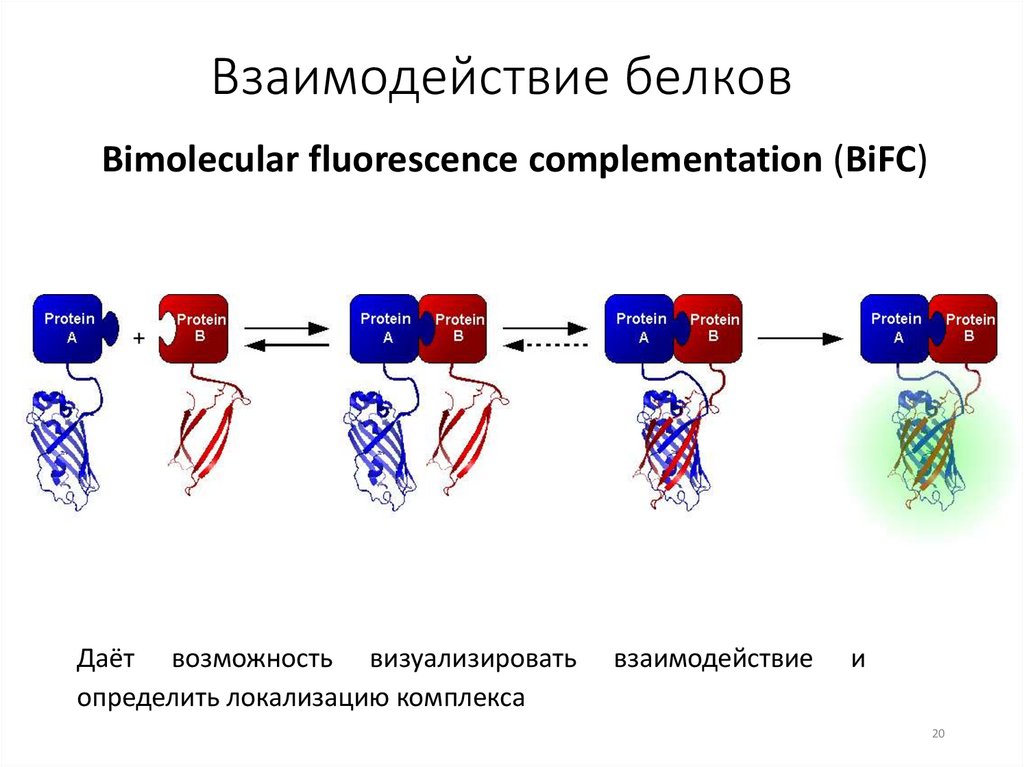
Взаимодействие белковBimolecular fluorescence complementation (BiFC)
Даёт возможность визуализировать
определить локализацию комплекса
взаимодействие
и
20
21.
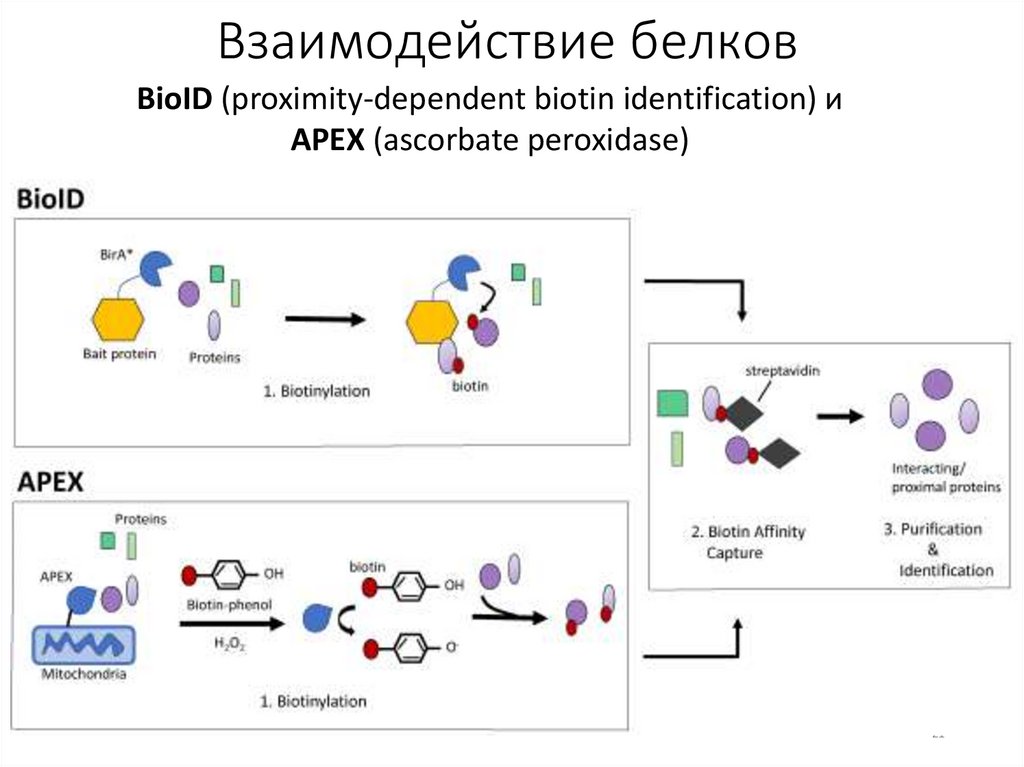
Взаимодействие белковBioID (proximity-dependent biotin identification) и
APEX (ascorbate peroxidase)
21
22.
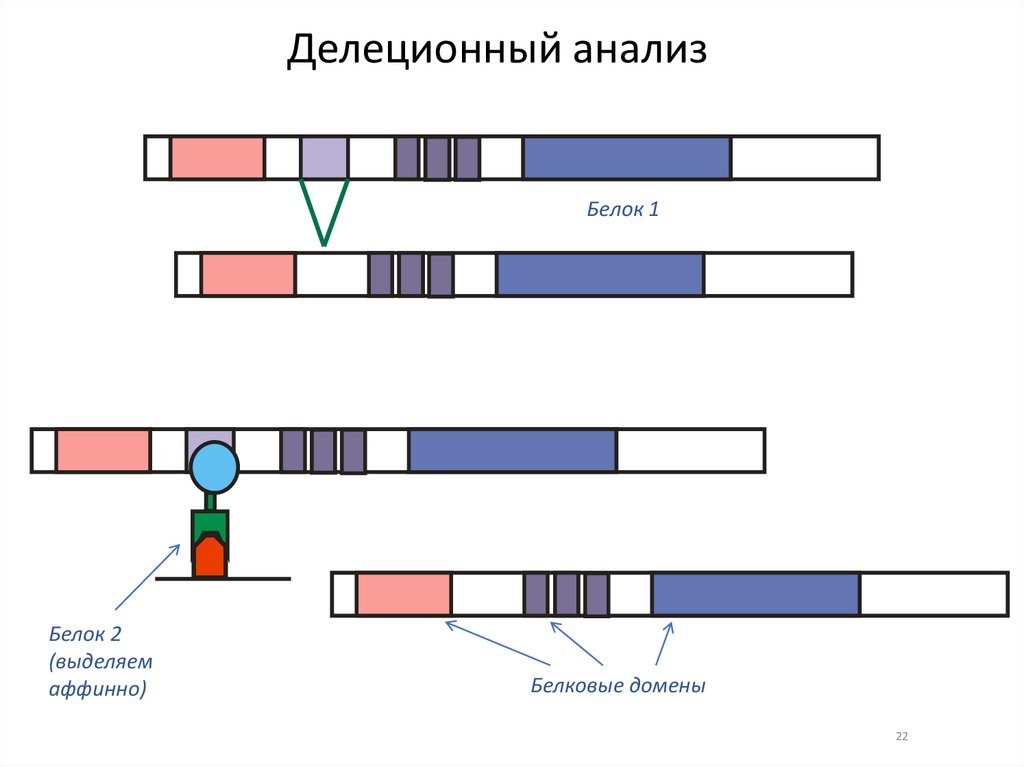
Делеционный анализБелок 1
Белок 2
(выделяем
аффинно)
Белковые домены
22
23.
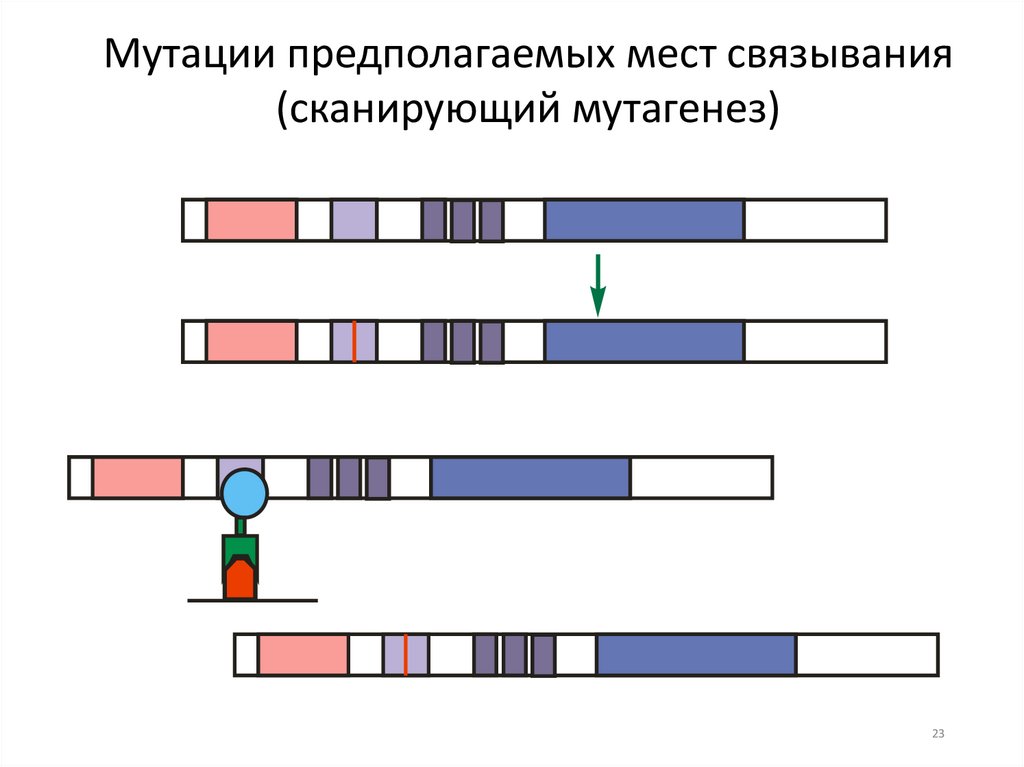
Мутации предполагаемых мест связывания(сканирующий мутагенез)
23
24.
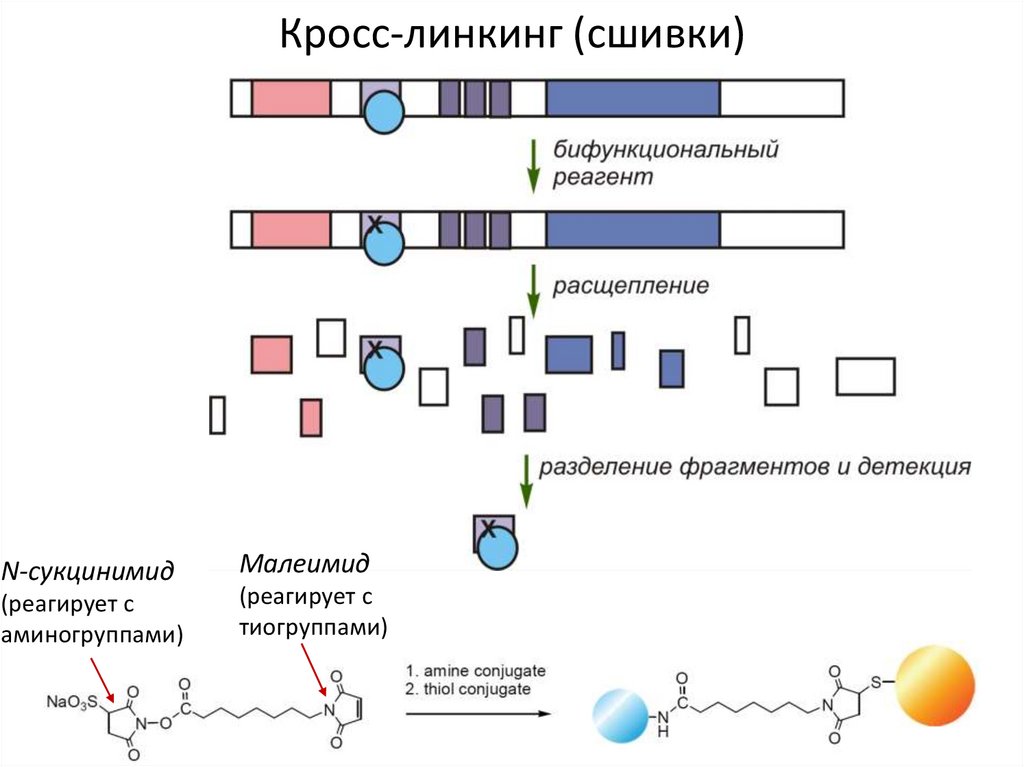
Кросс-линкинг (сшивки)N-сукцинимид
(реагирует с
аминогруппами)
Малеимид
(реагирует с
тиогруппами)
24
25.
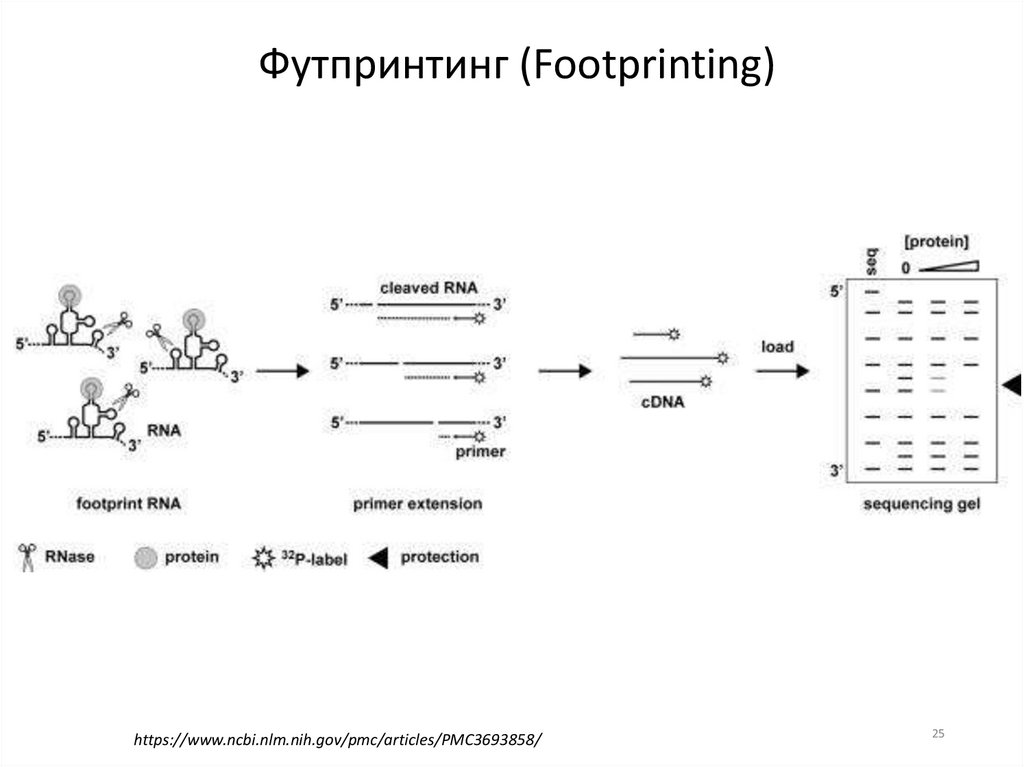
Футпринтинг (Footprinting)https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3693858/
25
26.
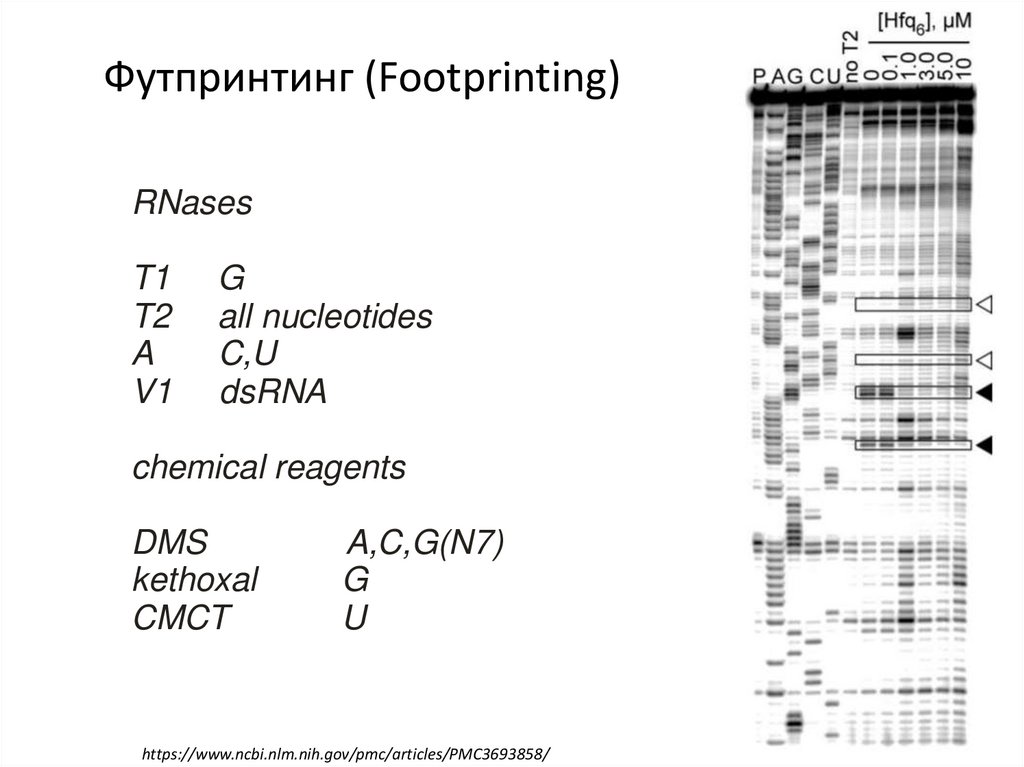
Футпринтинг (Footprinting)RNases
T1
T2
A
V1
G
all nucleotides
C,U
dsRNA
chemical reagents
DMS
kethoxal
CMCT
A,C,G(N7)
G
U
26
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3693858/
27.
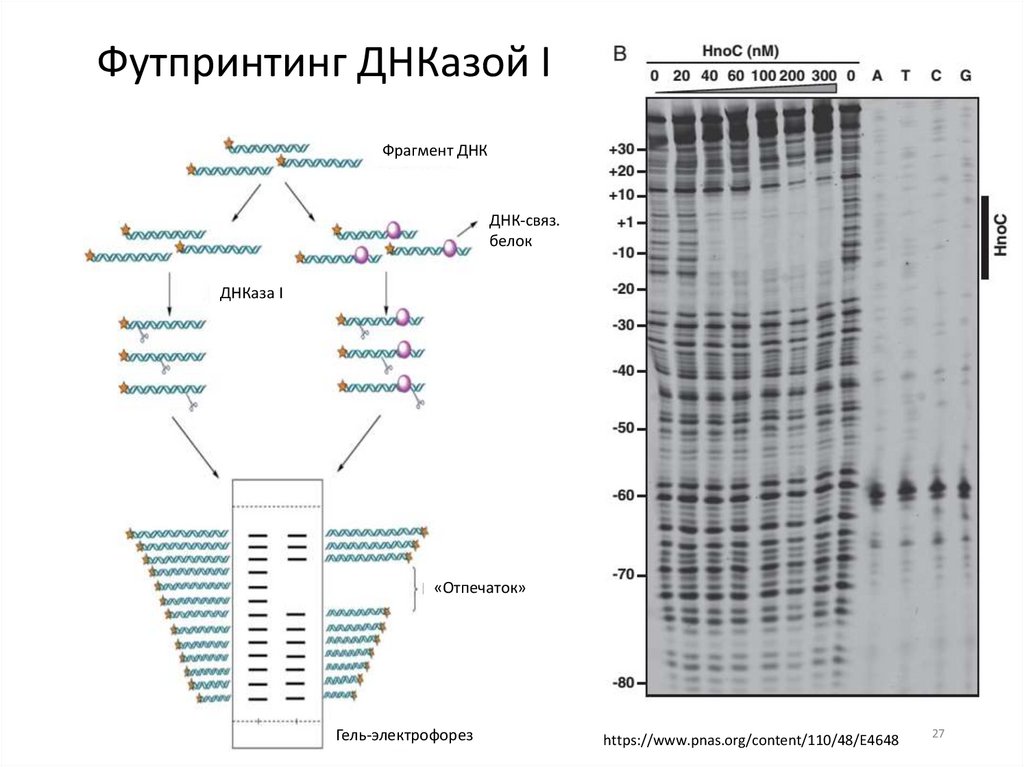
Футпринтинг ДНКазой IФрагмент ДНК
ДНК-связ.
белок
ДНКаза I
«Отпечаток»
Гель-электрофорез
https://www.pnas.org/content/110/48/E4648
27
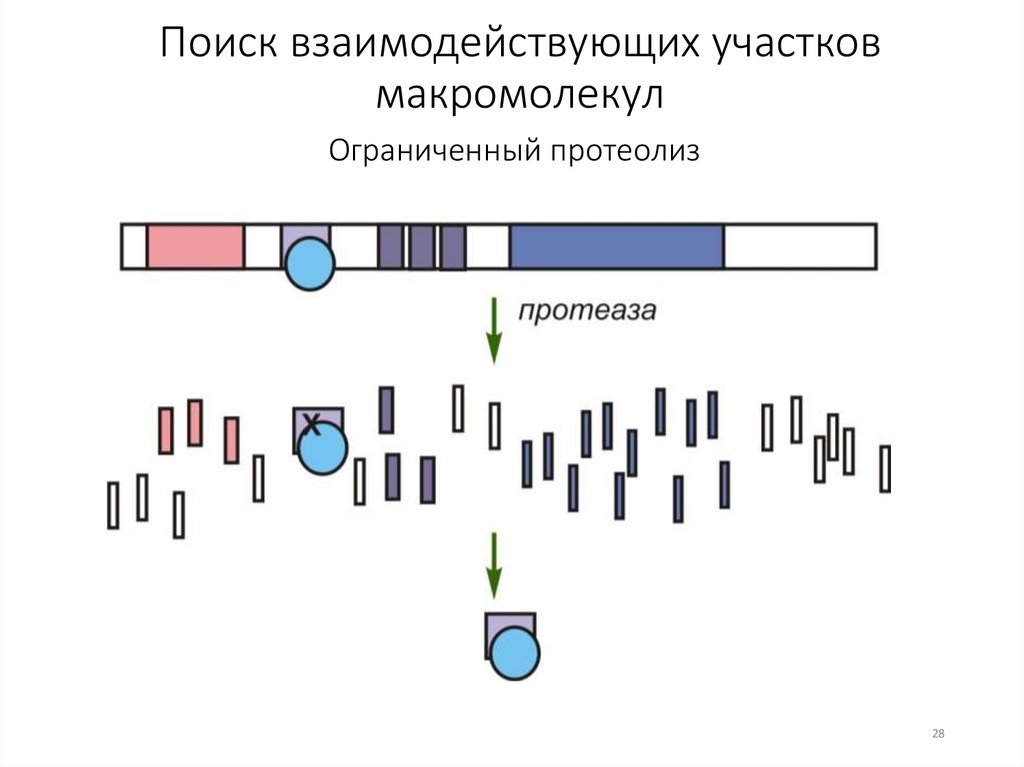
28.
Поиск взаимодействующих участковмакромолекул
Ограниченный протеолиз
28
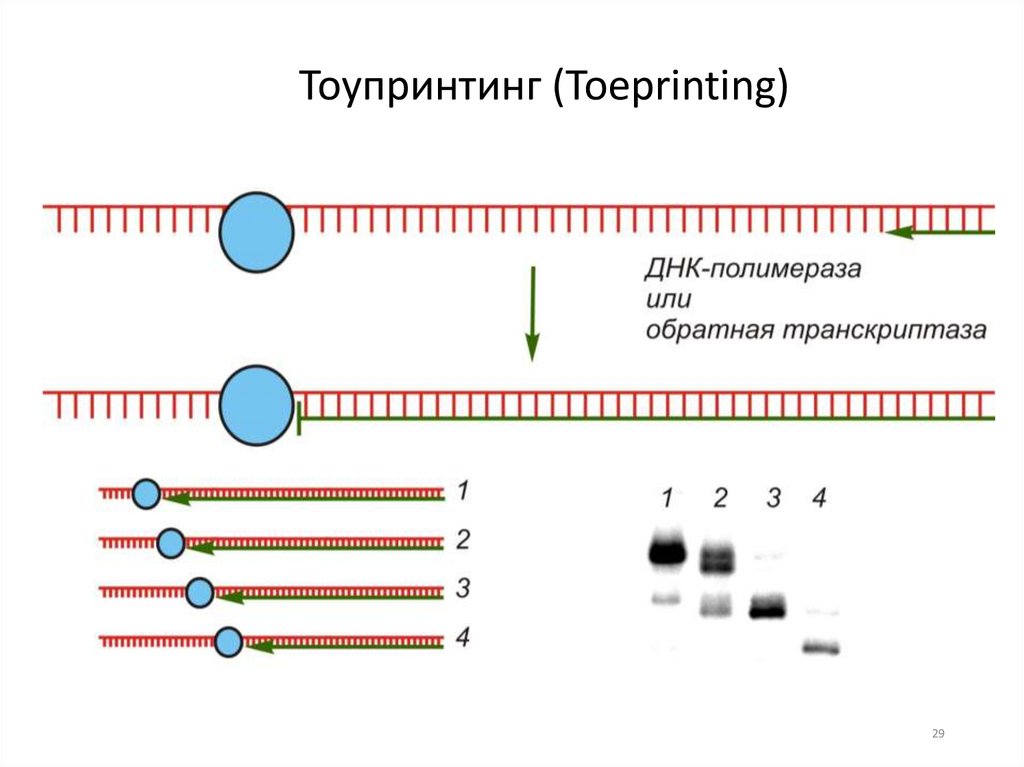
29.
Тоупринтинг (Toeprinting)29
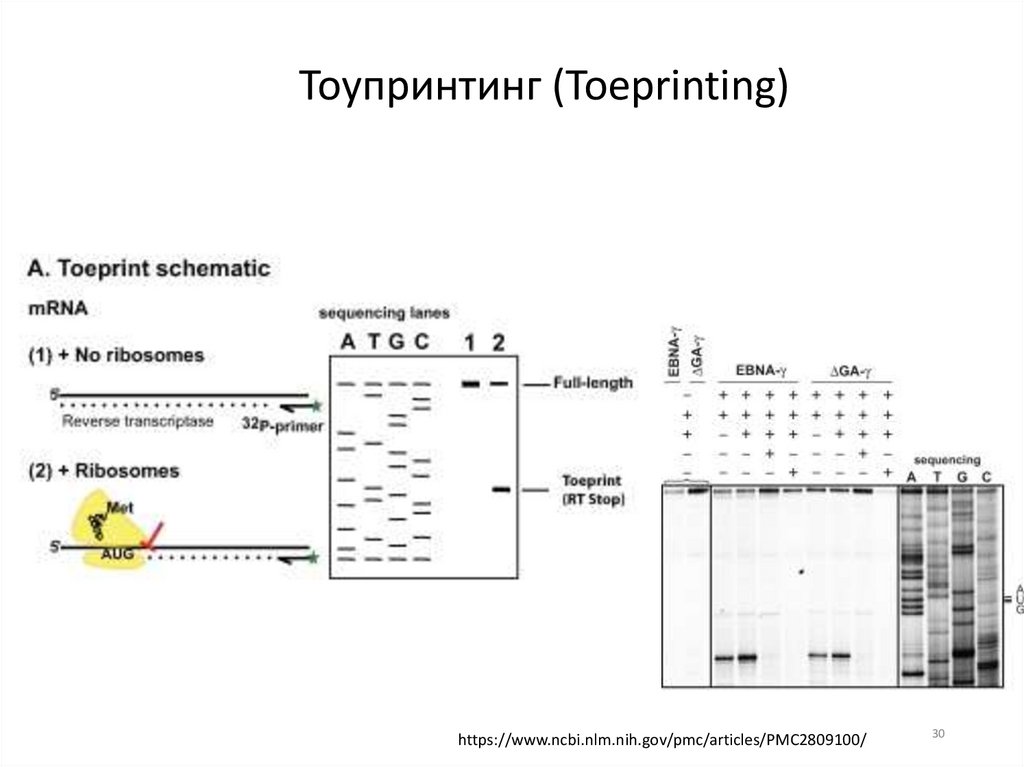
30.
Тоупринтинг (Toeprinting)https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2809100/
30
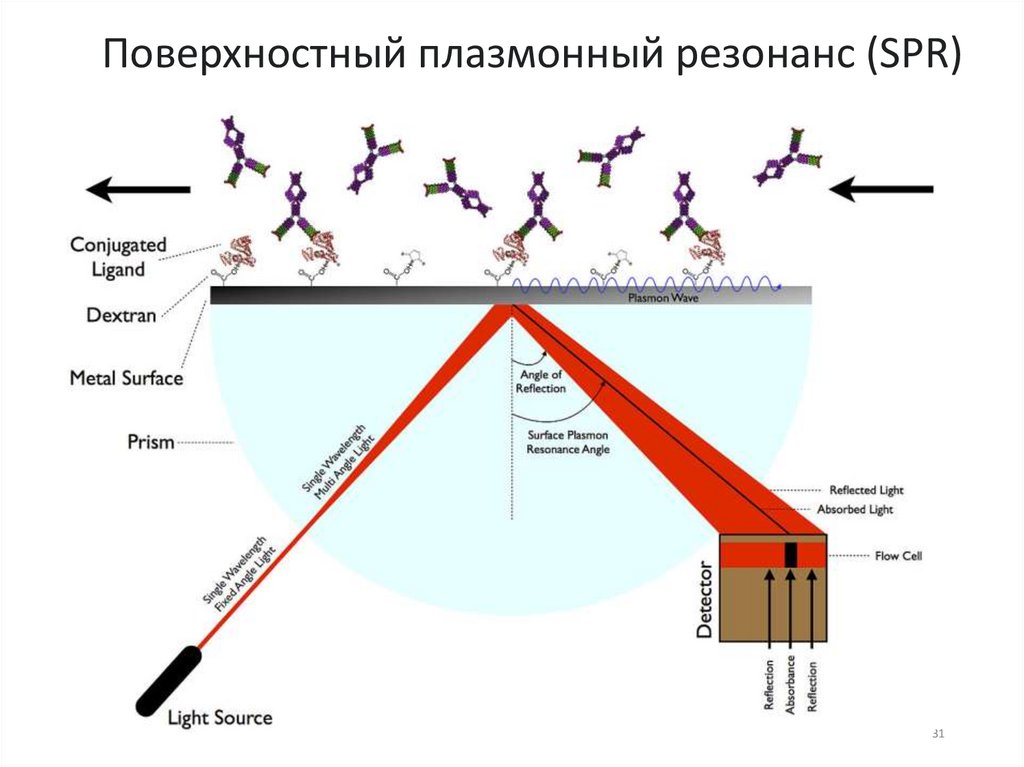
31.
Поверхностный плазмонный резонанс (SPR)31
32.
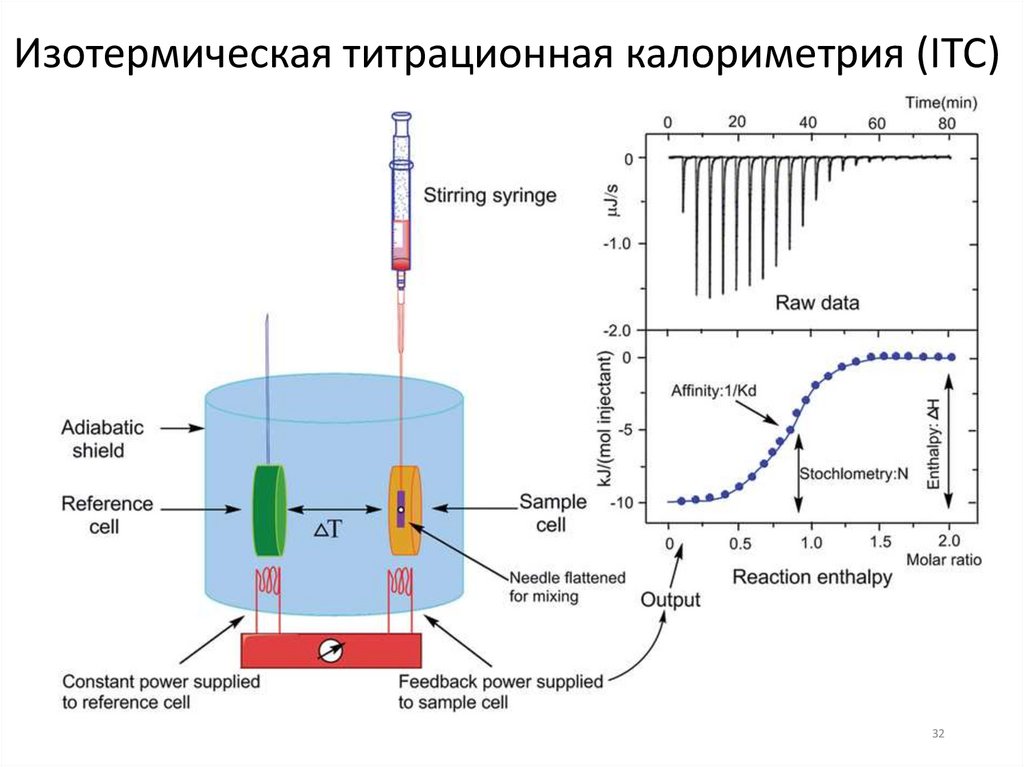
Изотермическая титрационная калориметрия (ITC)32
33.
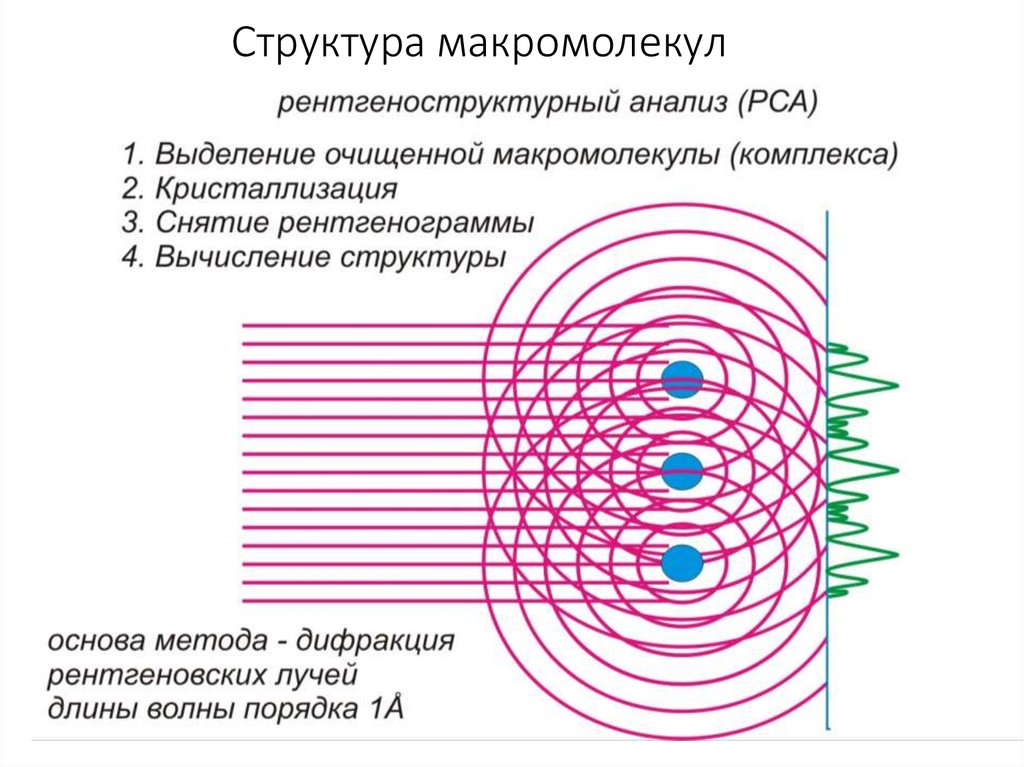
Структура макромолекул33
34.
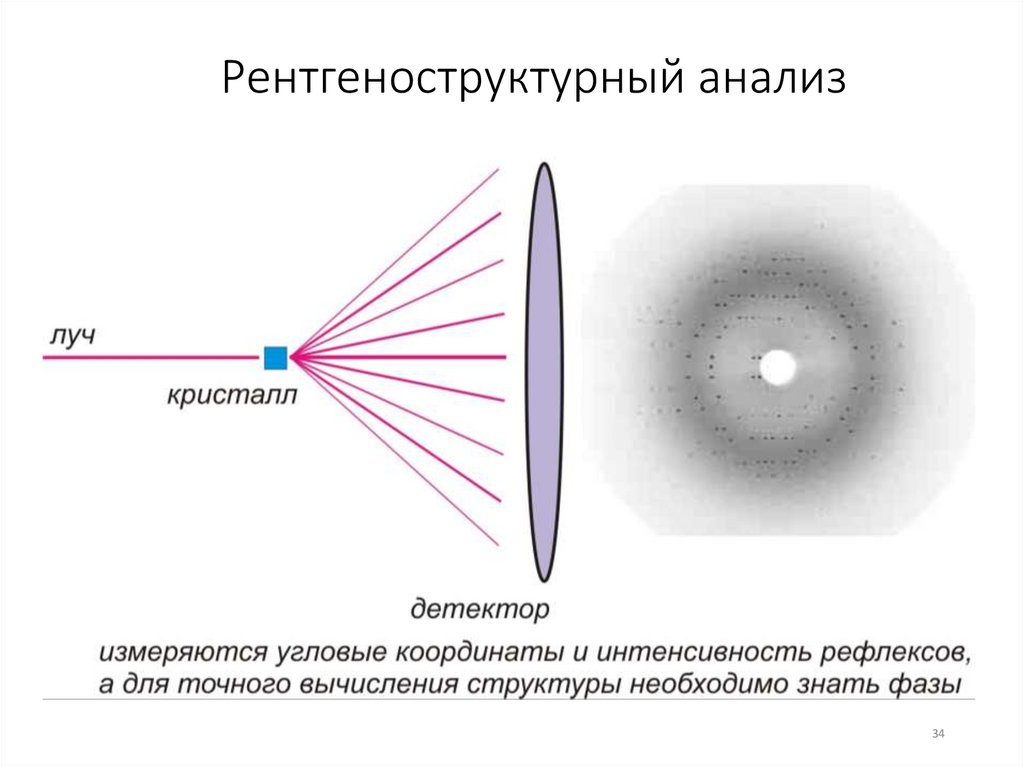
Рентгеноструктурный анализ34
35.
Рентгеноструктурный анализ35
36.
Рентгеноструктурный анализ36
37.
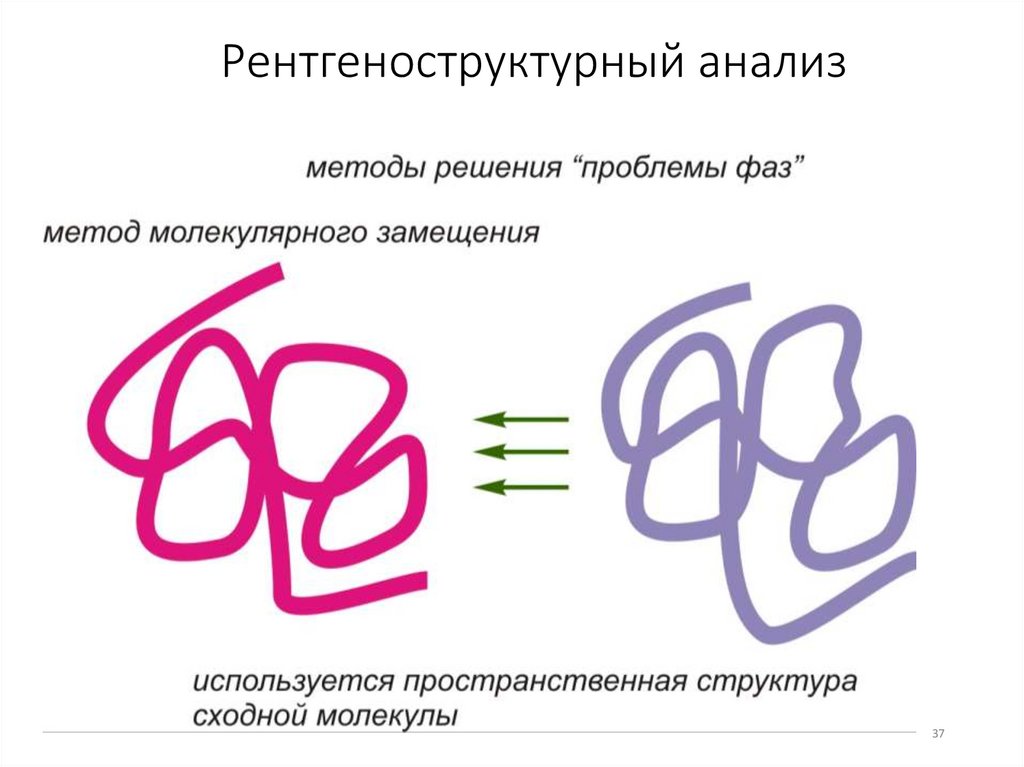
Рентгеноструктурный анализ37
38.
Рентгеноструктурный анализ38
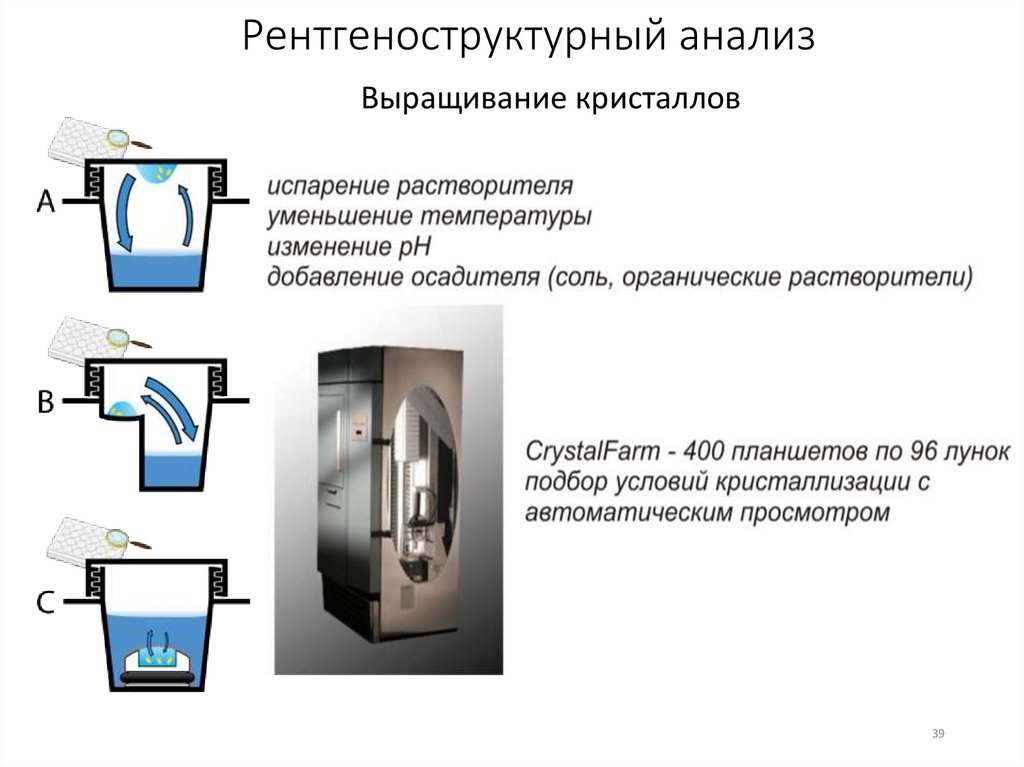
39.
Рентгеноструктурный анализВыращивание кристаллов
39
40.
Рентгеноструктурный анализСинхротрон
40
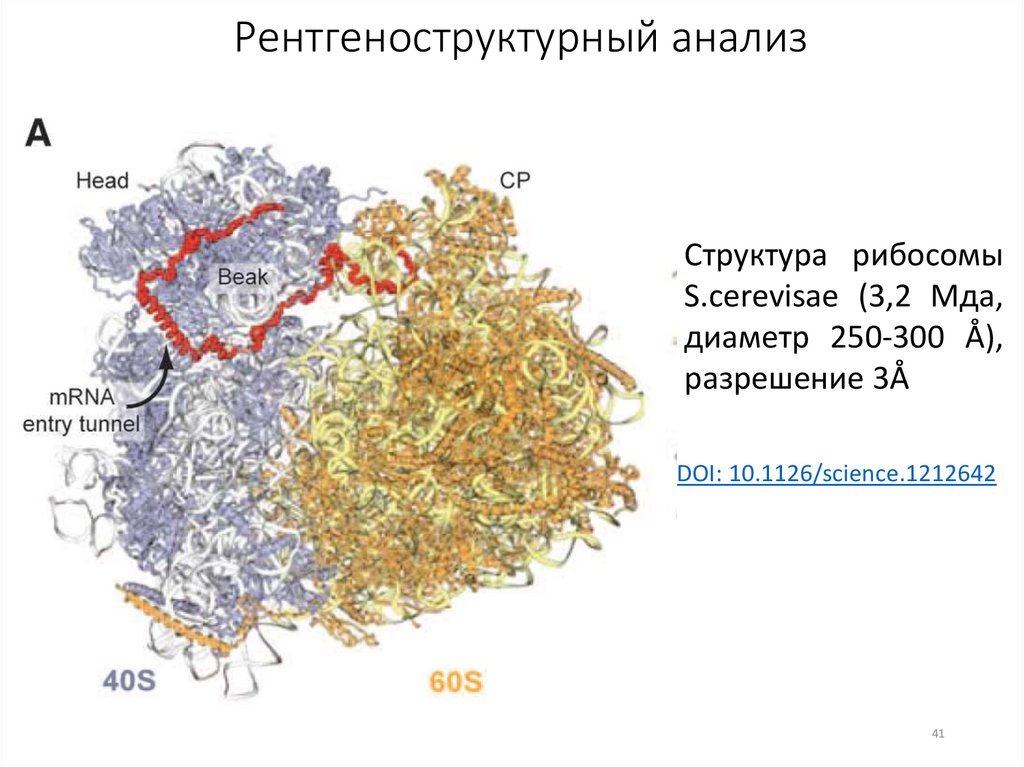
41.
Рентгеноструктурный анализСтруктура рибосомы
S.cerevisae (3,2 Мда,
диаметр 250-300 Å),
разрешение 3Å
DOI: 10.1126/science.1212642
41
42.
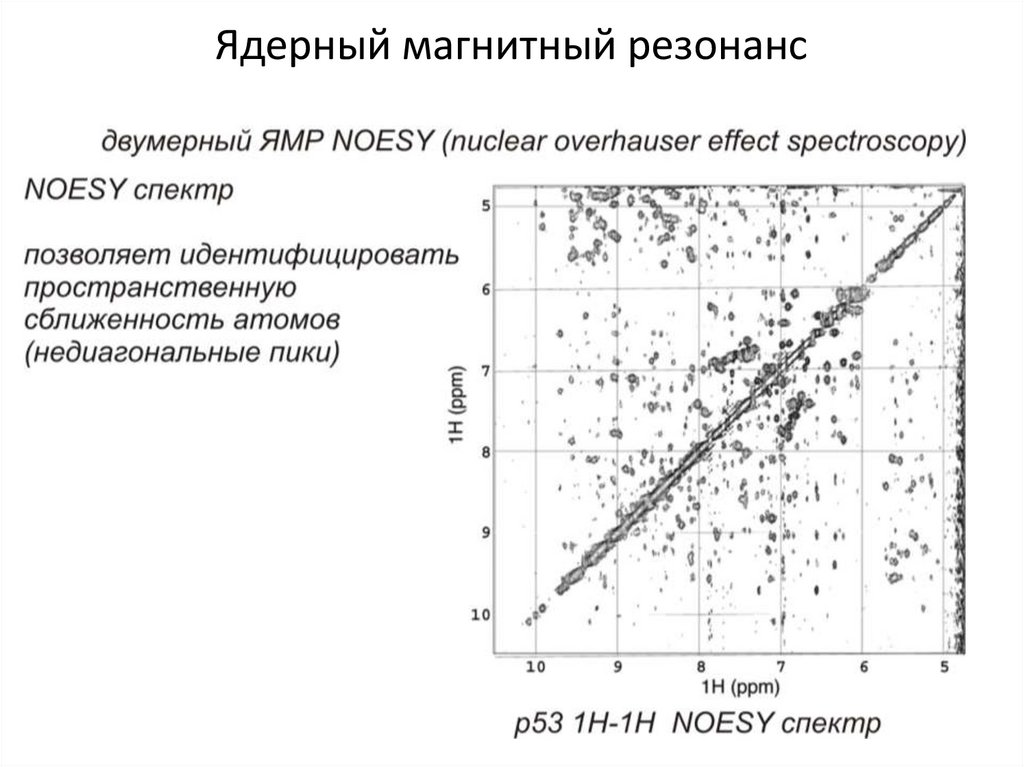
Структура макромолекулЯдерный магнитный резонанс
В магнитном поле у атомов с ненулевым
спином снимается вырождение
электронных уровней ядер
Атомы со спином ½ : 1H, 13C, 15N (белки), 31P (нуклеиновые кислоты), 19F
(аналог Н, малые молекулы), 111Cd/113Cd (аналог Zn в белках)
42
43.
Ядерный магнитный резонансЯМР-спектрометр
Резонансная частота, энергия
поглощенного излучения и
интенсивность
сигнала
пропорциональны
силе
магнитного поля.
ЯМР-спектрометр называют
по частоте (600 МГц, 800 МГц,
1000 МГц), на которой
резонируют в нем атомы 1H в
тетраметилсилане
43
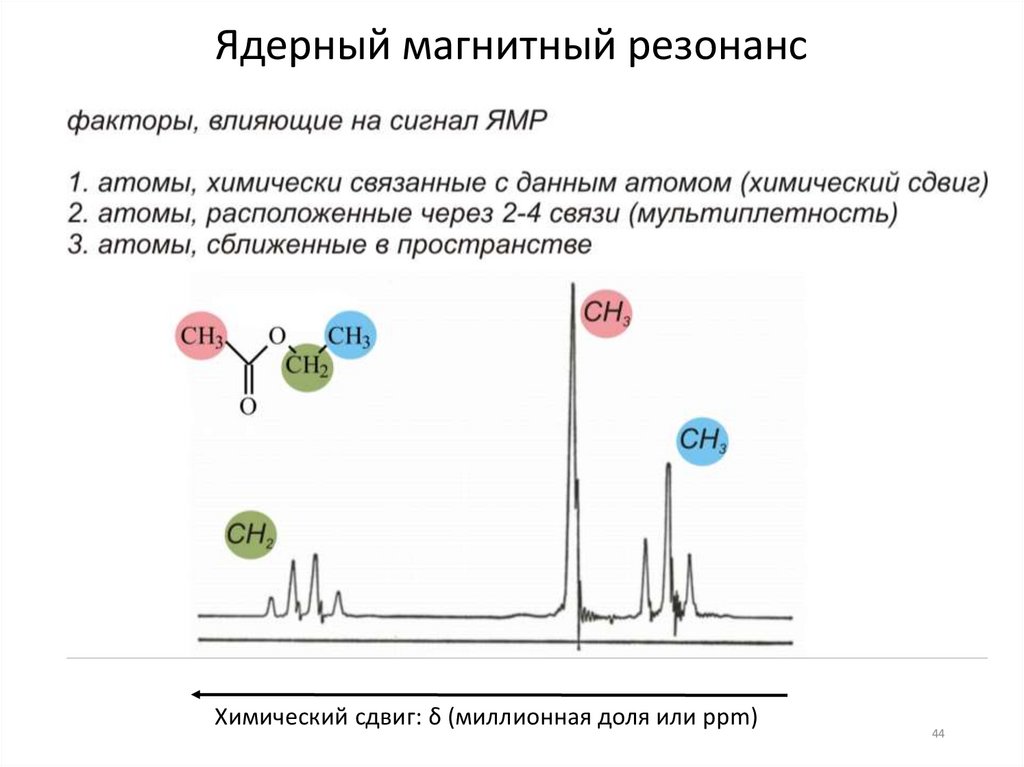
44.
Ядерный магнитный резонансХимический сдвиг: δ (миллионная доля или ppm)
44
45.
Ядерный магнитный резонанс45
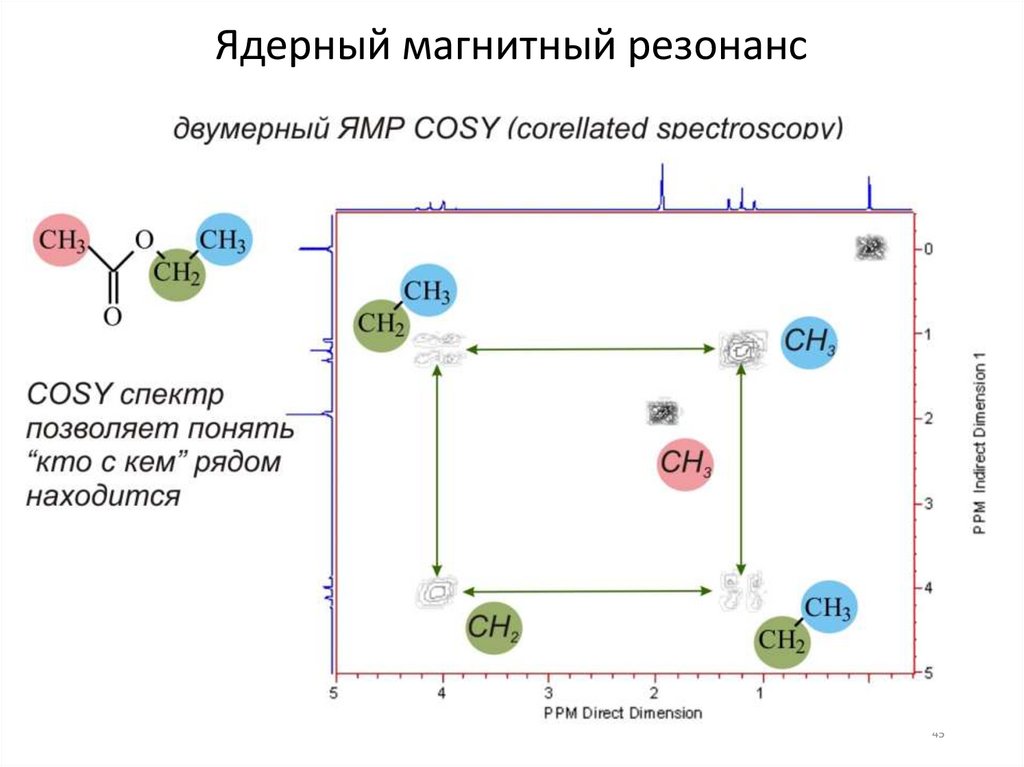
46.
Ядерный магнитный резонанс46
47.
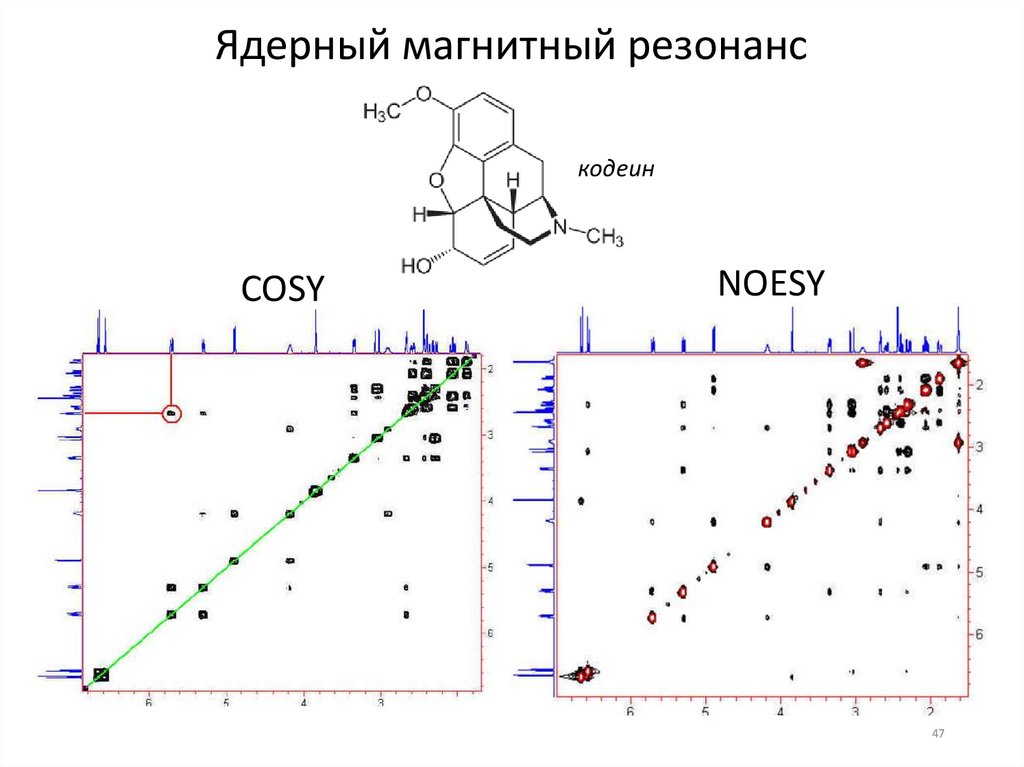
Ядерный магнитный резонанскодеин
COSY
NOESY
47
48.
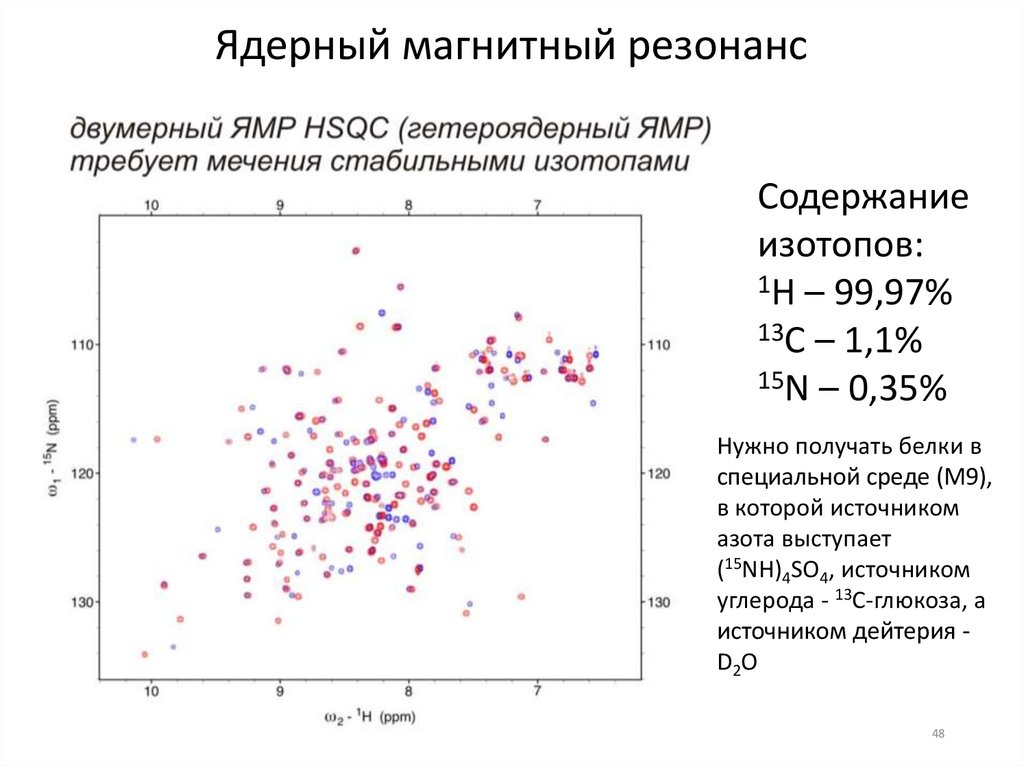
Ядерный магнитный резонансСодержание
изотопов:
1H – 99,97%
13C – 1,1%
15N – 0,35%
Нужно получать белки в
специальной среде (M9),
в которой источником
азота выступает
(15NH)4SO4, источником
углерода - 13C-глюкоза, а
источником дейтерия D2O
48
49.
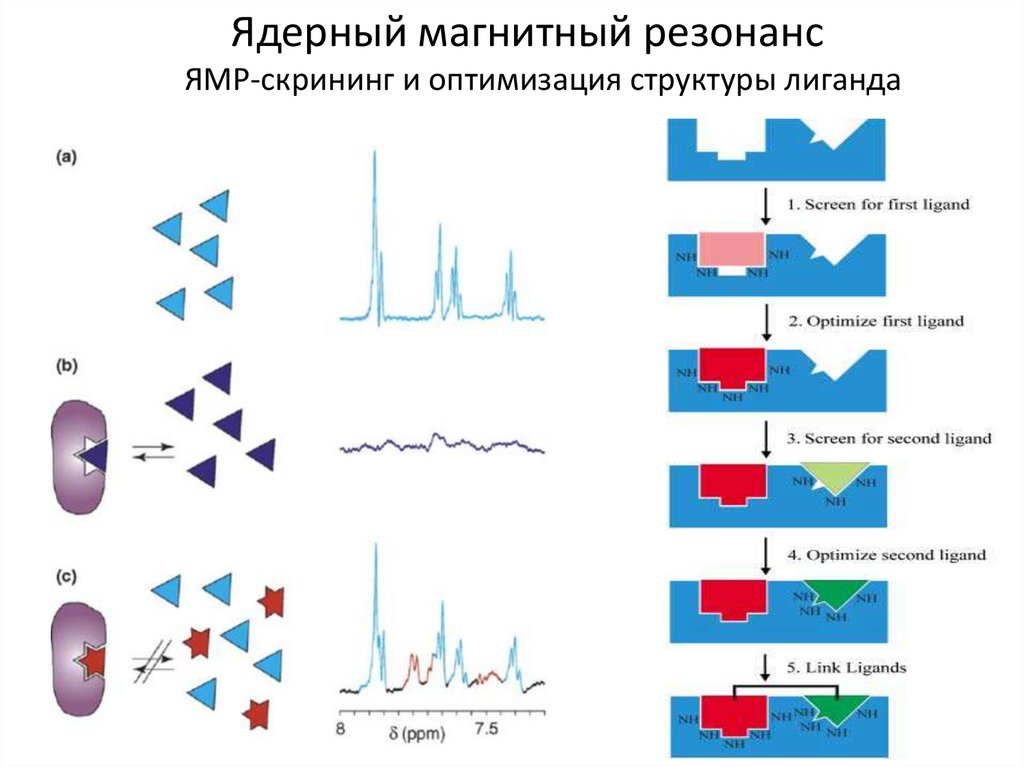
Ядерный магнитный резонансЯМР-скрининг и оптимизация структуры лиганда
49
50.
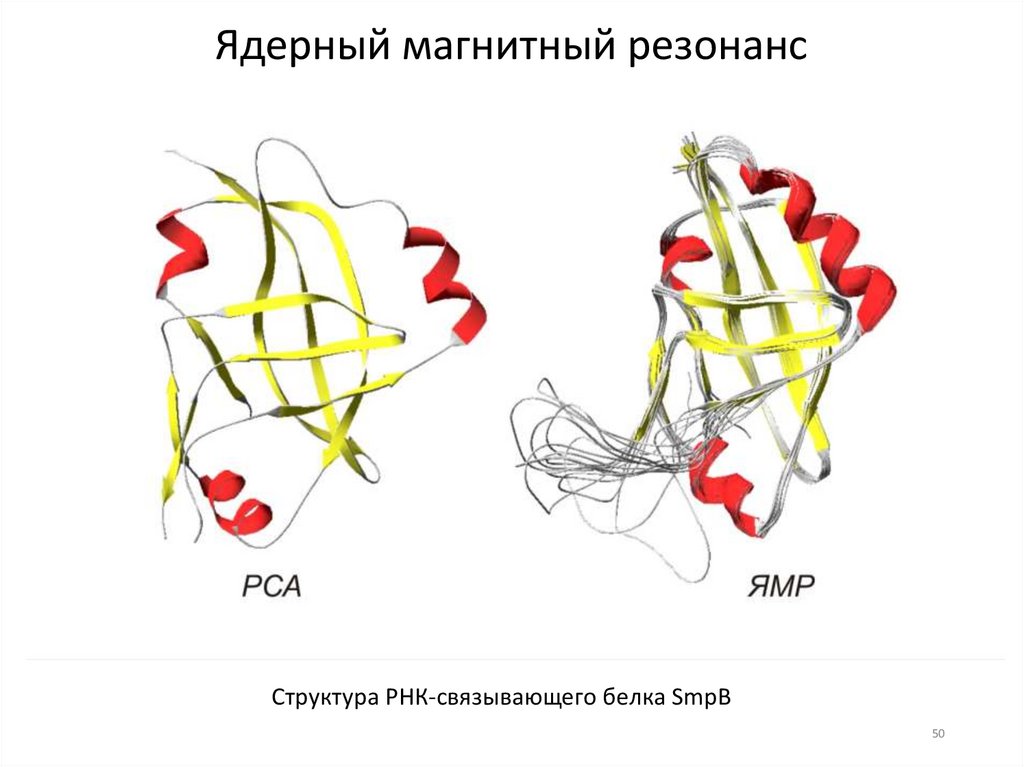
Ядерный магнитный резонансСтруктура РНК-связывающего белка SmpB
50
51.
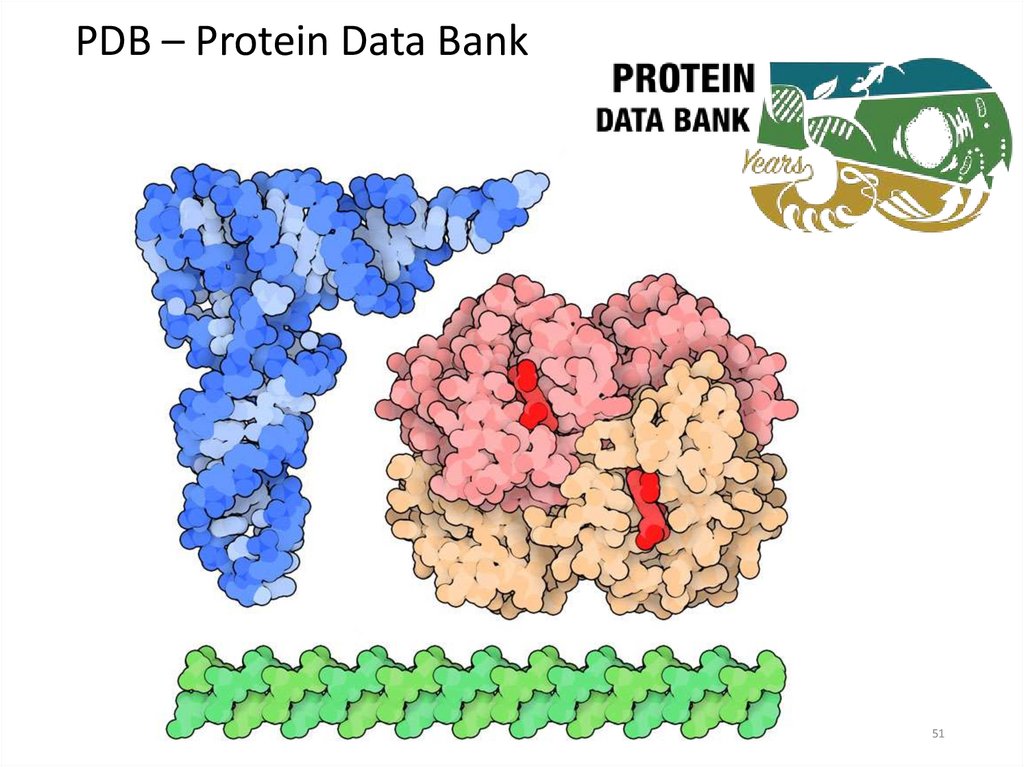
PDB – Protein Data Bank51
52.
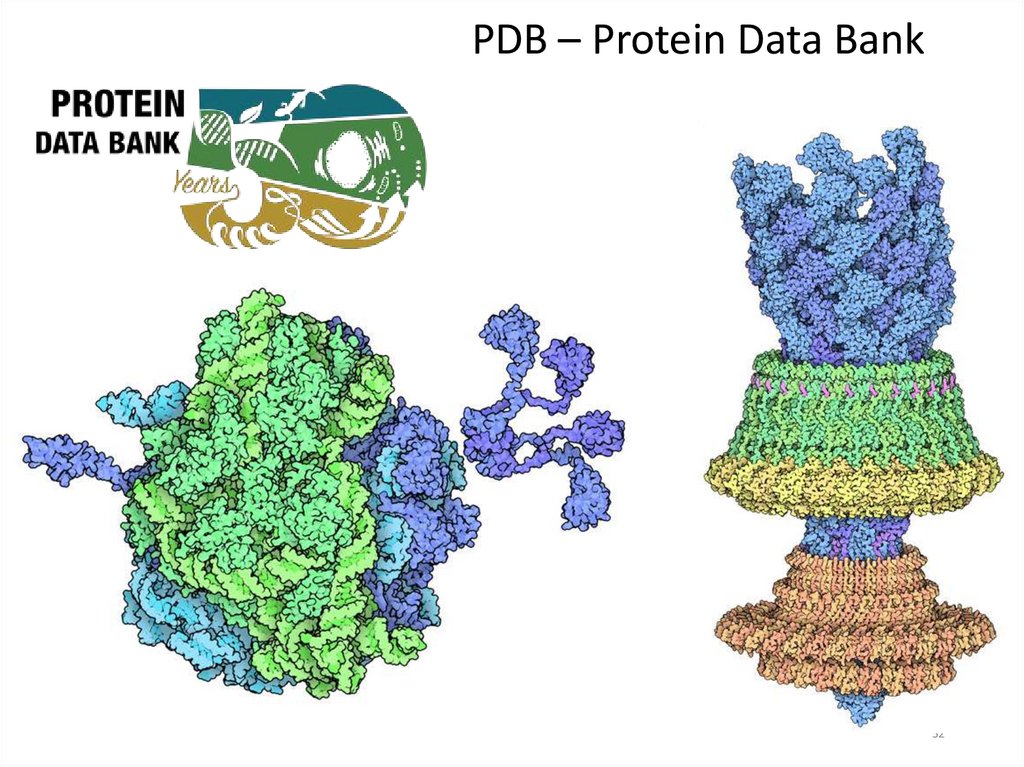
PDB – Protein Data Bank52
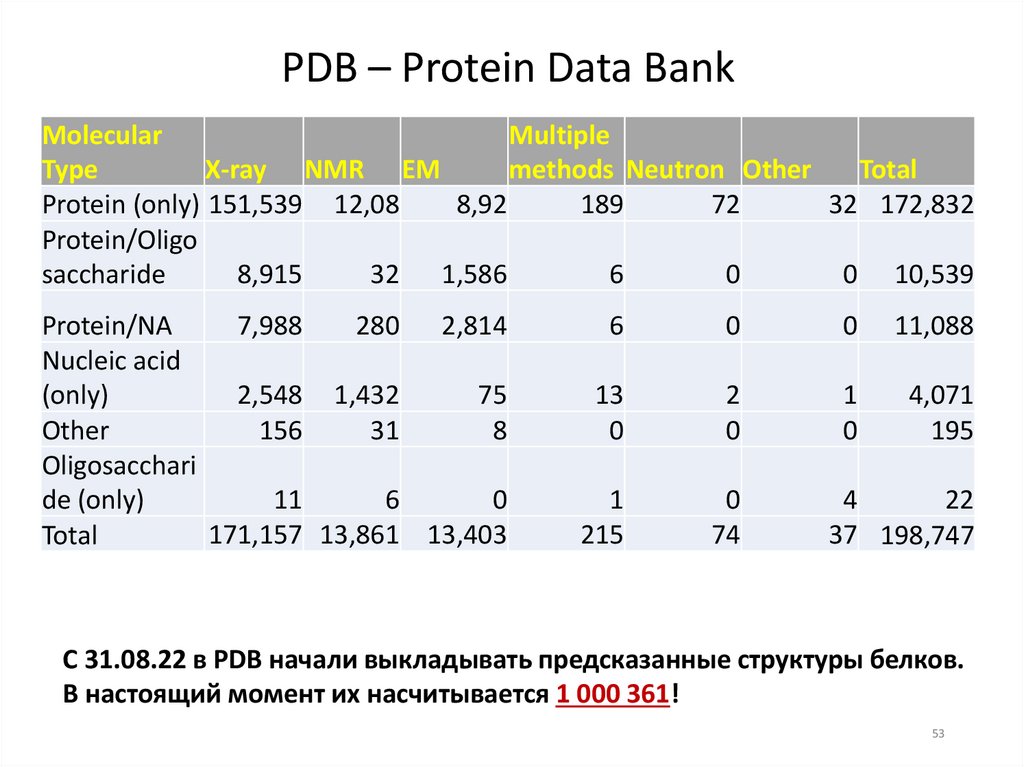
53.
PDB – Protein Data BankMolecular
Multiple
Type
X-ray NMR EM
methods Neutron Other
Total
Protein (only) 151,539 12,08
8,92
189
72
32 172,832
Protein/Oligo
saccharide
8,915
32 1,586
6
0
0 10,539
Protein/NA
7,988
280 2,814
Nucleic acid
(only)
2,548 1,432
75
Other
156
31
8
Oligosacchari
de (only)
11
6
0
171,157 13,861 13,403
Total
6
0
0
11,088
13
0
2
0
1
0
4,071
195
1
215
0
74
4
22
37 198,747
С 31.08.22 в PDB начали выкладывать предсказанные структуры белков.
В настоящий момент их насчитывается 1 000 361!
53
54.
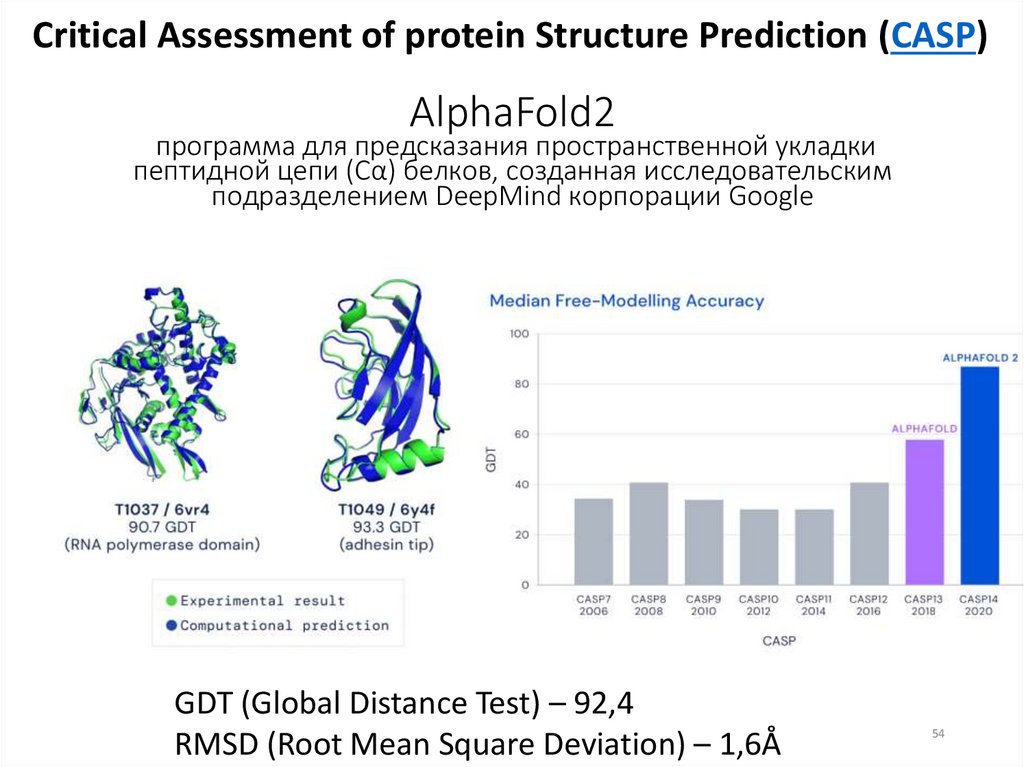
Critical Assessment of protein Structure Prediction (CASP)AlphaFold2
программа для предсказания пространственной укладки
пептидной цепи (Сα) белков, созданная исследовательским
подразделением DeepMind корпорации Google
GDT (Global Distance Test) – 92,4
RMSD (Root Mean Square Deviation) – 1,6Å
54









 biology
biology chemistry
chemistry








